【深度学习】实战深度学习检测疟疾
编译 | VK
来源 | Towards Data Science
在这个项目中,我们将通过美国国立卫生研究院提供的一个数据集,从150名感染了恶性疟原虫寄生虫的患者身上获取27558张不同的细胞图像,并与50名健康患者的细胞图像混合,这些图像可以通过这里的链接下载:
https://www.kaggle.com/iarunava/cell-images-for-detecting-malaria
我们的任务是建立一个机器学习/深度学习算法,能够对检测到的细胞是否被寄生虫感染进行分类。
在这个项目中,我们将使用一种深度学习算法,即卷积神经网络(Convolutional Neural Network, CNN)算法,它通过训练来分类某个细胞的图像是否被感染。由于这是一个超过330mb数据的大项目,我建议将其应用到jupiter Notebook中。
首先,我们需要导入必要的起始库:
import pandas as pd
import numpy as np
import os
import cv2
import random
你之前可能使用panda.read_csv()导入csv格式的数据集,但是,由于所有数据都是png格式的,因此可能需要使用os和cv2库以不同的方式导入它们。
OS是一个强大的Python库,允许你与操作系统进行交互,无论操作系统是Windows、Mac OS还是Linux。
另一方面,cv2是一个专门设计用于解决各种计算机视觉问题,如读取和加载图像。
首先,我们需要设置一个变量来设置我们的路径,因为我们以后会继续使用它:
root = '../Malaria/cell_images/'
in = '/Parasitized/'
un = '/Uninfected/'
因此,从上面的路径是名为“疟疾(Malaria)”的文件夹是我的细胞图像文件夹文件存储的地方,这也是寄生和未感染细胞的图像存储的地方。这样,我们就能更容易地操纵这些路径。
现在让我们操作系统列表目录(“path”)函数列出被寄生和未受感染文件夹中的所有图像,如下所示:
Parasitized = os.listdir(root+in)
Uninfected = os.listdir(root+un)
使用OpenCV和Matplotlib显示图像
例如,与csv文件不同,我们只能使用pandas的函数head列出多个数据,df.head(10) 显示10行数据,对于图像,我们需要使用for循环和matplotlib库来显示数据。
因此,首先让我们导入matplotlib并为每个寄生和未感染的细胞图像绘制图像,如下所示:
import matplotlib.pyplot as plt
然后绘制图像:
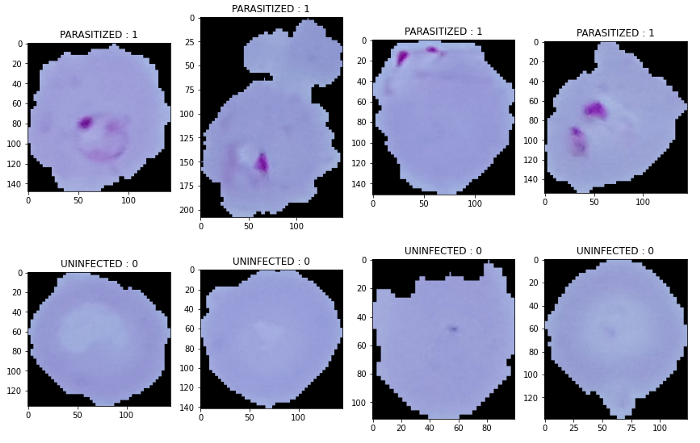
plt.figure(figsize = (12,24))
for i in range(4):
plt.subplot(1, 4, i+1)
img = cv2.imread(root+in+ Parasitized[i])
plt.imshow(img)
plt.title('PARASITIZED : 1')
plt.tight_layout()
plt.show()
plt.figure(figsize = (12,24))
for i in range(4):
plt.subplot(2, 4, i+1)
img = cv2.imread(root+un+ Uninfected[i+1])
plt.imshow(img)
plt.title('UNINFECTED : 0')
plt.tight_layout()
plt.show()
因此,为了绘制图像,我们需要matplotlib中的figure函数,然后我们需要根据figsize函数确定每个图形的大小。
如上所示,其中第一个数字是宽度,第二个数字是高度。然后在for循环中,我们将使用i作为变量来迭代range()中指示的次数。
在本例中,我们将只显示4个图像。随后,我们将使用library中的subplot函数来指示某个迭代的行数、列数和绘图数,我们使用i+1来实现。
由于我们已经创建了子图,但是子图仍然是空的,因此,我们需要使用OpenCV库,通过使用cv2.imread()函数导入图像,并包括其中的路径和变量i,这样它将继续循环到下一张图片,直到提供的最大范围为止。
最后,我们将cv2库导入的图像用plt.imshow()函数绘出,我们将获得以下输出:
将图像和标签分配到变量中
接下来,我们要将所有图像(无论是寄生细胞图像还是未感染细胞图像)及其标签插入到一个变量中,其中1表示寄生细胞,0表示未感染细胞。因此,首先,我们需要为图像和标签创建一个空变量。但在此之前,我们需要使用Keras的img_to_array()函数。
from tensorflow.keras.preprocessing.image import img_to_array
假设存储图像的变量称为data,存储标签的变量称为labels,那么我们可以执行以下代码:
data = []
labels = []
for img in Parasitized:
try:
img_read = plt.imread(root+in+ img)
img_resize = cv2.resize(img_read, (100, 100))
img_array = img_to_array(img_resize)
data.append(img_array)
labels.append(1)
except:
None
for img in Uninfected:
try:
img_read = plt.imread(root+un+ img)
img_resize = cv2.resize(img_read, (100, 100))
img_array = img_to_array(img_resize)
data.append(img_array)
labels.append(0)
except:
None
在为空数组赋值变量数据和标签之后,我们需要使用for循环插入每一张图像。
这里有一些不同之处,for循环中还包含了try和except。try用于在for循环中正常运行代码,另一方面,except用于代码遇到错误或崩溃时,以便代码可以继续循环。
与上面的图像显示代码类似,我们可以使用plt.imread(“path”)函数,但这一次,我们不需要显示任何子图。
你可能想知道我们为什么用plt.imread()而不是cv2.imread()。它们的功能是一样的,事实上,还有很多其他的图像读取库,例如,Pillow库的image.open()或Scikit-Image库的io.imread()。
然而,OpenCV以BGR或蓝、绿、红的顺序读取图像,这就是为什么上面显示的图像是蓝色的,尽管实际的图片是粉色的。因此,我们需要使用以下代码将其转换回RGB顺序:
cv2.cvtColor(img, cv2.COLOR_BGR2RGB)
另一方面,Matplotlib、Scikit Image和Pillow Image reading函数会自动按RGB顺序读取图像,因此,对于我们的单元格图像实际颜色,我们不需要再进行转换:
plt.imshow(data[0])
plt.show()

之后,我们可以通过使用OpenCV的cv2.resize()函数来调整图像的大小,以将加载的图像设置为一个特定的高度和宽度,如上面所示的100 width和100 height。
接下来,因为我们的图像是抽象格式的,我们以后将无法对其进行训练、测试或将其插入变量中,因此,我们需要使用keras的img_to_array()函数将其转换为数组格式。这样,我们就可以使用.append()函数将每个图像插入变量的方括号中,该函数用于在不改变原始状态的情况下将对象插入数组的最后一个列表。
所以在我们的循环中,我们不断地在每个循环中添加一个新的图像。
预处理数据
不像机器学习项目,我们可以立即分割我们的数据,在深入学习,特别是神经网络,以减少方差,并减少过拟合。在Python中,有许多方法可以随机化数据,比如使用Sklearn,如下所示:
from sklearn.utils import shuffle
或使用如下所示的随机方法:
from random import shuffle
但在这个项目中,我们将使用Numpy的随机函数。因此,我们需要将数组转换成Numpy的数组函数,然后对图像数据进行随机化,如下所示:
image_data = np.array(data)
labels = np.array(labels)
idx = np.arange(image_data.shape[0])
np.random.shuffle(idx)
image_data = image_data[idx]
labels = labels[idx]
首先,我们将数据和标签变量转换为Numpy格式。然后,我们可以使用np.arange()然后使用np.random.shuffle()功能。最后,我们将被随机数据重新分配到它们的原始变量中,以确保被随机的数据被保存。
用于训练、测试和验证的拆分数据
在数据被随机之后,我们应该通过导入必要的库将它们拆分为训练、测试和验证标签和数据,如下所示:
from tensorflow.keras.utils import to_categorical
from sklearn.model_selection import train_test_split
然后我们设置一个函数,将数据转换为32位数据以保存:
def prep_dataset(X,y):
X_prep = X.astype('float32')/255
y_prep = to_categorical(np.array(y))
return (X_prep, y_prep)
然后通过使用Sklearn库,我们可以将数据分为训练、测试和验证:
X_tr, X_ts, Y_tr, Y_ts = train_test_split(image_data,labels, test_size=0.15, shuffle=True,stratify=labels,random_state=42)
X_ts, X_val, Y_ts, Y_val = train_test_split(X_ts,Y_ts, test_size=0.5, stratify=Y_ts,random_state=42)
X_tr, Y_tr = prep_dataset(X_tr,Y_tr)
X_val, Y_val = prep_dataset(X_val,Y_val)
X_ts, _ = prep_dataset(X_ts,Y_ts)
由于存在验证数据,因此需要执行两次拆分。在spliting函数中,我们需要在前两个参数中分配数据和标签,然后告诉它们被划分成的百分比,随机状态是为了确保它们产生的数据总是以一致的顺序排列。
卷积神经网络模型的建立
在建立CNN模型之前,让我们从理论上对它有一点更深入的了解。CNN主要应用于图像分类,自1998年发明以来,由于其高性能和高精度,一直受到人们的欢迎。那么它是如何工作的呢?
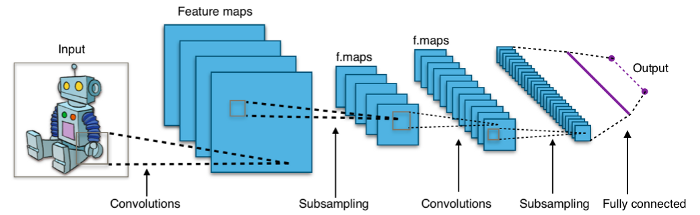
正如我们从上面的插图中看到的,CNN对图像进行分类,取图像的一部分,并将其通过几层卷积处理来分类图像。从这几个层中,可以将它们分为三种类型的层,分别是卷积层、池化层和全连接层。
卷积层
第一层,卷积层是CNN最重要的方面之一,它的名字来源于此。其目的是利用核函数从输入图像中提取特征。该核将使用点积连续扫描输入图像,创建一个新的分析层,称为特征图/激活图。其机理如下:
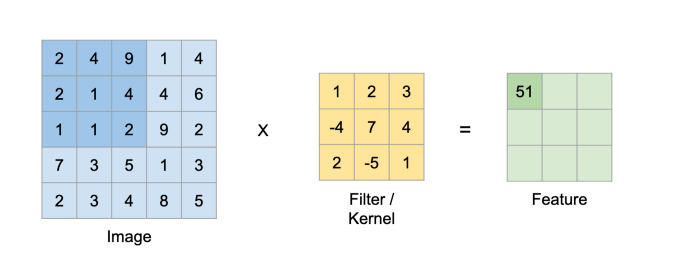
2*1 + 4*2 + 9*3 + 2*(-4) + 1*7 + 4*4 + 1*2 + 1*(-5) + 2*1 = 51
然后继续,直到所有特征图的单元格都被填满。
池化层
在我们创建了特征图之后,我们将应用一个池化层来减小其大小以减少过拟合。此步骤中有几个操作,但是,最流行的技术是最大池,其中特征图的扫描区域将仅取最高值,如下图所示:
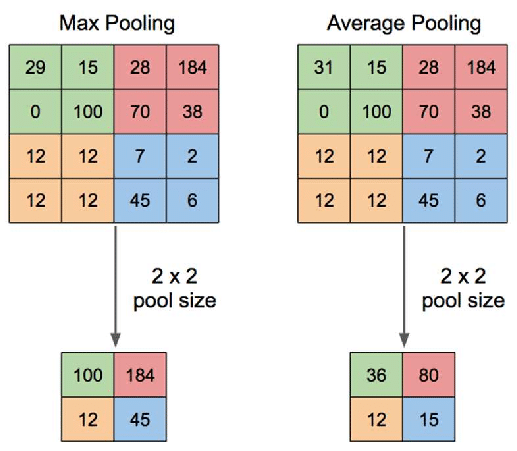
卷积和池的步骤可以重复,直到理想的大小是最后敲定,然后我们可以继续分类部分,它被称为全连接层。
全连通层
在这一阶段,变换后的特征图将被展平为一个列向量,该列向量将在每个迭代过程中经过前馈神经网络和反向传播过程,持续数个阶段。最后,CNN模型将区分主要特征和非主要特征,利用Softmax分类对图像进行分类。
在Python中,我们需要从Keras库导入CNN函数:
from tensorflow.keras import models, layers
from tensorflow.keras.callbacks import EarlyStopping
然后,我们将构建我们自己的CNN模型,包括4个卷积层和池化层:
model = models.Sequential()
#Input + Conv 1 + ReLU + Max Pooling
model.add(layers.Conv2D(32,(5,5),activation='relu',padding='same',input_shape=X_tr.shape[1:]))
model.add(layers.MaxPool2D(strides=4))
model.add(layers.BatchNormalization())
# Conv 2 + ReLU + Max Pooling
model.add(layers.Conv2D(64,(5,5),padding='same',activation='relu'))
model.add(layers.MaxPool2D(strides=2))
model.add(layers.BatchNormalization())
# Conv 3 + ReLU + Max Pooling
model.add(layers.Conv2D(128,(3,3),padding='same',activation='relu'))
model.add(layers.MaxPool2D(strides=2))
model.add(layers.BatchNormalization())
# Conv 4 + ReLU + Max Pooling
model.add(layers.Conv2D(256,(3,3),dilation_rate=(2,2),padding='same',activation='relu'))
model.add(layers.Conv2D(256,(3,3),activation='relu'))
model.add(layers.MaxPool2D(strides=2))
model.add(layers.BatchNormalization())
# Fully Connected + ReLU
model.add(layers.Flatten())
model.add(layers.Dense(300, activation='relu'))
model.add(layers.Dense(100, activation='relu'))
#Output
model.add(layers.Dense(2, activation='softmax'))
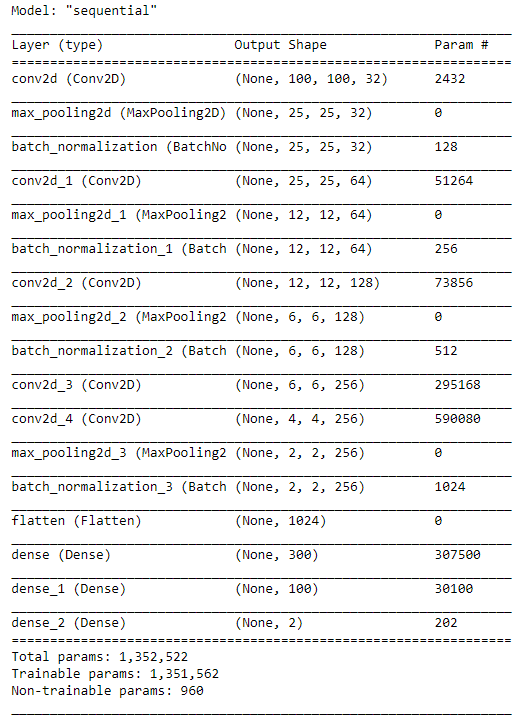
model.summary()
Keras中的卷积层
好的,在这段代码中,你可能会感到困惑的第一件事是model .sequential()。
利用Keras构建深度学习模型有两种选择,一种是序列模型,另一种是函数模型。他们两人之间的差异是序列模型只允许一层一层地建立一个模型,另一方面,函数模型允许层连接到前一层,多层,甚至任何层,你想建立一个更复杂的模型。由于我们正在构建一个简单的CNN模型,所以我们将使用序列模型。
我们之前了解到,CNN由卷积层组成,卷积层后来使用池化层进行简化,因此,我们需要使用Keras函数model.add()添加层,然后添加我们喜欢的层。
由于我们的图像是2D形式,我们只需要2D卷积层,同样使用卷积层的Keras函数:layers.Conv2D(). 正如你在每一层所看到的,第一层有一个数字32,第二层有一个数字64,乘以2并以此类推。这叫做滤波器。它所做的是试图捕捉图像的模式。
因此,随着滤波器尺寸的增大,我们可以在较小的图像上捕获更多的模式,尽管没有太多理论的证明,然而,这被认为是最优方法。
接下来,(5,5)和(3,3)矩阵是我们上面讨论的用于创建特征图的核。然后这里有两个有趣的部分:ReLu的激活和填充。那些是什么?我们先来讨论ReLu。它是由Rectified Linear Unit缩短而来的,其想法是采用更简单的模型,提高训练过程的效率。
下一个问题是填充。如我们所知,卷积层不断减小图像的大小,因此,如果我们在新的特征图的所有四个边上添加填充,我们将保持相同的大小。所以我们在处理后的图像周围添加新的单元格,值为0,以保持图像的大小。填充有两个选项:相同或零。接下来就是input_shape,它只是我们的输入图像,只在第一层中用于输入我们的训练数据。
Keras中的池化层
让我们转到池化层,我们可以使用Keras的layers.MaxPool2D()。这里有一种东西叫做跨步(stride)。它是核将在输入上每次移动的步数。因此,如果我们用5x5的核,并且跨步为4,核将计算图像最左边的部分,然后跳过右边的4个单元格来执行下一次计算。
你可能会注意到,每个卷积层和池化层都以批标准化结束。为什么?因为在每一层中,都有不同的分布,训练过程会变慢,因为它需要适应每一层。但是,如果强制所有层具有相似的分布,则可以跳过此步骤并提高训练速度。这就是为什么我们在每一层中应用批标准化,通过如下所示的4个步骤对每一层中的输入进行标准化:

Keras全连接层
现在我们已经到了CNN的最后阶段,也就是全连接的阶段。但在进入这个阶段之前,由于我们将在全连接阶段使用“Dense”层,所以我们需要使用Keras层将处理过的数据平铺成一维。Flatten()函数用于将垂直和水平的数据合并到单个列中。

我们将数据展平后,现在需要使用layers.Dense()函数将它们全连接起来,然后指定要用作输出的神经元数量,考虑到当前的神经元是1024个。所以我们先使用300个神经元,然后是100个神经元,并使用ReLu对其进行优化。
最后,我们到达输出阶段,这也是通过使用Dense函数来完成的,然而,由于我们的分类只有2个概率,即感染疟疾或未感染疟疾,因此,我们将输出单位设置为2。此外,在分类任务中,我们在输出层使用Softmax激活而不是ReLu,因为ReLu将所有负类设置为零。
综上所述,我们构建的模型如下:
然后,我们将把我们的训练和验证数据整合到模型中:
model.compile(optimizer='adam',
loss='categorical_crossentropy',
metrics=['accuracy'])
es = EarlyStopping(monitor='val_accuracy',mode='max',patience=3,verbose=1)
history= model.fit(X_tr,Y_tr,
epochs=20,
batch_size=50,
validation_data=(X_val,Y_val),
callbacks=[es])
由于数据量很大,这个过程需要一些时间。如下图所示,每个epoch平均运行9分钟。

评估
我们可以通过绘制评估图来评估模型性能,但在此之前,我们需要导入seaborn库:
import seaborn as sns
然后将准确度和损失绘制如下:
fig, ax=plt.subplots(2,1,figsize=(12,10))
fig.suptitle('Train evaluation')
sns.lineplot(ax= ax[0],x=np.arange(0,len(history.history['accuracy'])),y=history.history['accuracy'])
sns.lineplot(ax= ax[0],x=np.arange(0,len(history.history['accuracy'])),y=history.history['val_accuracy'])
ax[0].legend(['Train','Validation'])
ax[0].set_title('Accuracy')
sns.lineplot(ax= ax[1],x=np.arange(0,len(history.history['loss'])),y=history.history['loss'])
sns.lineplot(ax= ax[1],x=np.arange(0,len(history.history['loss'])),y=history.history['val_loss'])
ax[1].legend(['Train','Validation'])
ax[1].set_title('Loss')
plt.show()
这将给我们一个输出:
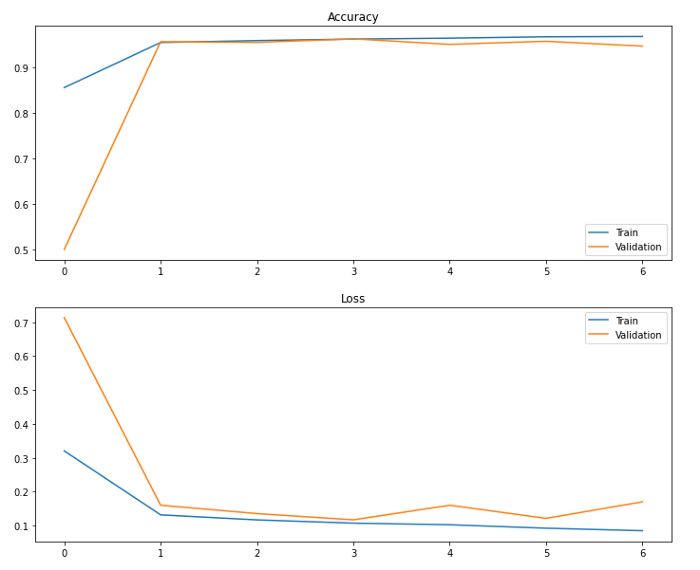
它的平均性能似乎超过90%,这是非常好的,正如CNN模型所预期的。不过,为了让我们更具体地了解数据性能,让我们使用Sklearn构建一个混淆矩阵,并预测输出:
from sklearn.metrics import confusion_matrix, accuracy_score
Y_pred = model.predict(X_ts)
Y_pred = np.argmax(Y_pred, axis=1)
conf_mat = confusion_matrix(Y_ts,Y_pred)
sns.set_style(style='white')
plt.figure(figsize=(12,8))
heatmap = sns.heatmap(conf_mat,vmin=np.min(conf_mat.all()), vmax=np.max(conf_mat), annot=True,fmt='d', annot_kws={"fontsize":20},cmap='Spectral')
heatmap.set_title('Confusion Matrix Heatmap\n¿Is the cell infected?', fontdict={'fontsize':15}, pad=12)
heatmap.set_xlabel('Predicted',fontdict={'fontsize':14})
heatmap.set_ylabel('Actual',fontdict={'fontsize':14})
heatmap.set_xticklabels(['NO','YES'], fontdict={'fontsize':12})
heatmap.set_yticklabels(['NO','YES'], fontdict={'fontsize':12})
plt.show()
print('-Acuracy achieved: {:.2f}%\n-Accuracy by model was: {:.2f}%\n-Accuracy by validation was: {:.2f}%'.
format(accuracy_score(Y_ts,Y_pred)*100,(history.history['accuracy'][-1])*100,(history.history['val_accuracy'][-1])*100))
这将为我们提供以下准确度:
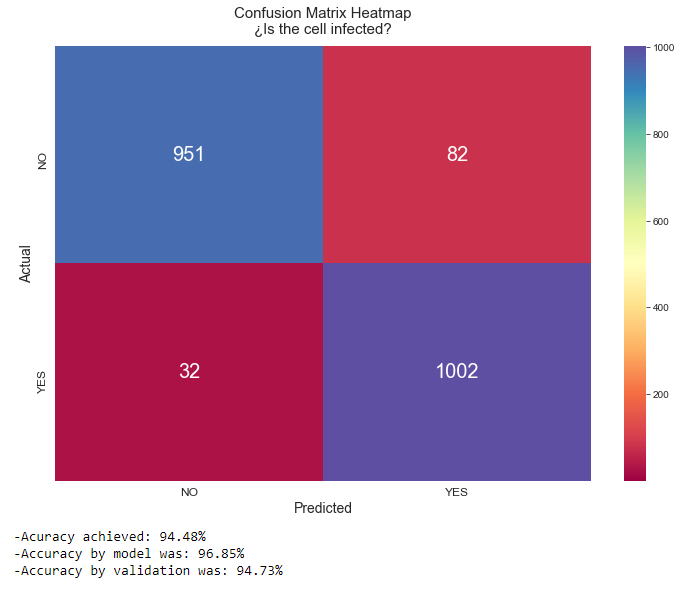
错误样本
让我们看看错误示例的样子:
index=0
index_errors= []
for label, predict in zip(Y_ts,Y_pred):
if label != predict:
index_errors.append(index)
index +=1
plt.figure(figsize=(20,8))
for i,img_index in zip(range(1,17),random.sample(index_errors,k=16)):
plt.subplot(2,8,i)
plt.imshow(np.reshape(255*X_ts[img_index], (100,100,3)))
plt.title('Actual: '+str(Y_ts[img_index])+' Predict: '+str(Y_pred[img_index]))
plt.show()
输出:
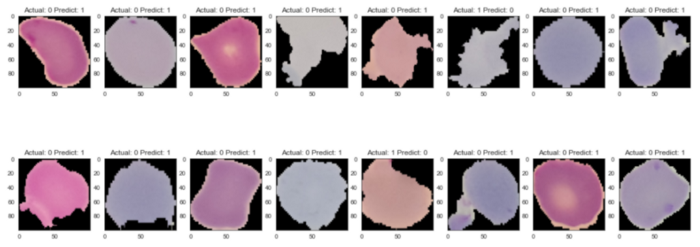
尽管未感染,但错误的预测图像似乎包含了细胞上的几个紫色斑块,因此,模型实际预测它们为感染细胞是可以理解的。
我想说CNN是一个非常强大的图像分类模型,不需要做很多预处理任务,因为处理包含在卷积层和池化层中。
希望通过本文的学习,让你能够利用卷积神经网络进行图像分类。
感谢你的阅读。
看到这里,说明你喜欢这篇文章,请点击「在看」或顺手「转发」「点赞」。
往期精彩回顾
本站qq群851320808,加入微信群请扫码:
