【CV】使用 scikit-image 进行图像处理
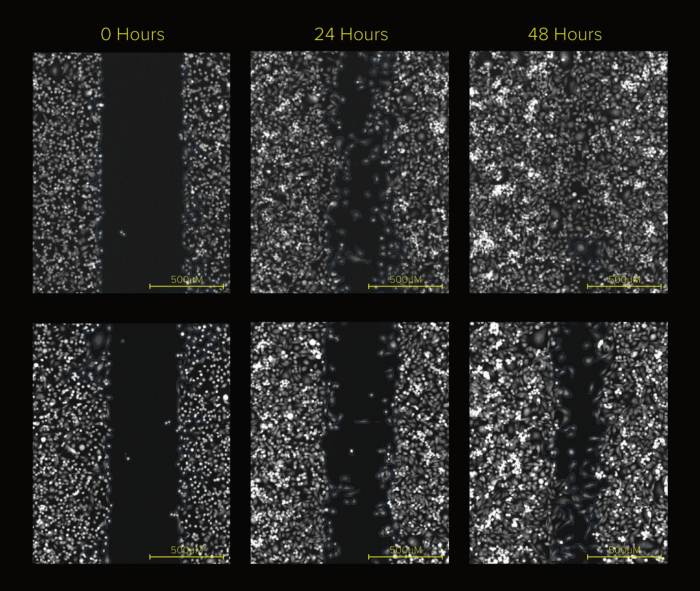
划痕试验时间序列分析。
图像处理在当今非常适用,对于2019冠状病毒疾病,无论是开发治疗方法还是寻找检测病毒的新方法,都给全球的研究人员带来了新的挑战。为了对 2019冠状病毒进行研究,研究人员通常需要对图像进行大量计算,以检测有助于得出新见解和结论的特定元素。
在这篇文章中,我将介绍图像处理的基础知识和一个小教程,详细介绍如何将图像处理用于阈值和分割,这是图像处理中两种非常强大的技术。在文章中,我们将使用 scikit-image,因此可能需要 Python 的基本背景知识和一些高等数学知识才能完全理解该概念。
除非我们有丰富的生物学经验,否则大多数人可能不知道什么是划痕试验,我将简要概述什么是划痕检测,然后直接进入教程。
划痕试验是生物学家用来跟踪细胞运动和相互作用的一种技术,具体来说,生物学家在培养皿中采集细胞样本,并在细胞平面上制造人工伤口,在开发人造伤口后,生物学家会定期拍摄任何细胞运动的图像,以开发出一系列用于分析的图像。
在下面的教程中,我们将处理下面提供的图像,因此在开始之前下载此图像并将其添加到我们的项目目录中。

从显微镜获得的划痕分析图像
首先,我们需要确定我们要试图做什么来处理此图像,为了从该图像中获得更多的信息,我们将使用各种分割技术来分离细胞区域和划痕区域(空白区域)。如我们所见,由于整个图像的像素值基本相同,因此我们不能仅根据颜色对该图像进行分割,但是我们可以使用纹理作为参数来确定培养皿的哪个区域有细胞,哪个区域没有细胞。为此,在开始分割图像之前,我们将对图像应用一个特殊的过滤器。
首先,让我们将所有必要的库导入我们的 Python 脚本:

然后,导入我们上面提供的图像,我们在下面的代码片段中将图像命名为“scratch.jpg”。

在上面的代码中,我使用了scikit image中的io包来使用imread函数,该函数允许我输入图像数据,此函数的工作原理与用于 CSV 文件的 Pandas read_csv 方法完全相同。此外,我们使用了 scikit-image 的形态学包中的熵过滤器,这将允许我们通过搜索某些像素中的无序情况来分析图像的纹理。如果小伙伴们想进一步了解所有这些函数的功能和参数,请访问 scikit-image 文档。我们可以使用磁盘参数,但对于此图像,磁盘值为 10 时可获得最佳结果。

现在,我们将应用Otsu阈值滤波器,该滤波器来自skiliage.filters包,这个简单的单行命令将允许我们应用Otsu阈值滤波器。Otsu阈值是一种特定算法,它迭代图像中的每个像素并将像素分为两类:背景和前景。一旦我们实际绘制了图像,我们就会理解其效果。
阈值过滤器只返回单个数字,该值是 Otsu 算法为优化图像分割计算的精确值,将帮助我们生成最终图像。


最后,在这里我们可以看到图像分割的结果。在这段代码中,我们定义了一个子图,其中将显示原始图像、使用熵过滤器的图像以及同时使用熵过滤器和 Otsu 过滤器的图像。“带 Otsu 过滤器的分段试验”图像将是我们划痕试验分析的最终结果,让我们看看所有这些代码的输出:
正如我们在上面的图像中看到的,熵过滤器在原始图像中产生了巨大的变化,熵过滤器检测图像像素中的微小变化,并根据图像某些区域的复杂性划分像素。正如我们在第二张图片中看到的,黑色条带代表没有细胞的区域,而灰色区域代表有细胞的区域。Otsu 过滤器通过实现一个阈值来巩固这种分割,使我们能够在划痕分析的准确时刻准确地定位细胞的位置。

最后,我们可以打印代表检测中无细胞区域的像素数,以便更好地了解细胞是如何随着时间移动和增殖的。
这个具体的项目对于理解图像处理的一些基本方面非常有用,由于所有像素具有相同的 RGB 值,因此在本教程中分析的图像非常难以分割。然而,在熵过滤器的帮助下,我们能够生成一个基于纹理处理的图像,然后我们可以使用 Otsu 阈值来更深入地了解该时间点的细胞位置。
划痕分析由一个时间序列组成,我们只分析了该时间序列中的一张图像,生物学家在多个图像中多次循环此过程,以生成细胞运动的时间序列表示。当他们浏览每张图像时,生物学家会记录细胞运动和位置的变化,这为他们提供更多有关细胞增殖率和细胞迁移模式的信息。
许多图像处理过滤器需要复杂的高斯数学知识,而大多数人没有这方面的经验,但是 scikit-image 消除了实现此类过滤器所需的高级数学知识。然而,对高斯核和其他此类计算工具的了解,可以让研究人员获得预包装软件能够提供的更准确的可视化效果。
我在下面添加了完整的代码要点。
In [ ]:from skimage.filters.rank import entropyfrom skimage.morphology import diskfrom skimage.filters import threshold_otsufrom skimage import ioimport matplotlib.pyplot as pltimport numpy as npimport skimageIn [ ]:img = io.imread('images/scratch.jpg') # Reading Imageentropy_img = entropy(img, disk(10)) # Applying entropy filter for better segmentationIn [ ]:thresh = threshold_otsu(entropy_img) # Applying Otsu filter for optimal thresholdingIn [ ]:# Creating image in which all pixels with value less than threshold is equal to truebinary = entropy_img <= thresh # Composed of 0s and 1s (False and True)In [ ]:# Displaying Imagesfig, ax = plt.subplots(nrows=1, ncols=3, figsize=(12,10))ax[0].imshow(img)ax[0].set_title("Original Assay")ax[1].imshow(entropy_img)ax[1].set_title("Assay w/ Entropy Filter")ax[2].imshow(binary)ax[2].set_title("Segmented Assay w Otsu Filter")In [ ]:print(np.sum(binary == 1)) # the number of pixels that correspond to the clear space in the assay
往期精彩回顾 本站qq群851320808,加入微信群请扫码:
