iMeta | 北大陈峰组综述口腔微生物组的标准化研究:从技术驱动到假说驱动
口腔微生物组:从技术驱动到假设驱动

https://onlinelibrary.wiley.com/doi/10.1002/imt2.19

4.11
iMeta
REVIEW ARTICLE
● 2022年4月11日,北京大学口腔医院高楚琪等在iMeta在线发表题为“Standardized Studies of the Oral Microbiome: From Technology-driven to Hypothesis-driven”的综述文章。
● 近期研究聚焦于确定统一的微生物学临床诊断标准,应对个体差异造成的异质性。本综述依据是否有明确的研究假设,将现有研究分为技术驱动型和假设驱动型。这种划分方法揭示了口腔微生物学研究方向的转变。
● 第一作者:高楚琪
● 通讯作者:陈峰
(chenfeng2011@hsc.pku.edu.cn)
● 通讯作者:陈宁
(chenning79@139.com)
● 合作作者:李萱韬#,赵小乐#,杨沛岳,王晓,陈晓莉(#表示共同第二作者)
● 主要单位:
北京大学口腔医院(Peking University Hospital of Stomatology, Beijing, China)
北京大学人民医院(Peking University People’s Hospital, Beijing, China)
微生物群与宿主之间存在共生关系。在人体内众多微生物栖息地中,口腔微生物群尤为复杂。最近的研究不再反复确认口腔和全身疾病的生物标志物,而是聚焦于确定统一的微生物学临床诊断标准,以应对个体差异造成的异质性。研究者们也在进行其他具有更大临床意义的课题,包括细菌的致病机制、药物和治疗手段的疗效。本综述中,我们依据是否有明确的研究假设,将现有研究分为技术驱动型和假设驱动型。这种划分方法揭示了口腔微生物学研究方向的转变。基于这一转变,我们提出建立明确的假说可能是解决重大研究挑战的方法。
关键词:技术驱动,假设驱动,口腔微生物组,宏基因组学,致病机制
● 基因组学技术无疑推动了口腔微生物学研究的进程,增进了人们对疾病的了解
● 由基因组学技术驱动的研究倾向于获取并描述特定人群的微生物学画像
● 然而,最近的研究正在逐渐将目标具体化、明确化,揭示着全球研究进程已来到由技术驱动向假设驱动转变的关键转折点
● 目前假说驱动的研究主要聚焦于对致病机制的验证和对治疗方法的有效性评估上
Bilibili:https://www.bilibili.com/video/BV1Sr4y1n7Yg/
Youtube:https://youtu.be/ZzlkYW0pTco
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
引 言
微生物组被视为第二个人类基因组,我们与它之间存在一种共生关系。纵观生命科学的发展历程,我们才刚刚开始揭示这种关系的性质和强度,以及它对生理学、病理学的影响。在人体内的众多微生物栖息地中,口腔微生物群尤为复杂,其多样性位居第二。口腔是连接外与消化道和呼吸道,其内的微生物可以抵御外界有害因素。菌群失调与口腔疾病相关,如牙周炎、种植体周围炎、口腔黏膜疾病和龋齿,也与全身性疾病有关,如胃肠道、内分泌、免疫和神经系统疾病等。
由于一些疾病缺乏公认的驱动因素(如吸烟、饮酒和人乳头瘤病毒),口腔微生物作为一个潜在风险因素开始引起关注。此外,认识口腔微生物组也有助于解释其他风险因素在疾病发展中的作用。
随着下一代测序(NGS)技术的普及,基于测序的口腔微生物学发文数量达到了一个前所未有的水平,这无疑是人类口腔微生物学一次巨大的突破。而口腔微生物学发展至今,其研究方向出现了一个重要转变。
技术驱动型研究的最新进展
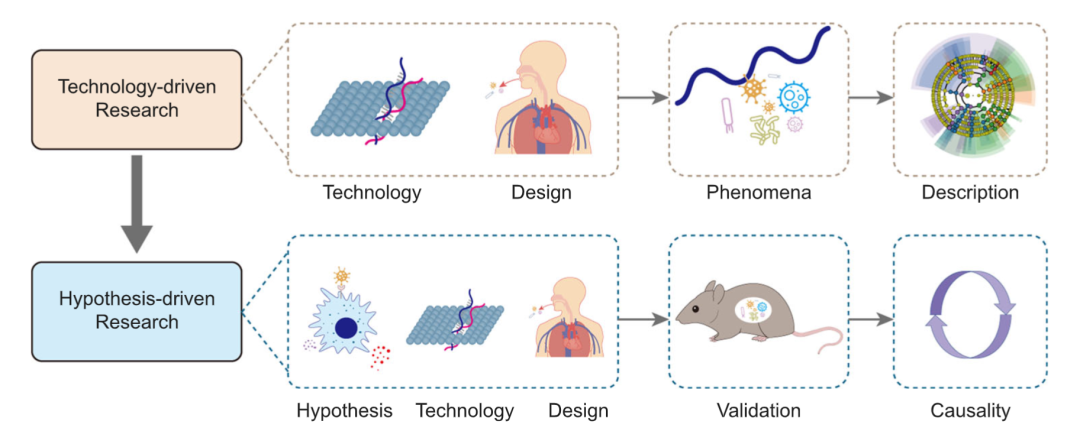
描述疾病的微生物学特征有助于推断其发病机制,即由现象推断因果关系(图1)。技术驱动型研究的重点包括数据库的更新、新的测序技术和方法,以及确定新的目标人群和新的疾病相关性。

图1. 技术驱动型研究是指描述性的研究
也就是说,此类研究利用NGS技术获取并描述特定人群的微生物学画像。而通过设计实验来进行科学假设的验证,从而得出因果关系的,称为假设驱动型研究。
数据库的更新
人类口腔微生物组数据库(HOMD)是基于16S rRNA基因参考序列的宝贵学术资源。扩展的人类口腔微生物组数据库(eHOMD)涵盖了栖息在口腔和鼻腔中的众多微生物物种。然而,由于进化关系相近的物种间16S序列高度保守,在注释和分类中区分它们有所难度。为解决这一问题,属和种之间出现了一个新的分类水平"超种",由共享相似序列的物种组成,减少了16S rRNA短读和长读的错误率。
然而,HOMD来自于微生物培养获得的数据。受培养条件和其他因素作用的影响,HOMD中20-60%的口腔微生物是不可培养的。而借助对从口腔分离的细菌进行宏基因组测序,一些无法培养的口腔微生物正在逐渐被人们认识。有研究者利用3346个口腔宏基因组的鸟枪法测序数据和808个公开的样本数据,获得了56213个宏基因组数据。3589个物种水平的基因组箱(bin)中超过64%含有未公开发表过的基因组。这项研究为数据库的补充做出了重要贡献。
用舌和牙菌斑样本重建790个非冗余基因组发现,其中43个属于TM7家族,可归为6个进化枝。TM7家族的多样性及其与口腔黏膜感染性疾病的联系此前已有报道。此外,在47个人类样本中发现了42个TM7的特异性新位点(斑块与舌头),其中包括Saccharibacteria的G3和G6进化枝。Qi等人从一个儿童的口腔中分离出C17T的宏基因组。Beall等人则使用单细菌细胞的DNA扩增和测序获得了几乎完整的Tannerella sp. BU045的基因组。一项基于PromethION的长读宏基因组研究在口腔环境中首次鉴定出多达10个大型口腔噬菌体/原噬菌体(包括质粒样成分)。
除了寻找新的分类群,还有研究对不可培养的细菌进行宏基因组组装。一项大样本的大规模宏基因组数据还可以组装重要的口腔菌株,如Porphyromonas和Neisseria。组装后的重叠区中显示,50%的基因是单个宏基因组样本所独有的。这揭示了微生物组作为一种人类表型无法解释的异质性。
还有一些新的细菌先前已被分离出来,但其功能尚不清楚,如Streptococcus sp. A12。有研究通过功能基因组学确定了这种微生物抵御拮抗因素的分子机制,促进了生物标志物和治疗药物的开发。Saccharibacteria门显示出与口腔黏膜感染性疾病之间的相关性。有趣的是,许多口腔微生物群含有与肠道细菌同源的编码酶,更与多达41种人类用药有关。总之,HOMD会随着新发现的源源不断而持续更新(图2)。
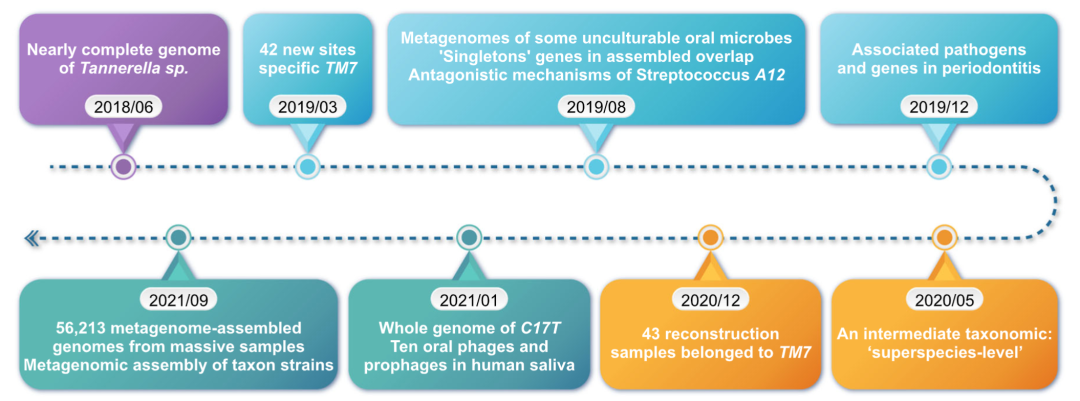
图2. HOMD仍在不断扩大和更新
图为2018年6月至2021年9月的数据库发展情况
新方法和新技术
在这一领域,研究方法的优化和改进至关重要。Yano等人比较了使用四种采集方法采集的人类口腔微生物样本,发现采集方法对口腔微生物组有明显影响。为了克服这种差异性,一种能从人类口腔样本中富集更多微生物DNA的新方法诞生了——渗透裂解后再进行叠氮溴化丙锭(PMA)处理。这一方法还可能扩展至其他样本类型。
与基于参考的16S rRNA基因分析相比,一种更高分辨率、更低成本的测序方法,能够通过结合高多样性标记基因(核糖体16-23S的基因间隔区)的扩增子测序与基于概率误差建模的降噪算法DADA2,实现微生物群落分析分辨率的显著提高。
此外,口腔细菌基因组学的数据统计方法也有新的进展。作为一种相对成熟但尚未广泛应用于口腔微生物领域的方法,共现网络分析已被证实可以应用于分析口腔与其他部位微生物组的关联,以及口腔微生物组与代谢产物的潜在关系。同样,基因组规模的建模也已应用于人类特定部位微生物组的研究。
有研究者采用机器学习算法证实,颊黏膜和龈上间隙的口腔微生物组谱可一定程度上表征龈下斑块的微生物组成。这些微生物群有希望成为早期诊断牙周病的标志物。
迄今为止,多项新技术已经逐渐应用于口腔微生物研究。在16S rRNA基因测序技术的基础上,人类口腔微生物鉴定微阵列技术(HOMIM)可以同时检测大约300种常见口腔微生物,包括那些尚不能在体外生长的细菌种类。HOMI NGS是一种相比HOMIM更全面的半定量技术,可以更好地实现口腔微生物的表征。
然而,16S rRNA基因测序仅限于16S rRNA片段,而鉴别微生物应考虑整个基因组。多组学技术(如基因、转录、蛋白组学)已经应用在口腔微生物基因组中。结合人类基因组计划(HMP)与宏基因组数据的超基因组学方法下,人们发现了舌背、颊黏膜及龈上菌斑分布的微生物群差异。另有蛋白质组学和代谢组学分析表明,牙菌斑和牙石之间存在着系统的微生物差异,这与生物膜的生理学有关。多组学技术与微生物学研究正在越来越密不可分。
传统的体外动物模型和基因组学的结合尚有很大潜力。成年猕猴模型被用于研究牙周炎进展过程中口腔微生物组与牙龈细胞凋亡、自噬和缺氧通路基因表达模式的相互作用关系。粪便移植已被用于研究肠道微生物,提示了一种将患者的口腔细菌移植到无菌小鼠的新实验设计思路。将健康口腔的微生物群落复制或移植给患者,可能不失为一种治疗口腔疾病乃至全身疾病的潜在新疗法。但到目前为止,对这一新疗法的微生物学评估和临床结果报道仍是空白。
由此可见,新的研究方法、技术及其应用放大了人类观测微生物视野。将组学技术与其他技术的结合,或是将其它领域的优秀技术应用于口腔微生物学研究,都或可为新的研究阶段拉开序幕。
新的研究人群
对不同流行病学特征下人群的口腔微生物组基线水平的研究已经取得了一些结果,扩大了我们对口腔微生物的理解(图3)。
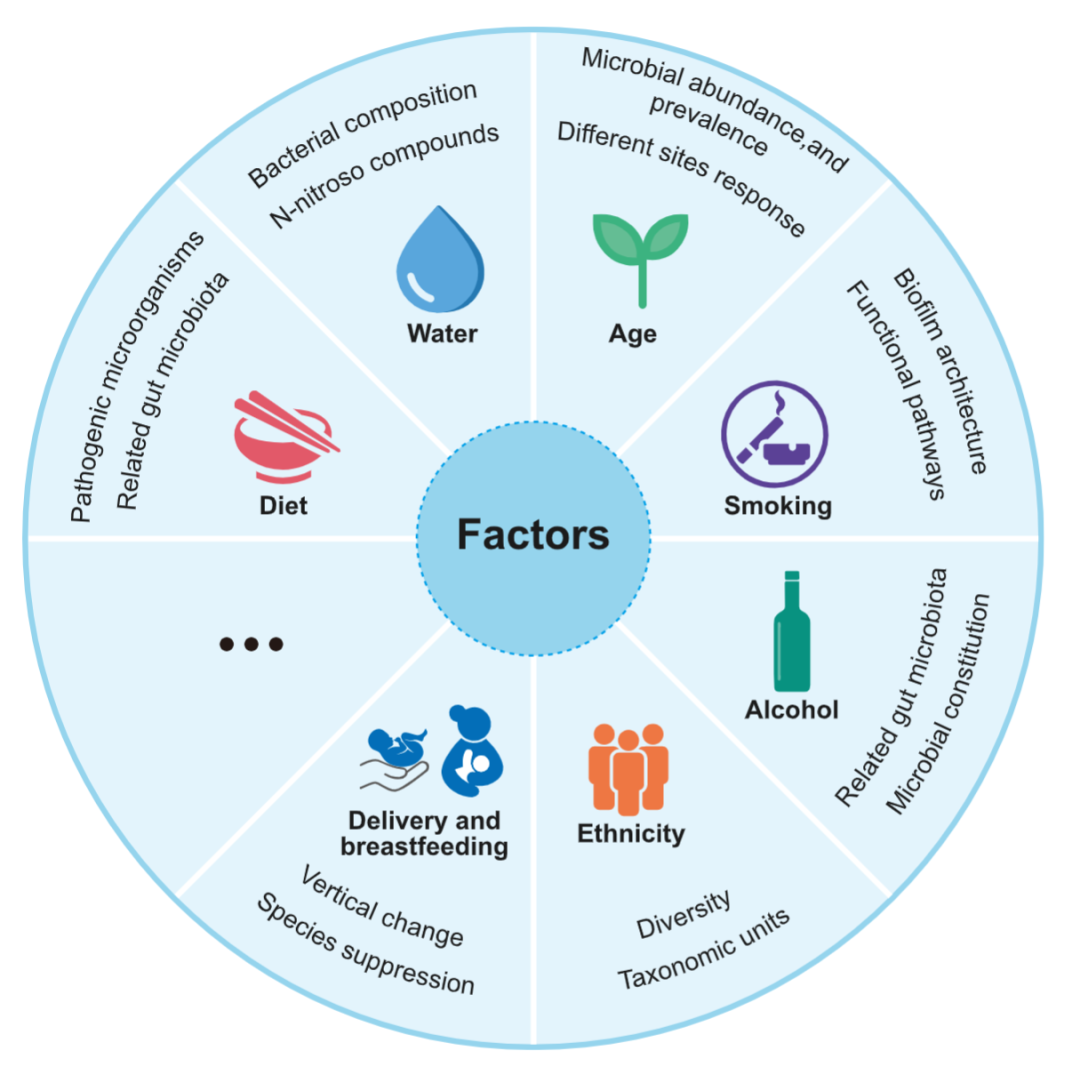
图3. 口腔微生物的影响因素研究还在继续,包括年龄、吸烟、酒精、种族、饮食、水、分娩和母乳喂养等
上述研究都是使用下一代测序技术进行的。不过许多研究不再局限于观察多样性和物种分布,而是逐渐向结构性、机制性或纵向的角度发展,使人们得以更清楚地了解这些因素背后的临床意义 。
口腔微生物组与年龄具有精确且可观的关联性,包括数量、丰度和流行率。不同位点的微生物群落对衰老的反应呈现钟形趋势。不同地区的人类群体在个体之间和个体内部的多样性程度上都有很大差异。在加拿大的人群中发现了28个物种级别的操作分类单位(OTU)在不同的种族群体中具有明显差异。
吸烟者和电子烟使用者的口腔中都有丰富的病原体,其最重要的影响是改变了生物膜结构。吸烟也可能导致相关疾病功能途径的转变。饮酒的水平和类型与整体微生物成分和单个分类群的丰度有关。口腔微生物组的香农多样性比肠道低。饮用水的成分也可能影响塑造口腔微生物群,有研究证明其与几个菌属丰度的主要变化有关。饮用高硝酸盐的水会增加口腔硝酸盐还原菌的含量,这可能导致N-亚硝基化合物的增加。
素食者和杂食者的唾液微生物群组成的差异存在于系统级以下的所有分类水平,包括与牙周病有关的物种。口腔和肠道微生物群与特定的饮食成分相关,如蔬菜和甜食的摄入。有研究证实了分娩方式与口腔微生物组中原始细菌含量之间的关系。出生后两年内,母乳喂养时间较短和抗生素处理与成长后期的细菌组成特异性有关。3个月的婴儿中,母乳喂养和配方奶粉喂养的婴儿的口腔微生物群有所不同。
人类的口腔微生物特征受到多种因素的影响,但其基本机制尚不清楚,例如关于肥胖者与非肥胖者的唾液微生物组差异的研究。Džunková等人对接受减肥手术的肥胖成年人的唾液样本进行了纵向分析,发现除体重以外的多个个体特异性因素影响了唾液微生物组的分布。对口味偏好的改变和潜在的口腔健康缺陷之间的相关性进行进一步研究是个合理的思路。还有其他一些因素也不能明确与微生物组的因果关系。与现代生活方式相关的各种习惯可以减少人类口腔微生物组的多样性,如高糖饮食、饮酒、吸烟等。再比如,母婴微生物组传播的病理过程仍不清楚。此外,吸烟者和电子烟使用者的口腔微生物群含有病原体,但其细菌组成不同。这些研究反映了人类微生物组中无法解释的异质性。接下来有必要解释这种异质性是如何产生的,解决办法之一是继续扩大研究人群,包括迄今为止被忽视的群体,如青少年和婴儿。
疾病的新描述
一些疾病与口腔微生物的关联已经更新,如阿尔茨海默病(AD)、帕金森病(PD)、人类免疫缺陷病毒(HIV)感染和妊娠期糖尿病(GDM)。
最近发现AD和口腔微生物群之间有很强的相关性。其中一个原因可能是AD患者通常会出现认知障碍,导致口腔运动能力差,从而缺乏进行口腔护理的能力,增加了患牙周炎和龋齿的风险。Holmer等人报告说,龈下微生物群在认知障碍或AD患者中表现出典型的牙周病特征。Fleury等人对唾液和龈下斑块的微生物群进行了采样,利用16S rRNA基因扩增子测序,发现早期和中期PD患者的口腔微生物群发生了变化,这可能与口腔的局部炎症有关。Xu等人通过16S rDNA测序分析发现第三孕期下的GDM与口腔微生物之间存在联系。肠道和口腔微生物群落的变化可作为监测妊娠期GDM的非侵入性生物标志物。这些研究一个共同的局限性是,它们关注的是相关性而不是因果关系。不过,尽管这些疾病与口腔微生物群之间的因果关系尚未得到确证,这种相关性本身也有助于人们对疾病进行诊断与预防。
还有一些疾病的研究也存在着局限性,如不良妊娠结局(APO)。目前与APO的免疫过程有关的临床证据还依赖于横断面病例对照研究。然而,妊娠期包括促炎症和消炎症两个阶段,这两个阶段都受到女性性激素波动的影响。这使得此类研究在未来的机制探索中缺乏潜力。
另一方面,基因组学技术为人类描摹出更加清晰的人类口腔微生物基因图景,帮助人们更加清楚地认识疾病。技术研究人员从尚未被命名的物种中识别出数千个微生物基因组,扩展了人类相关微生物的基因图景。通过宏基因组测序和微阵列分析,研究人员从20个受试者数据中首次获得了健康口腔微生物组(HOM)及抗性组的全面视图。利用25名3-5岁患严重龋齿的学龄前儿童的样本和19名年龄匹配的健康对照组,首次构建了患龋儿童的微生物组基因目录。同样,16S rRNA测序还提供了根尖周炎微生物群落和唐氏综合征患者口腔微生物组的首个全面快照。研究者借助生物地理学变化的已知知识,确定了龈下社区组成的从前到后的梯度。然而,这些研究的样本量仍然不够大,需要更大的样本量来证实上述结果。
新的诊断工具和模型
个体之间的差异限制了以往的研究得出微生物组的普遍结论。这意味着仅凭个体微生物的分布异常预判疾病并不可靠,需要一个统一的标准。
然而,遗传多样性尚未得到全面量化。Tierney等人对来自口腔和肠道两个人体微环境的基因组进行了跨研究的荟萃分析,涵盖了来自13个研究的3655个样本,鉴定出总共45,666,334个非冗余基因(23,961,508个来自口腔,22,254,436个来自肠道),该资源可通过https://microbial-genes.bio访问。
基于更大的样本量,一些大规模研究陆续提出了疾病的口腔微生物诊断工具和预测模型。大样本量和一些统计方法削弱了个体差异对结果的干扰,这就是标准化过程。只有实现个体差异的标准化,疾病诊断工具和预测模型的可靠性才值得信任。原发性斯约克伦综合征和系统性红斑狼疮患者之间的口腔微生物群组成存在显着差异,提示可以此区分二者。Xu等通过16S rDNA测序对144名3岁儿童龋齿的发生和发展进行了1年的纵向观察,其中包括对照组的10名患龋儿童和19名健康儿童,他们得到的预测模型准确性高达93.1%。考虑到龋病的高患病率,微生物学诊断工具具有重要意义,这需要我们继续对个体差异标准化进行探索。
技术驱动型研究的最新进展
在已有理论和科学经验的基础上提出一个可能合理的假说,并通过实验或临床验证,是研究的关键步骤(图1)。新细菌的分离、新研究人群的识别、以及疾病与口腔微生物组之间的联系,共同为假说驱动的研究奠定了基础。通过进行验证性实验,假说驱动型研究可以得出疾病的微生物学或免疫学机制、治疗效果等因果性结论。
致病机制中的口腔微生物组
牙菌斑及其与宿主之间代谢和功能上复杂的相互作用参与了健康维持机制。Wang等人纵向跟踪了人类口腔生物膜在遭受干扰后的重新组装过程,揭示了强烈干扰后口腔微生物群的恢复和长期稳定性,并确定了群落变化和结构恢复最明显的关键时间点和阶段。
很多口腔疾病已被证实与口腔微生物的代谢有关,如龋齿、牙周炎和口腔粘膜疾病。人们对口腔疾病的认识从单一致病菌理论向微生态失衡理论转变。微生态失衡是微生物群中细菌、真菌和病毒相互作用的结果。例如,在严重的儿童早期龋齿发病机制中,牙菌斑中葡萄糖基转移酶的活性被白色念珠菌明显提高。
基因组学技术已广泛应用在针对致病机制的免疫学研究中,挖掘出一些以前认为与牙周炎无关的新推定病原体和新基因。一项纵向研究提供的证据表明,参与碳水化合物相关代谢(如甲烷代谢、能量代谢)的基因,在晚期口腔鳞状细胞癌中各项指标都更高,而负责氨基酸代谢的基因则与健康对照组明显相关。对唾液微生物群进行分级聚类分析,发现口味偏好相关的基因及其等位基因的突变与蔗糖摄入双向有关。最近的生物信息学研究探索了产生许多小分子的生物合成基因簇,突显了人类微生物组编码的信号分子具有令人难以置信的生化潜力。例如,Uranga等在甘油条件下培养并检测R.mucilaginosa和R.dentocariosa产生的儿茶酚类铁载体合成基因簇,发现Rothia Mucilaginosa在健康儿童的唾液中富集。Mohan等人则检查了现有的元转录组数据集,以识别通常差异表达的转录本和代谢转变背后潜在的RNA调控机制。这些对基因及其转录过程的研究可能为个体口腔的共性和多样性提供了全新的见解,以期建立一个更全面的口腔微生物图景。
牙周疾病与口腔微生物组有关已成为共识。炎症作为宿主免疫状态失衡的结果,又为病原体提供了适宜的营养环境,进一步促进失衡的持续存在。因此,炎症和失衡会以自我维持的方式相互促进,成为牙周病的驱动因素。作为炎症反应,牙周病是一个体现了微生物与宿主之间相互作用的典型动态过程,后续研究应更加关注这种动态性质,以更好地了解疾病的发病机制。
口腔菌群失衡可能会导致胃肠道、内分泌、免疫和神经系统的疾病。症状性慢性牙周炎是慢性牙周损害的一种形式,被认为是全身性疾病的表现之一,通常与影响牙周结构或宿主免疫防御关键要素的主要基因缺陷有关。另外,牙周炎也有可能通过口腔微生物失衡引发菌血症,进而导致全身性疾病。因此,口腔微生物群和全身性疾病之间存在着双向互动。
目前,在细菌转移的相关理论上,全身性疾病与口腔微生物组的因果关系得到了加强。Medini等人评估了炎症和免疫功能异常的细胞和可溶性标志物,发现细菌和真菌口腔微生物组均可能在HIV感染患者的慢性全身免疫激活中起作用。Moentadj等进行的前瞻性队列研究在RA患者及其一级亲属、健康对照者的的口腔微生物群中发现,来自RA的和HC相关的S. parasalivarius菌株的链球菌细胞壁在小鼠中诱发了类似的轻度慢性关节炎。
口腔微生物组可能通过作用于肠道来驱动全身性疾病的发展和恶化。例如,口腔微生物组在肝硬化患者的下肠道中占比过高,有可能导致疾病的发展和加重。肠道炎症是一些全身性疾病的公认致病因素,例如关节炎、银屑病和葡萄膜炎。然而,其机制究竟是口腔炎症蔓延,还是口腔细菌的直接作用尚不清楚。
牙周炎对人体内的肠道炎症有促进作用。先前已有研究发现,Th17细胞缺陷与牙周炎症和骨质流失减少有关。Kitamoto等人使用小鼠牙周炎和肠炎模型表明,牙周炎在口内产生的反应性Th17细胞迁移至肠道会引发结肠炎。另外,Klebsiella spp.还是Th1细胞的强力诱导剂。
口腔充当了潜在的肠道病原体储存库,口腔-肠道轴的微生物转移途径是一个受到广泛认同的假设。一个口腔-肠道细菌运输的多阶段模型强调了口腔-肠道轴中微生物学和免疫学区域划分的重要性。根据Abed等人的一项关于口腔化脓性细菌转运到结肠肿瘤的研究,血行转移可能是最主要的转移方式。摄入或移植到肠道的Klebsiella和Enterobacter可能激活结肠单核吞噬细胞的炎症反应 (图4)。
各种生物和环境因素影响疾病的易感性、进展情况和严重程度,但关于这些影响的纵向研究还很少。Freire等人通过纵向研究对同卵双生和单卵双生的龈上菌斑微生物组进行了分析,发现口腔微生物组的变化主要由环境引起。另外,与经阴道分娩的婴儿相比,剖腹产的婴儿最初细菌含量异常,但随着年龄的增长而恢复。这一发现表明,可将口腔微生物的发展视为一场生态演替。
Surana等人发明了一种简单的生物信息学方法,对可能影响疾病发病机制的微生物群成员进行三角测量,使测序方法得以符合科赫定理。Sanna等人和Zhuang等人则采用了双向孟德尔随机化分析来评估因果关系。这些方法可进一步用于证明口腔微生物组与疾病之间的因果关系。
值得单独一提的是,宏基因组测序显示口腔微生物组与癌症明显相关,这意味着癌症有了新的预防方法。其可能的机制包括上调与膜生物合成、鞭毛合成和组装、铁运输、趋化性、溶血素和粘附素有关的毒力因子。此类癌症相关的研究目前主要集中在口腔、结直肠、胰腺、肺和食管癌。
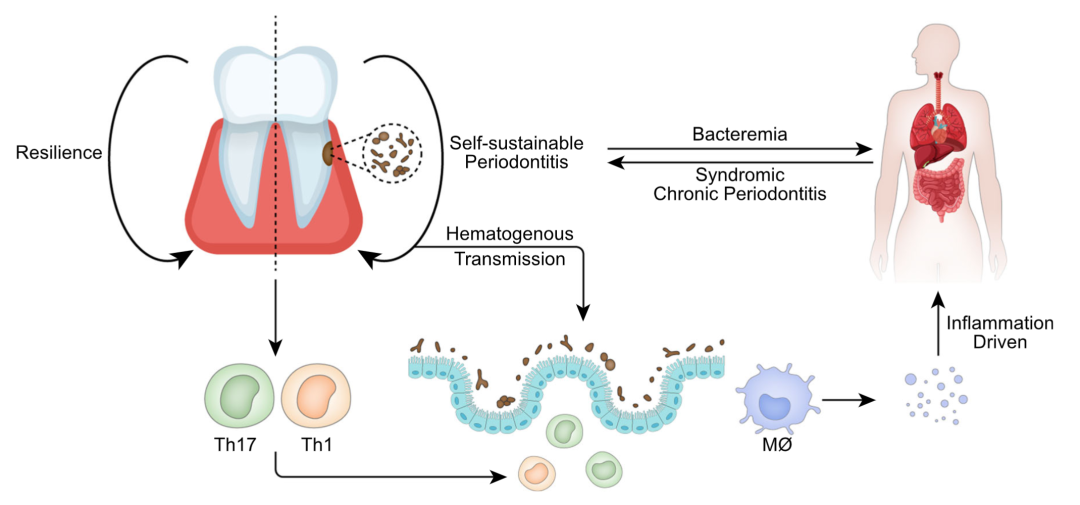
图4. 口腔疾病与全身性疾病关联的潜在机制
健康的口腔通过复原力维持,而口腔炎症则是自我维持的,并与全身性疾病呈双向驱动关系。其机制可能是牙周细菌可以通过血液循环进入肠道引发炎症,或者可以诱导口腔内的Th17、Th1和其他细胞进入肠道。炎症产生的炎症因子借由免疫反应来诱发全身性疾病。
疾病治疗中的口腔微生物组
炎症性肠病是包含口腔-肠道的微生物转移途径的典型常见疾病,可采用微生物失衡相关的药物治疗。Simon-Soro等最近的一项研究表明,口腔和肠道菌群在药物(例如通佐溴胺)的干预下彼此不受影响,但口腔菌群改变可能导致口腔特定菌群出现在肠道中。一个可能的解释是:虽然药物对菌群的干预具有原位性,但机体全身肠道菌群的互通依然会传递药物的治疗作用。这一结论很好地支持了未来通过建立健康口腔治疗胃肠道疾病的可能性。另一项研究使从唾液微生物群分离出的菌群在肠道内定植,发现Klebsiella对多种抗生素具有抗性。鉴定出能够抗口腔细菌定植的健康肠道菌群成员,可能会为未来针对耐药细菌的药物开发和慢性炎症的有效治疗方法开辟新的途径。
口腔微生物还可一定程度上指导癌症治疗。急性骨髓性白血病(AML)患者接受碳青霉烯类药物后,其粪便和口腔样本多数丧失了α多样性。在一项对90名AML患者的前瞻性队列分析中,没有发现S. maltophilia感染与其在粪便中相对丰度的关联。相反,口腔微生物组对AML化疗患者的S. maltophilia感染有预测作用。研究还指出,累积接触美罗培南与S. maltophilia感染风险增加有关。该结论有助于癌症治疗中广谱抗菌药物的疗效与间接损害的权衡。
口腔粘膜炎(OM)是化疗中最常见的并发症之一。在癌症治疗中,动态的口腔微生物群落可以诱导口腔黏膜炎。一项针对头颈部鳞状细胞癌患者的研究显示,治疗过程中微生物属的丰度变化与严重OM的发作有关。Hong等人通过纵向评估了一个周期内接受基于5-氟尿嘧啶或多柔比星的化疗的49位受试者的宏基因组数据。结果表明,化疗引起的OM和微生物群失衡密切相关,并且炎症相关的不良生物转变会加剧口腔黏膜上皮的损伤。他们推测控制口腔微生物群失衡是改善口腔粘膜炎的一种新型预防方法。
总之,得益于技术驱动的研究成果建立的庞大数据库,人类对口腔微生物拥有了前所未有的高度认识。而之后假设驱动的研究应聚焦于科学问题,将这些微生物学知识转化为重要工具应用于临床诊断。
总 结
目前,口腔微生物学研究已经遇到了瓶颈。最近的研究不同于更早的大部分研究,没有再对口腔疾病和全身性疾病特定生物标志物进行反复确认,而是期望找到统一的微生物学临床诊断标准,减少个体差异带来的不稳定因素。同时将研究重心转到一些更具临床意义的问题,例如细菌致病的机制,以及治疗的效果。在我们看来,口腔微生物学研究有以下两个主要挑战。
第一,是对假设驱动而非技术驱动的研究的需求。技术驱动型研究缺乏明确的假设,只是单纯探索空白领域,从而可能得出矛盾或缺乏临床意义的结论。相对而言,假设驱动型研究建立在过去20年积累的庞大数据库之上,又有具体的研究目的,因而可能更能显示出临床意义。因此,微生物学研究应该是假设驱动的,而不是技术驱动的。这种划分方法是首次被提出,它清楚地展示了口腔微生物学研究方向上的一个重要转变。
另一个主要挑战是样本量不足。NGS技术使得大型数据库的建立成为可能,同时过程中也揭示了明显的个体差异性。因此,标准化在处理新创建的数据集的时候至关重要。之前口腔微生物学的综述大多忽略了标准化,还有一些只在概念层面上提及。我们特别强调标准化的重要性,更应在未来的研究中伴随明确的假说。
口腔微生物对组织、器官和系统的影响牵一发而动全身,一方面可能干扰对其对单一疾病影响的判断,另一方面则可能给药物治疗提供了新的思路。因此,不管从预防、诊断还是治疗来看,口腔微生物组都具有强大的潜力。然而,与肠道微生物组相比,人们对口腔微生物组实在缺乏关注。虽然二者有所关联,但肠道微生物的很多现象可能并不适用于口腔微生物。因此,本综述的宗旨是给予那些希望以最合理有效的方式来研究口腔微生物组的研究人员指导,以加快口腔微生物学的研究的进程。
引文格式:
Chuqi Gao, Xuantao Li, Xiaole Zhao, Peiyue Yang, Xiao Wang, Xiaoli Chen, et al. 2022. Standardized Studies of the Oral Microbiome: From Technology-driven to Hypothesis-driven. iMeta 1: e19. https://doi.org/10.1002/imt2.19
高楚琪
● 北京大学口腔医学院,2018级本科生
● 在微生物学领域从事科研工作2年,研究方向:口腔微生态与疾病研究,微生物宏基因组学。本科阶段发表SCI二区论著一篇,统计源、北大、CSCD核心期刊综述一篇,另有待审查实用新型专利一项。
陈峰(通讯作者)
● 北京大学口腔医学院,副研究员,博士生导师
● 2006年毕业于清华大学生命科学学院并获得理学博士学位,2007-2010年在美国加州大学洛杉矶分校牙医学院从事博士后研究,2011年至今在北京大学口腔医学院工作,负责中心实验室微生物平台。目前担任中华口腔医学会口腔生物医学专委会委员、口腔科研管理专委会青年委员及北京生物工程学会人体微生态分会常委。作为第一/通信作者已发表学术论文逾百篇,包括J Dent Res、Adv Sci、STTT等国内外权威杂志(2018年发表于Protein Cell的“Oral microbiomes: more and more importance in oral cavity and whole body为ESI高被引论文),并受邀作为iMeta期刊编委、J Dent Res、Ebiomedicine、IJOS等期刊审稿人,Front Pharmacol杂志客座编辑。主持国家自然科学基金项目3项,省部级科研基金项目2项。课题组主要研究方向为口腔微生态与疾病研究(微生物宏基因组学与唾液蛋白/多肽组学),以及口腔遗传病(唇腭裂)机制研究。
陈宁(通讯作者)
● 主任医师,副教授,现任职北京大学人民医院消化内科副主任、免疫介导消化疾病临床研究中心副主任,北京大学博导
● 研究领域:炎症性肠病,肠道微生态,肠粘膜屏障功能。先后承担包括3项国家自然科学基金在内的10余项科研项目,在国内外专业学术期刊发表论文50余篇,以第一完成人获得国家发明专利1项,参与编撰专业书籍10余部。学术兼职:中华医学会消化分会青年委员,中华医学会消化分会炎症性肠病学组委员,中华医学会消化病学分会疑难重症协作组委员兼秘书,北京医学会消化分会委员,北京医学会消化分会第一届青年委员会副主任委员。北京医学会肠道微生态及幽门螺杆菌分会委员,iMeta期刊青年编委、Gut中文版胃肠专刊青年编委,JCC中文版编委等。
(▼ 点击跳转)
iMeta文章中文翻译+视频解读
▸▸▸▸
iMeta|西工大钟杨权威等-根系菌群沿环境梯度的响应机制
▸▸▸▸
iMeta | 华中科大宁康组综述用于蛋白质结构预测的宏基因组定量分析
▸▸▸▸
iMeta | 中科院李小方等膳食甘草促进小鼠镉解毒并调节肠道菌群代谢
▸▸▸▸
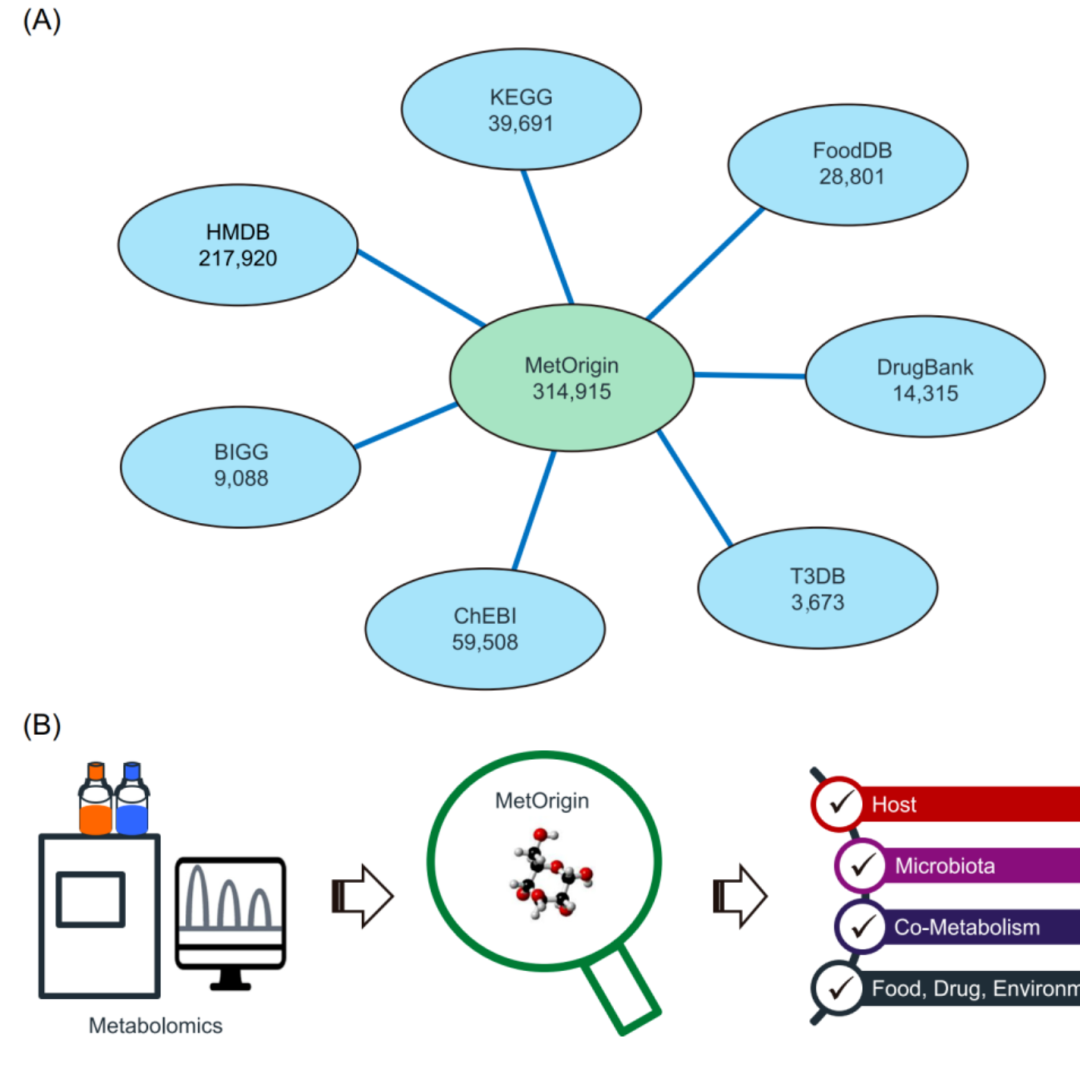
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
▸▸▸▸
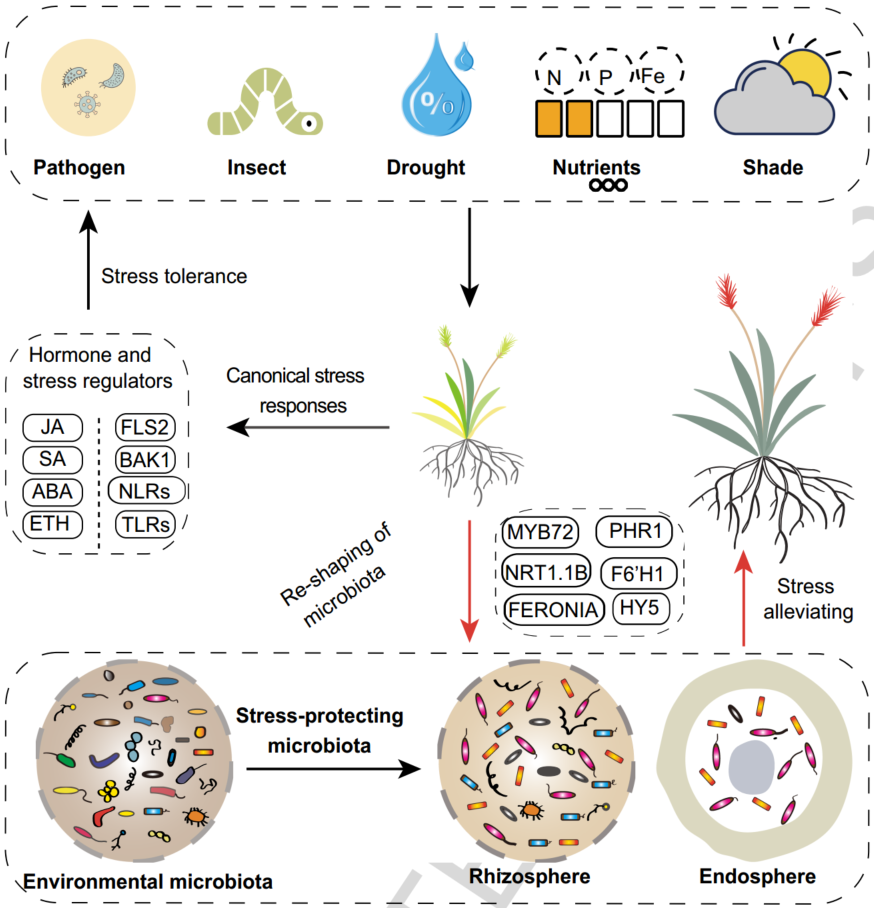
iMeta | 南科大宋毅组综述逆境胁迫下植物向微生物组求救的遗传基础(附招聘)
▸▸▸▸
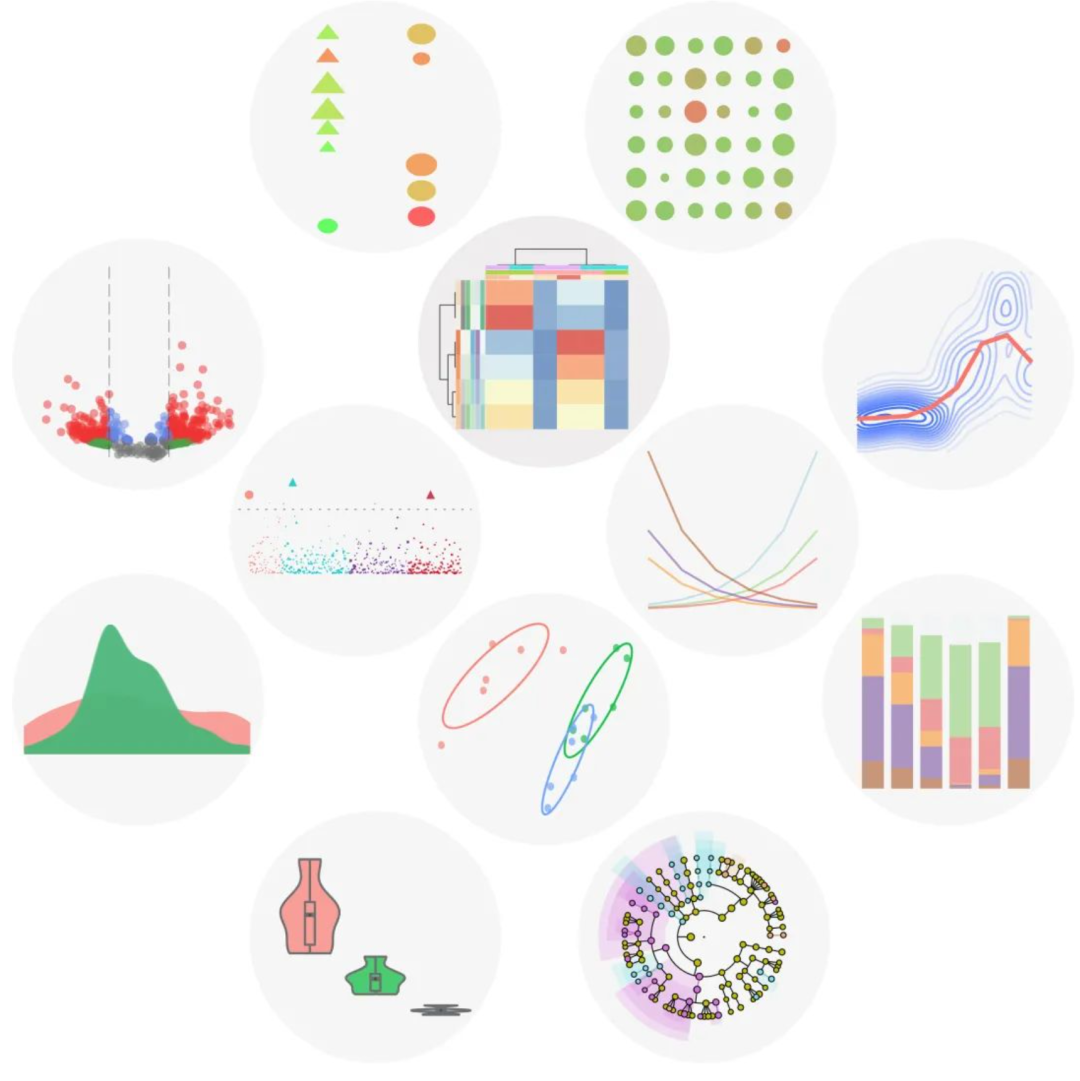
iMeta:高颜值高被引绘图网站imageGP
iMeta教你绘图
使用ImageGP绘图热图Heatmap
▸▸▸▸
使用ImageGP绘图富集分析泡泡图

“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
微信公众号
iMeta
责任编辑
微微