TransBTS_3D多模态脑肿瘤分割Transformer阅读笔记
文章目录
1 前言 2 TransBTS 整体结构概述 3 Network Encoder 4 Network Decoder 5 实验部分 6 总结 7 参考链接
1 前言

这是医学图像处理系列的第三篇文章,arXiv 链接在文末,为什么选择写下 TransBTS 这篇论文的阅读笔记?因为才有时间整理了。此外,与之前的两篇相比,它们都有各自的代表性。第一篇(https://zhuanlan.zhihu.com/p/505483978)是 2D Transformer 医学图像分割,第二篇(https://zhuanlan.zhihu.com/p/506716802)是轻量级网络 2.5D 的,在这一篇阅读笔记中,讲解的则是 3D 多模态脑肿瘤分割 Transformer,也接受在 MICCAI 上。后面文章过多的话,我会整理成目录的形式。
Transformer 由于自注意力机制具有捕获全局依赖特征的能力,且允许网络根据输入内容动态收集相关特征,在自然语言处理和 2D 图像分类方面取得了成功。
然而,局部特征和全局特征对于 dense 的预测任务同样重要,尤其是对于 3D 医学图像分割。那么在 3D CNN 中利用 Transformer 是值得关注的,这篇文章中提出了基于编码器-解码器结构的新型网络 TransBTS,BTS 指的是 MRI 的脑肿瘤分割。
为了获取局部的三维上下文信息,编码器首先利用 3D CNN 提取三维空间特征图。同时,对特征映射进行了细致的改造,将处理后的向量(token)输入到 Transformer 中进行全局特征建模。解码器利用 Transformer 嵌入的特征,进行渐进式上采样来预测详细的分割图。
本篇阅读笔记首先对论文进行概述;再详细介绍论文提出的 TransBTS 结构,设计一个神经网络,通过利用 Transformer 来有效地对 3D 医学图像的空间和深度维度上的局部和全局特征进行建模;最后分析了实验和分割可视化部分,并简单的总结。
2 TransBTS 整体结构概述
这部分我们直接看 TransBTS 的整体结构,然后再分析具体细节(分别看 Encoder 和Decoder)。关于 Transformer 在医学图像上应用的细节,可以在第一篇阅读笔记中发现。
对于给定输入图像 X,尺寸为 C×H×W×D,空间分辨率为H×W,深度维度为 D(切片数)和 C(模态数),首先利用 3D CNN 生成了捕获空间和深度信息的紧凑特征图(下采样),然后利用 Transformer layer 在全局空间中对长距离相关特征进行建模。
之后,重复叠加上采样和卷积层,以逐渐产生高分辨率的分割结果,因为,高分辨率是医学图像必须的特性。如下图,这里我们先不关注具体细节,网络结构还是很清晰的。
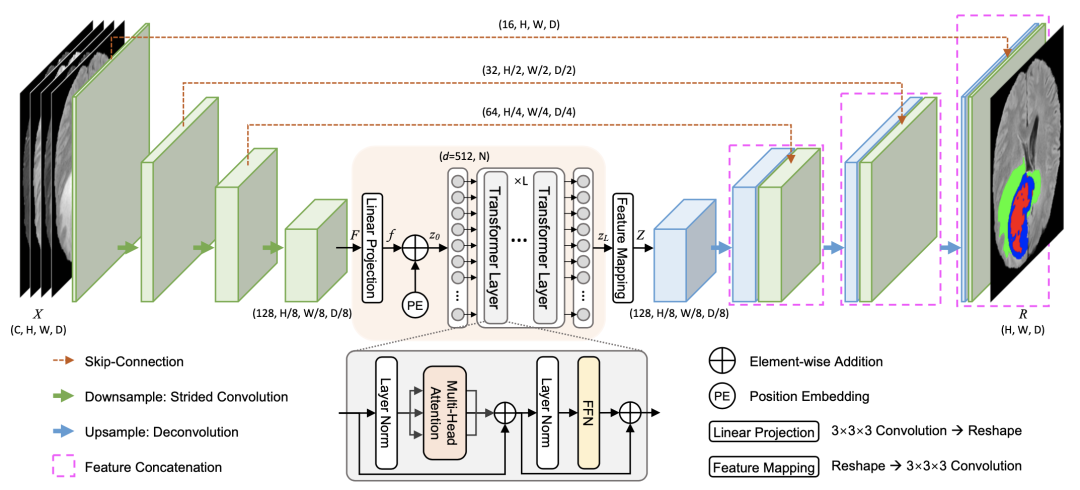
3 Network Encoder
这部分我们先看一下网络的编码器部分,有一些细节需要介绍。由于 Transformer 的计算复杂度是 token 数量(即序列长度)的二次方,因此直接将输入图像展平为作为 Transformer 输入的序列是不切实际的。
因此,ViT 将图像拆分为固定大小 16 × 16 的 patch,然后将每个 patch 作为一个 token,将序列长度减少到 16 的平方。同理,对于 3D 图像数据,则转换为 3D patch 就好(为了方便下面理解,也就是说输入的特征图多了深度)。
然而,上面的策略使得 Transformer 无法跨空间和深度维度对图像局部上下文信息进行建模以进行 3D 分割(局部 3D 上下文特征获取难)。
为了解决这个关键问题,这篇论文中的解决方案是使用下采样(stride=2 的卷积)堆叠 3 × 3 × 3 卷积块,以逐渐将输入图像编码为低分辨率的高级特征表示 F,尺寸为 K × H/8 × W/8 ×D/8 (K = 128),既 H、W 和 D 是输入维度的 1/8(总步长 = 8)。
通过这种方式,丰富的局部 3D 上下文特征有效地嵌入到 F 中。然后,将 F 输入到 Transformer 编码器中,以进一步学习全局感受野的相关特征。
关于论文中的 Feature Embedding of Transformer Encoder。给定上面提到的特征图 F,为了确保每个 volume 的全面表示,使用线性投影(一个 3×3×3 卷积层)将通道维度从 K = 128 增加到 d = 512。Transformer 层期望一个序列作为输入。
因此,需要将空间和深度维度折叠为一维,得到一个 d×N(N = H/8 × W/8 × D/8)特征图 f ,也可以看作是 N 个 size 为 d 的 token。
此外,为了对分割任务中重要的位置信息进行编码,使用了可学习的 position embedding,并直接将它们与特征图 f 相加,这个操作在上面的整体网络结构图中可以看到哈,我就不赘述了。
然后就输入到 Transformer Layers 了,用 Transformer 的一些规则计算(同样可以在第一篇阅读笔记中发现),在这篇阅读笔记中,我也不详细介绍了。至此,就是 TransBTS 的编码器部分。
4 Network Decoder
下面我们来看下 TransBTS 的解码器部分。MRI 脑肿瘤分割需要在原始 3D 图像空间(H×W×D)中生成分割结果,所以引入了 3D CNN 解码器来执行特征上采样和像素级分割,也就是第二部分网络结构中的右半部分。
我们注意到,在右半部分大概有两个值得关注的内容,分别为 Feature Mapping 和 Progressive Feature Upsampling。
关于 Feature Mapping,它在解码器的首部,为了适应 3D CNN 解码器的输入维度,特征映射模块将序列数据投影回标准的 4D 特征映射。此外,为了降低解码器的计算复杂度,又采用卷积块将通道维度从 d 减小到 K。那么现在特征图的恢复到 K × H/8 × W/8 × D/8。
继续往下看,还有 Progressive Feature Upsampling 操作。也就是通过一系列的上采样操作和卷积块,得到 H × W × D 的分割结果。此外,还有跳过连接,以获得更精细的分割结果和更丰富的空间细节。解码器相对简单,到这就结束了。
5 实验部分
在这一部分,首先来看下这个 3D 多模态数据集。数据集中每个样本由四种脑磁共振成像扫描模式组成,即 T1、T1ce、T2 和 FLAIR。每个图片都是 240 × 240×155,并排列在同一个空间中。
标签包含 4 类:背景(标记 0)、坏死和非增强肿瘤(标记 1)、水肿(标记 2)和增强肿瘤(标记 4)。分割精度通过用于增强肿瘤区域(ET,标记 1)、肿瘤核心区域(TC,标记 1 和 4)和整个肿瘤区域(WT,标记 1、2 和 4)的 Dice 评分和 Hausdorff 距离(95%)度量来测量。
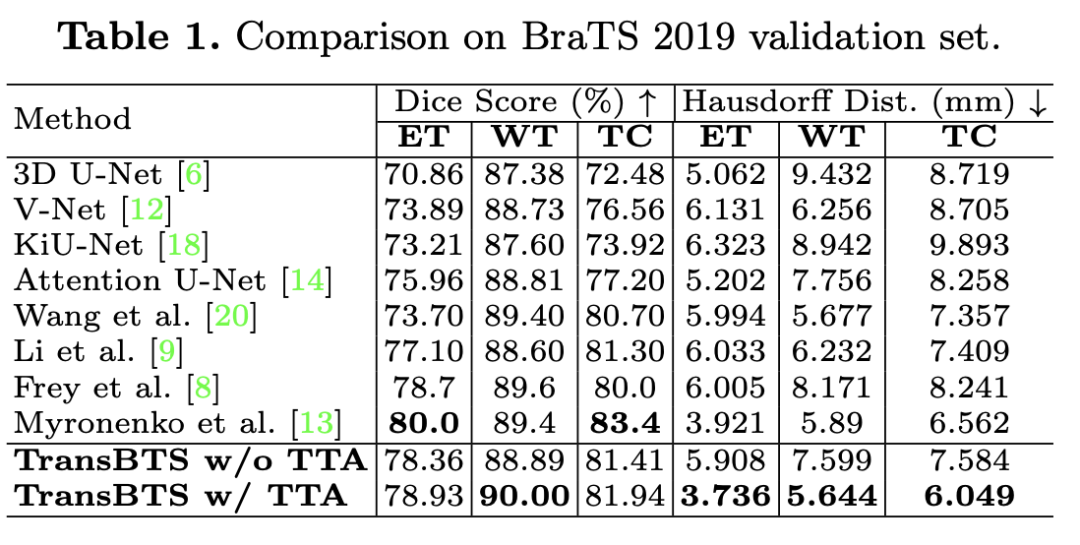
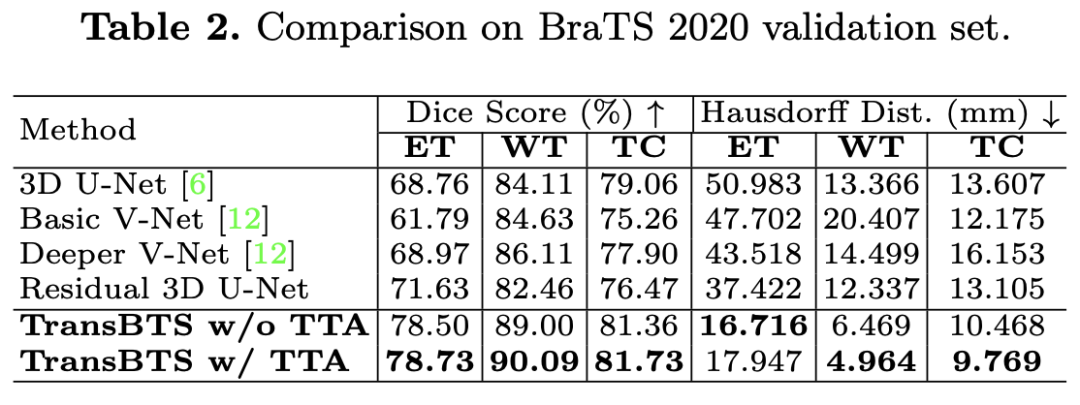
实验表明,TransBTS 在 3D MRI 扫描分割上实现了优于最先进的脑肿瘤分割方法的性能,对比实验结果如下。
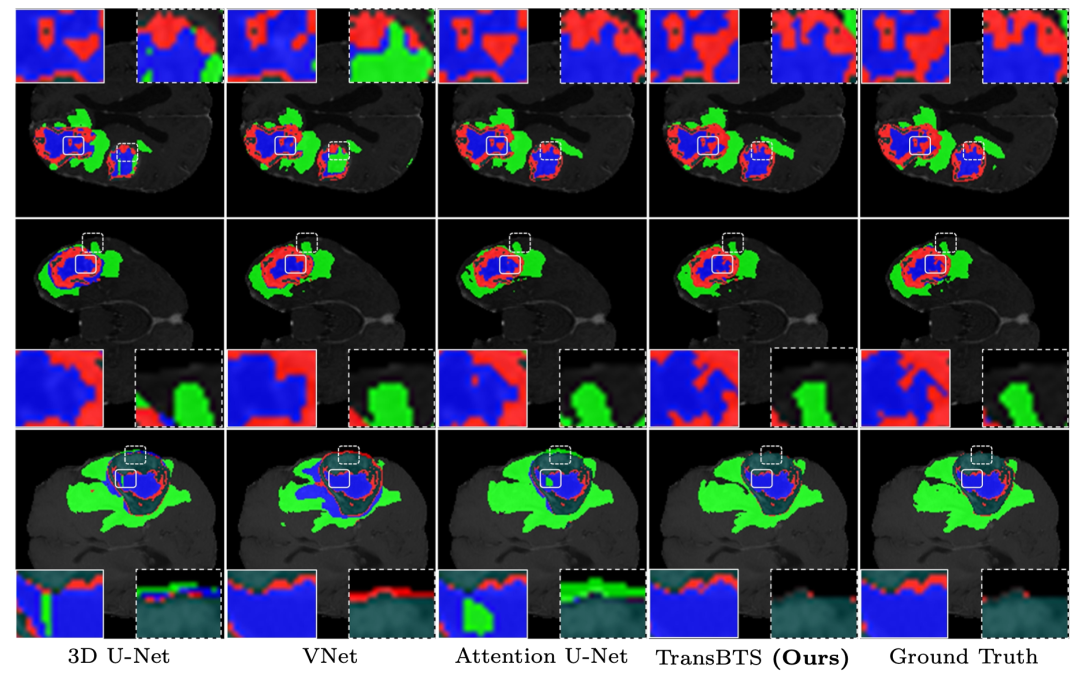
相应的,对结果进行可视化,如下图。可以明显看出,TransBTS 可以更准确地分割出脑肿瘤,通过对每个体积之间的相关性进行建模来生成更好的分割 mask。
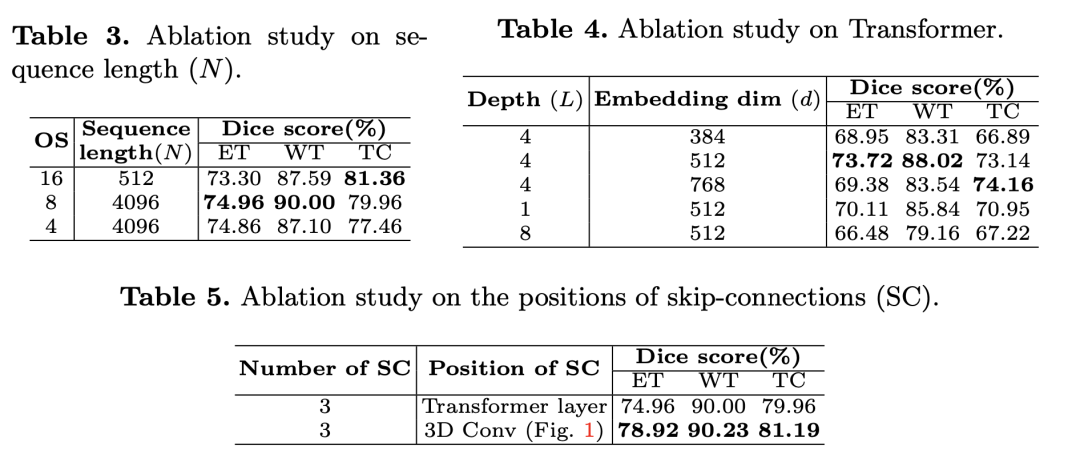
最后,我们来看下消融实验的部分。分别为序列长度消融实验、Transformer 位置的消融实验(下采样四次效果最好)和跳跃连接位置的消融研究,如下所示。
6 总结
这篇阅读笔记介绍了一种新的医学图像分割框架,该框架有效地结合了 3D CNN 和Transformer,用于磁共振成像中的多模态脑肿瘤分割。TransBTS 不仅继承了 3D CNN 建模局部上下文信息的优势,而且利用了 Transformer 学习全局语义相关性。最后,在 BraTS 2019 数据集上的实验结果验证了所提出的 TransBTS 的有效性。
这是医学图像处理系列的第三篇阅读笔记,欢迎交流和纠正!
7 参考链接
https://arxiv.org/pdf/2103.04430 https://github.com/Wenxuan-1119/TransBTS

