经典算法回顾 - 快速 get 核-PCA 的要点
引言
在机器学习中,将数据点按行摆放,所有数据点就构成一个矩阵(也可以看成表格、二维数组)。矩阵的一行对应一个数据,矩阵的一列对应一个特征,因此也称为特征矩阵。如下图所示,用矩阵
对于已经零中心化(即
上面矩阵的元素对应特征之间的协方差,这种定义形式简单也好理解。
以上是在原始特征空间中作主成分分析,但本篇的主题是核主成分分析。所谓核,则是为在更高维的特征空间作分析服务的。首先,假设我们通过以下方式将数据非线性映射到特征空间
为什么要将数据映射到更高维空间中呢,不是增加了计算量吗?主要目的是想让数据在更高维空间中线性可分。因为拿一个超平面将数据一分为二是行走江湖的基本招术。记住,机器学习的一个基本招术 - 劈柴功。

然而,在原始空间中往往不让你施展这一招。比如下面的例子,原始数据在二维空间中,两个类别分布在不同的同心圆上,此时如果想使用线性分类器来区分两类是做不到的。
那怎么办呢?可以这么来办吧,改变斧子或者数据。一个办法就是改变斧子,为某个问题专门设计某种斧子,似乎也是可行的,只是斧子一般不再是平的了。还有个办法就是改变数据,让它变得线性可分。第二个办法更通用,所以我们来看它。
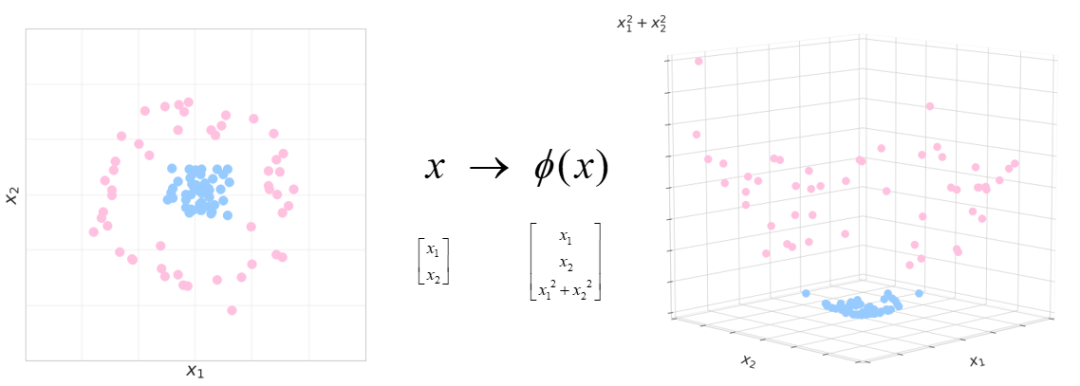
这个简单例子中,仅仅对数据增加一个维度即可,在三维空间中就可以施展劈柴大功将两类轻松分开。
我们回到核 PCA,可以证明,即使
但这里我们需要解决一个问题,那就是我们并不知道映射函数
一个是如何计算新特征的协方差矩阵;
另一个在高维空间中往新坐标轴投影得到新的坐标。
答案是,并不需要直接计算这些值,而是通过使用所谓的核技巧来实现的。下面就来看这是如何推导出来的。
核 PCA
假设将数据映射到新的特征空间中,并且
换句话说,我们只知道目标是将数据映射到更高维的空间中去,但并不给出怎么映射过去。那么问题来了,如何在高维空间中计算相应的协方差矩阵呢?类似于矩阵奇异值分解的秩-1 展开式,协方差矩阵也可以写为另一种形式,即展开为
对照式 (1) 和式 (2),会发现它们其实是同一个协方差矩阵的两种表示形式。回到核问题,此时将高维空间中的点代入,得
同样的,我们不知道映射函数
回顾一下 PCA(可以看这篇),我们需要找到满足
仔细一看,发现所有解
张成的空间内。即存在系数
从 PCA 方法那里可知,我们只需要知道上式中的系数就知道了所谓的主方向 PC。因此,问题转化为如何求解这些系数
我们可以考虑一个等价系统,即对于所有
将式 (3) 和式 (4) 代入式 (5),将出现我们真正需要计算的新特征间的内积,即
接下来,我们如下定义一个
由上面两式可得,
其中
显然,式 (8) 的所有解都满足式 (7)。
如果对解
而对于投影到主成分,我们可以根据以下公式计算测试点
到此,第二个问题,即在高维空间中的投影问题也被搞定了。
注意,式 (6) 和式 (10) 都不需要提供映射函数
,仅需内积即可。
因此,我们能够使用核函数来模拟这些内积,而无需给出实际的映射函数。常用的核函数有多项式核,
径向基核
以及 sigmoid 核
等等。可以证明,次数为
小结
不知道映射函数
仔细一想,在高维特征空间中计算协方差矩阵也好,投影到主成分也好,其实都是以内积的形式出现的。
因此,完全可以绕过对映射函数的依赖,直接用某些核函数来模拟高维空间中向量的内积来间接执行 PCA。当然,这个内积的效果好不好,也只有试了才知道。
附录
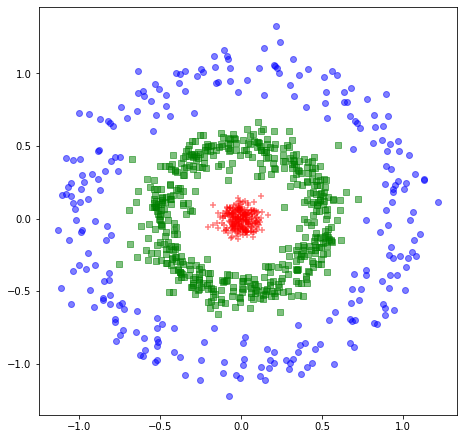
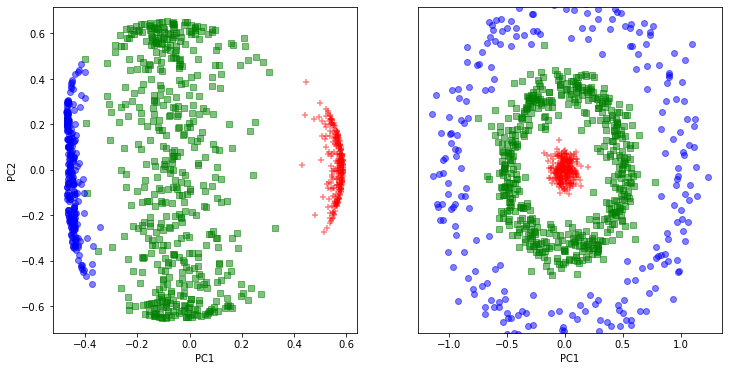
最后,来一个小例子快速实验一下,我们使用 sklearn.decomposition 实现好的版本分别采用 rbf 核和多项式核来对三个同心圆数据执行 KPCA,发现这个例子中 rbf 的效果优于多项式核。
X1, y1 = make_circles(n_samples=500, random_state=123, noise=0.1, factor=0.1)X1 = X1*0.5
X2, y2 = make_circles(n_samples=500, random_state=123, noise=0.1, factor=0.5)X = np.r_[X1, X2]y = np.r_[1-y1, 2-y2]plt.figure(figsize=(7.5,7.5))plt.scatter(X[y==0, 0], X[y==0, 1], color='red', marker='+', alpha=0.5)plt.scatter(X[y==1, 0], X[y==1, 1], color='green', marker='s', alpha=0.5)plt.scatter(X[y==2, 0], X[y==2, 1], color='blue', marker='o', alpha=0.5)plt.show()
from sklearn.decomposition import KernelPCAkpca_0 = KernelPCA(kernel='rbf',fit_inverse_transform=True,gamma=3.5)X_kpca_0 = kpca_0.fit_transform(X)kpca_1 = KernelPCA(kernel='poly',degree=1,fit_inverse_transform=True,gamma=1.0)X_kpca_1 = kpca_1.fit_transform(X)
fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(12,6))ax[0].scatter(X_kpca_0[y==0, 0], X_kpca_0[y==0, 1],color='red', marker='+', alpha=0.5)ax[0].scatter(X_kpca_0[y==1, 0], X_kpca_0[y==1, 1],color='green', marker='s', alpha=0.5)ax[0].scatter(X_kpca_0[y==2, 0], X_kpca_0[y==2, 1],color='blue', marker='o', alpha=0.5)ax[1].scatter(X_kpca_1[y==0, 0], X_kpca_1[y==0, 1],color='red', marker='+', alpha=0.5)ax[1].scatter(X_kpca_1[y==1, 0], X_kpca_1[y==1, 1],color='green', marker='s', alpha=0.5)ax[1].scatter(X_kpca_1[y==2, 0], X_kpca_1[y==2, 1],color='blue', marker='o', alpha=0.5)ax[0].set_xlabel('PC1')ax[0].set_ylabel('PC2')ax[1].set_ylim([-1, 1])ax[1].set_yticks([])ax[1].set_xlabel('PC1')plt.show()
参考资料
kpca: https://people.eecs.berkeley.edu/~wainwrig/stat241b/scholkopf_kernel.pdf
