Nvidia力作:医学图像分割网络
来源:投稿 作者:梦飞翔
编辑:学姐
引自Unetr: Transformers for 3d medical image segmentation
1.序言
本文将以Nvidia团队最近提出的一种新的医学图像分割网络作为切入点,结合所用开源数据集,为各位同学提供一份从下载数据集到搭建网络训练医学任务的完整攻略,希望可以为各位医工交叉领域的同学提供一条捷径,力争少走弯路。
2.开源数据集获取与使用
本节将以论文作者使用的BTCV[1]这一CT数据集为例,展示使用方法和注意事项。
2.1 数据集介绍
BTCV数据集来自医学全能分割比赛中,脏器分割的挑战任务。其中,输入图像包含1个通道,即CT电子密度图像,标签图像包含14个通道(13种器官加上背景点的分割)。分割任务可由下图表示,其中可以看出,原始图像的对应GT中 各个脏器已经进行了人工标记。

2.2 获取方法
在获取开源数据集之前,需要了解开源方式,常见的开源方式主要由是否需要署名、是否允许商业化、是否强制相同方式共享、是否允许演绎(修改)四个方面组成,诸如BTCV等比赛数据集一般采用署名非商业强制相同方式共享,大家可以放心下载,只要署名致谢即可。
获取方式主要分为两种,第一种便是直接去官网申请账号后,点击下载链接直接下载,有的网站会给出百度网盘链接,有的是直接http下载。
下面将简单介绍这种方法:
首先,打开全能竞赛官网,
https://www.synapse.org/#!Synapse:syn3193805/wiki/217752,可以看到如下画面,点击红框选中区域,进入数据区。

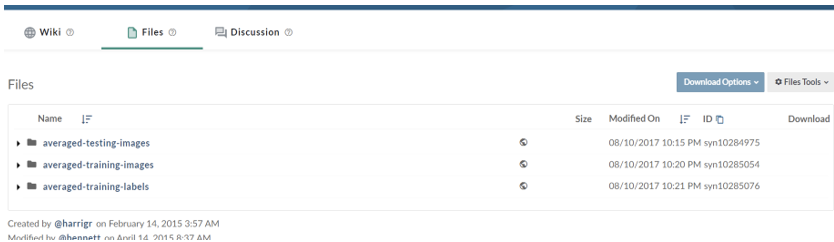
然后,如下图所示,选择下载文件进行下载,我们可以看到,数据分为训练(training)、标签(label)、测试(test)三种,这里介绍下,一般分割比赛是将训练数据集以一定比例划分为训练集和开发集,训练集训练网络,开发集简单验证泛化性能,而官方给出的测试集则是对每位选手赋分的依据,不提供GD,因此,这里我们选择下载前两者数据文件,点击后会提示注册,按照指引注册即可。
另一种获取数据的方式为,大赛开始前后比赛数据集在会集成一个API,里面会包含相关下载链接和相应下载指令,只需要自己根据官方手册编写简单脚本,即可获取到指定数据,这里我写了一个简单下载脚本,有需要可以联系我发过去。
3.网络复现与实验
在数据集到位以后,我们便可以下载作者给出的代码对他的工作进行复现,通过这种方式既可以提高码力,也可以加深对论文的理解。首先,我们从论文中找到github链接:
https://github.com/Project-MONAI/research-contributions/tree/main/UNETR
点击进去后可以看到作者将自己的工作拆分为数据预处理、模型训练、模型微调、模型验证四个部分。
第一步我们可以直接拿作者的相关代码实现,即对nii格式图像进行一些简单的图像增强和解析。
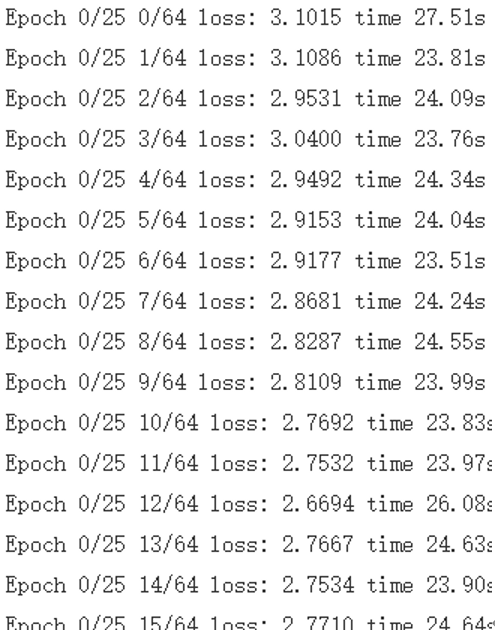
第二部分就有一些难度了,由于我们的工作站和作者工作站的环境有所差异,因此,需要先创建一个新的虚拟环境,再搭建requirements.txt里面提到的环境。其次,如下图所示,根据作者在网页中提到的一些parser参数进行赋值,最后调试一些小问题即可正常运行。

到了这里需要注意一个地方,那便是作者的日志部分可能会存在一些无法解释的问题,如果因为日志阻挡程序运行,那我们可以通过自定义简单日志替换相关代码,只要保留作者主要思想,复现便是成功的。
下面的微调是基于作者给出的检查点文件,类似于预训练模型进行小幅度训练,以期望达到更好的性能,按照作者相关代码实现即可,测试部分也同样不再赘述。
4.总结
本文主要以Unter这一篇文献为例,介绍了基于公开数据集的医学图像分割复现流程,具体代码的调试和介绍如果各位同学有兴趣了解,可以多点赞通知我呀,如果有错误请及时指出,希望能与各位小伙伴一起进步成长!
引用
[1] B Landman, Z Xu, J Igelsias, M Styner, T Langerak, and A Klein. Miccai multi-atlas labeling beyond the cranial vault–workshop and challenge. In Proc. MICCAIMulti-Atlas Labeling Beyond Cranial Vault—Workshop Challenge, 2015.
