2020 数字人体视觉挑战赛宫颈癌风险智能诊断_算法赛道亚军VNNI赛道冠军_LLLLC团队攻略分享
【GiantPandaCV导语】:本文为笔者年初参加天池举办的宫颈癌细胞检测比赛的方案整理,本次比赛同时举办算法赛道和VNNI赛道,算法赛道为线上赛,选手无法“直接”接触数据,训练和推理只能在阿里云提供的PAI平台完成。VNNI赛道为线下赛,训练数据可下载,提交docker完成线上推理。笔者的队伍分别取得了算法赛道亚军和VNNI赛道冠军。
比赛主页: “数字人体”视觉挑战赛--宫颈癌风险智能诊断赛题与数据-天池大赛-阿里云天池(https://tianchi.aliyun.com/competition/entrance/231757/information)
团队名称:LLLLC
代码开源:
算法赛道代码( https://github.com/LiChenyang-Github/tianchi_Cervical_Cancer_top4)VNNI赛道代码( https://github.com/lin-honghui/tianchi_Cervical_Cancer_VNNI_top1)
1. 赛题简介
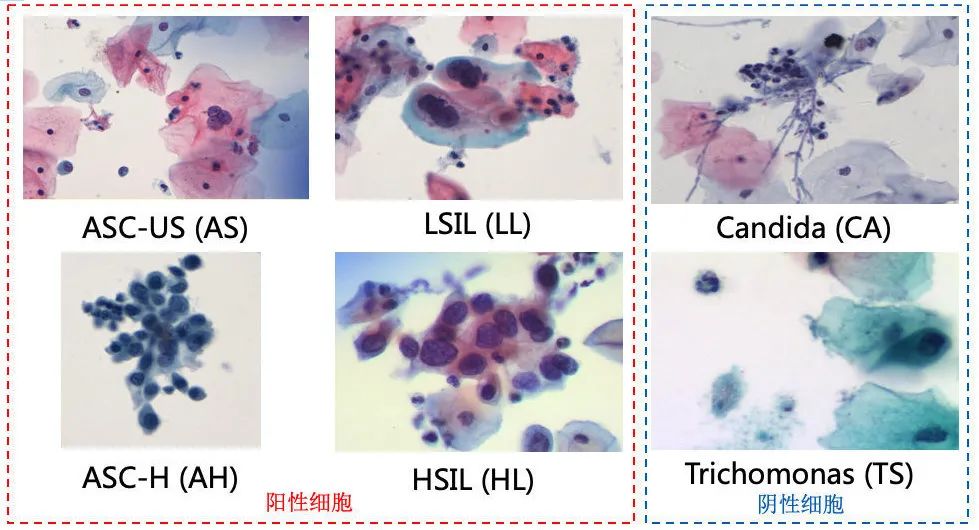
本次比赛的任务是对异常鳞状上皮细胞进行检测。初赛只需要检测出异常病变细胞,复赛时,VNNI赛道延续初赛任务,但新增QPS指标。算法赛道则需要进一步区分六类异常细胞,其中四种为阳性类别:“ASC-H” (AH,非典型鳞状细胞倾向上皮细胞内高度)、“ASC-US” (AS,非典型鳞状细胞不能明确意义)、“HSIL” (HL,上皮内高度病变)、“LSIL” (LL,上皮内低度病变),两种为阴性类别:“Candida” (CA, 念珠菌)、“Trichomonas” (TS, 滴虫)。阳性类别为鳞状上皮细胞本身发生病变,而阴性类型是细胞受到微生物 (念珠菌、滴虫)感染。
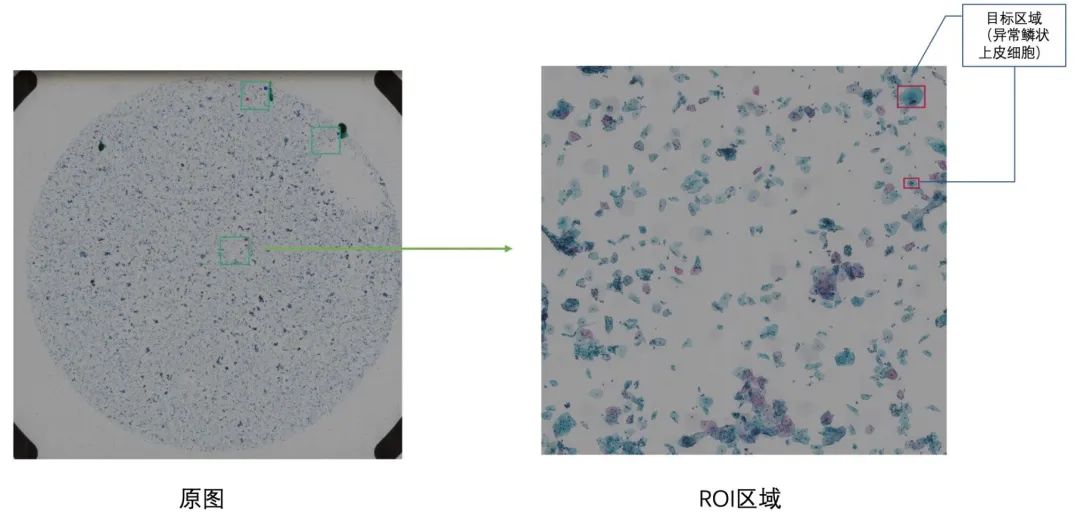
图片数量方面,初赛提供宫颈癌细胞学图片800张,其中阳性图片500张(注意区分于上面的阳性类别),阴性图片300张。阳性图片并未完全标注 (分辨率为上万的数量级,全部标注较耗时),而是提供多个ROI区域(1212 ROI),在ROI区域里面标注异常鳞状上皮细胞位置,阴性图片不包含异常鳞状上皮细胞,无标注。
复赛共提供1690张宫颈癌细胞学图片,其中1440张为阳性图片,250张为阴性图片,阴性图片不含有6类异常细胞,因此也没有标注信息。1440张阳性图片标注了3670个ROI区域 (分辨率为几千的数量级),并提供ROI区域的标注信息 (ROI以为的区域可能也存在异常细胞,但未进行标注)。
2. 算法赛道
2.1 数据分析
我们首先对数据进行了病理学和数据统计两方面的分析,通过这两方面的分析,得到一些可以利用的先验以及任务的主要挑战。
2.1.1 病理学先验
结合病理学知识的一些发现:
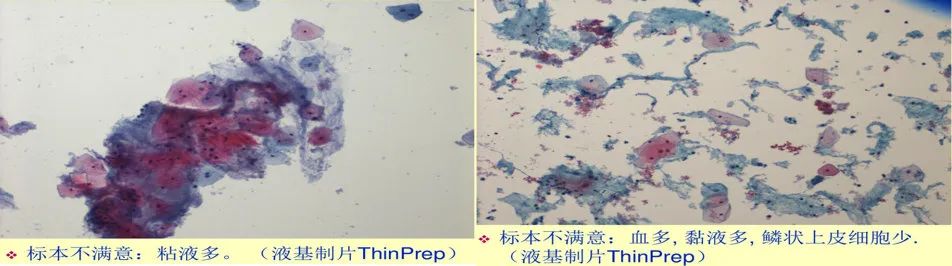
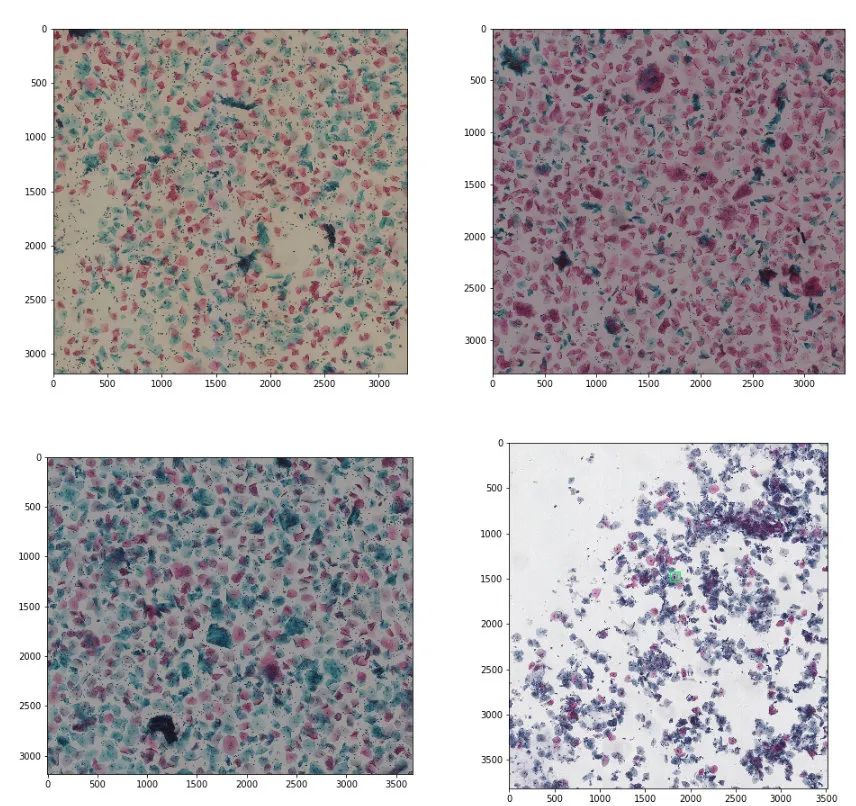
细胞形态方面。四类阳性细胞较为相似,它们是出于不同时期的病变细胞,主要的区别在于核质比的变化;两类阴性细胞具有比较明显的特点,CA主要是受念珠菌感染的细胞,呈矛状,体积较大,TS是受滴虫感染的细胞,滴虫主要附着于细胞外部,体积较小; 细胞位置随机均匀分布。如图2-1是液基细胞学检查的制片过程,其中包括了振荡和离心过程,所以可以粗略认为细胞在玻片上是均匀随机分布的,这对后面的数据增强策略具有指导作用; 不共存先验。念珠菌和滴虫适合生存的pH环境是不同的,也就是在大部分情况下,念珠菌和滴虫只能单独存在,这对后面的模型设计以及后处理具有指导作用; 背景因素干扰。如图2-2是一些不满意的标本的示意图,可以看出,某些玻片可能存在粘液,血等背景的干扰,如何提升背景的多样性也是值得考虑的问题; 细胞成像颜色多样性。如图2-3所以,受染色剂种类、配比、染色时间长短及不同数字扫描仪等因素影响,细胞成像颜色具有多样性,提升模型对染色泛化能力也是本次比赛需要考虑的问题;

2.1.2 数据统计
通过数据统计的一些发现:
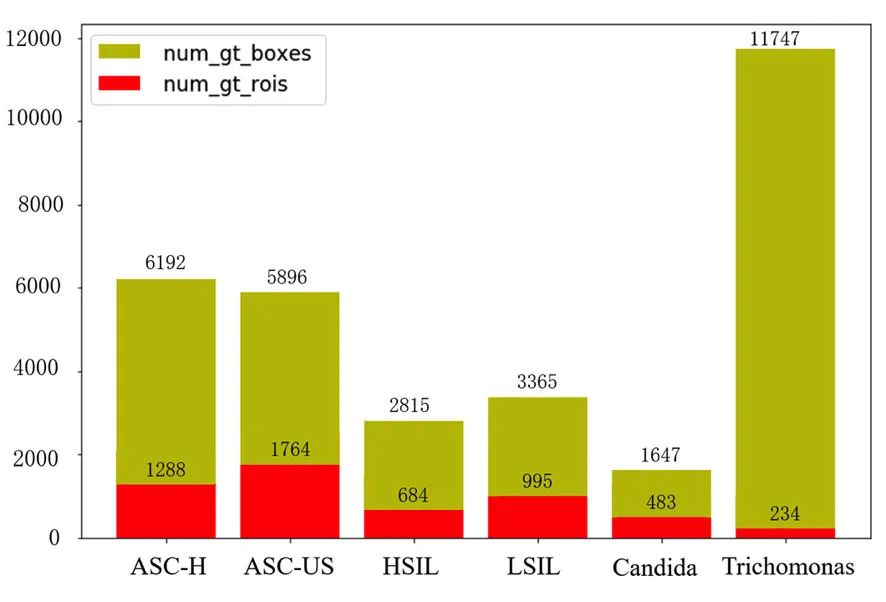
类别数量不平衡。如图2-4所示,是每一类细胞的ROI数量以及标注框 (gt_boxes)数量示意图,可见存在类别不平衡问题,且这种不平衡有以下的特点:TS类以最少的ROI (234个ROI),占据了最多的标注框 (11747个标注框),即ROI最少的类别标注框反而最多。这一点在后面的训练策略中需要进行考虑;
标注框尺度变化大。如图2-5所示,可以看出,四种阳性细胞的尺寸只要在几十到两百之间;CA的尺寸较大,存在几百甚至上千的尺寸;TS的尺寸较小,大部分为五十以下;
类别不共存先验。如图2-6所示,我们使用邻接矩阵的形式来统计六个类别的两两共存情况,可以发现,CA和TS基本上是单独出现,四类阳性细胞之间互为共存,这也和前面2.1.1的病理学分析是吻合的;


2.2 方案介绍
我们的整体方案可以分为数据、模型、预测与后处理三大部分进行介绍。
2.2.1 数据
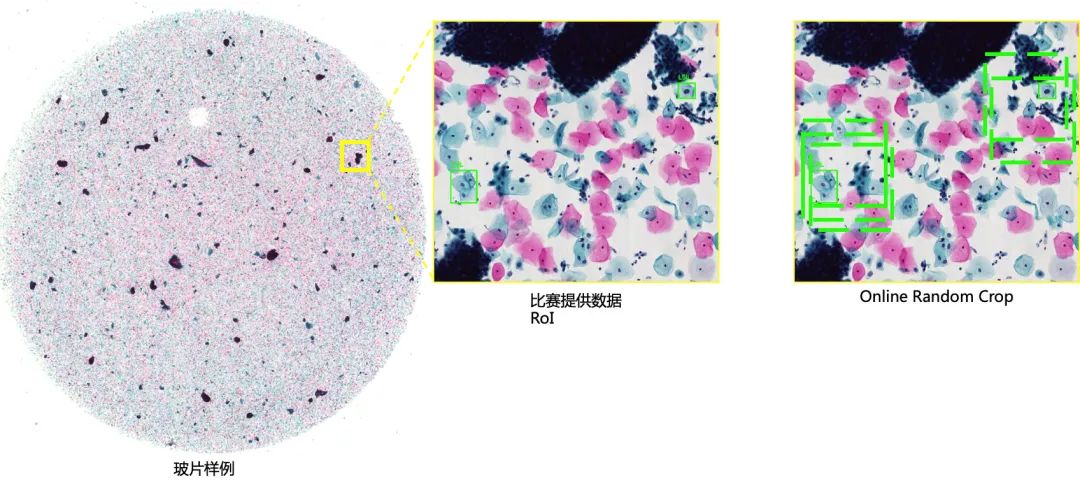
数据采样:在训练阶段,我们采用Online Random Crop的采样方式:在ROI区域中,以病变细胞为中心产生随机偏移,裁剪固定大小区域作为训练图片,如图2-7所示。所以一个epoch就是遍历所有的3670个ROI。
数据增强:除常规的数据增强方式如随机翻转、随机旋转、随机颜色扰动、随机标注框抖动外,我们针对本次比赛背景干扰和细胞染色问题,使用了下面的数据增强方法:
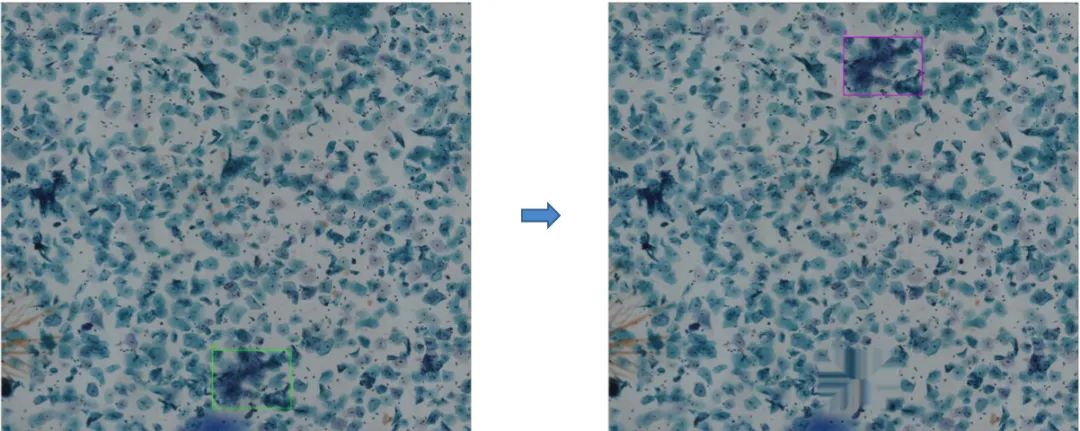
随机标注框移动(阳性样本背景利用):OnlineRandomCrop是以病变细胞为中心的背景随机采样,阳性图片中其它区域背景可能未被充分利用,我们通过随机移动标注框方式来增加背景多样性,如图2-8所示,我们将标注框进行随机移动,并用inpainting操作对原来的位置进行填充。通过这种方式来改变标注框周围的背景;
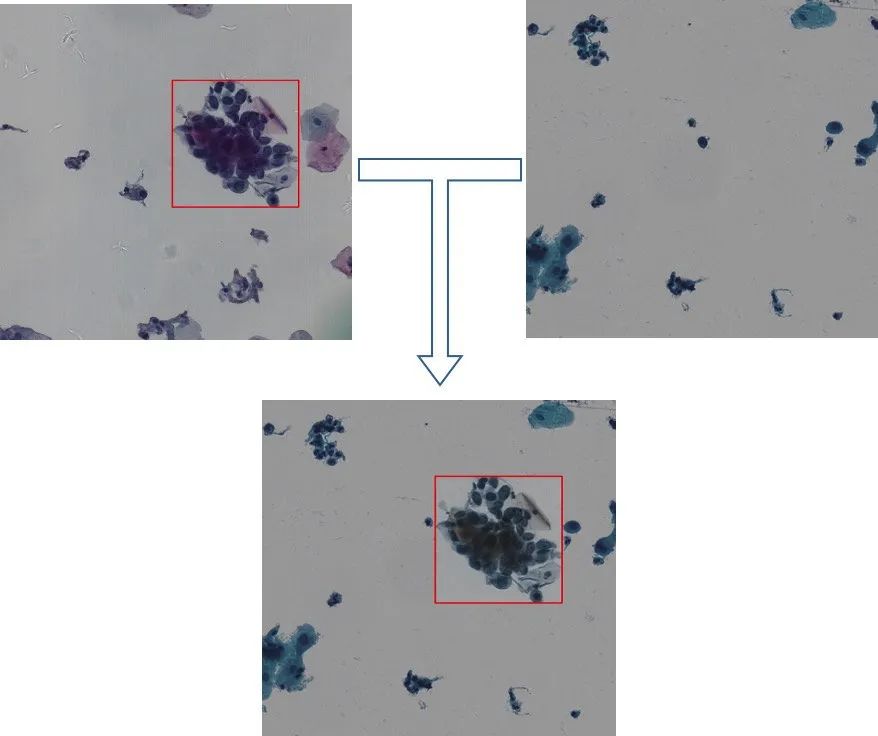
CopyPaste + StainNorm(阴性样本背景利用+染色剂归一化):如上所述,本次比赛除了提供1440张阳性图片以外,还提供了250张阴性图片,阴性图片的利用对模型假阳抑制有较大帮助。具体地,如图2-9所示,将阳性图片中的异常细胞复制(copy),再以一定概率随机贴到纯阴性背景数据中进行样本扩充(paste),利用阴性图片来增加背景多样性,从而改变标注框周围的背景,增加分类器negative样本的多样性。需要注意的是,在利用阴性图片的时候需要注意防止截取到图片的四个角的纯色背景部分,如图1-2所示,玻片中的细胞是位于一个圆形的区域内,如果截取到四个角的话会产生纯色的背景,影响分类器训练;
此外,阳性样本背景利用中随机移动标注框是在同个样本ROI中进行,不存在染色差异问题,但将copy的阳性细胞目标框直接贴合到新的背景中,可能因为颜色差异大而十分“违和”。我们采取的方式是以通过染色剂归一化算法[1],将阴性背景作为target image,将copy的阳性细胞目标框都根据背景进行染色归一化,降低颜色差异(这里背景是动态随机选取,target image一直在变。引入阴性样本一定程度也引入了样本颜色多样性)。

2.2.3 模型
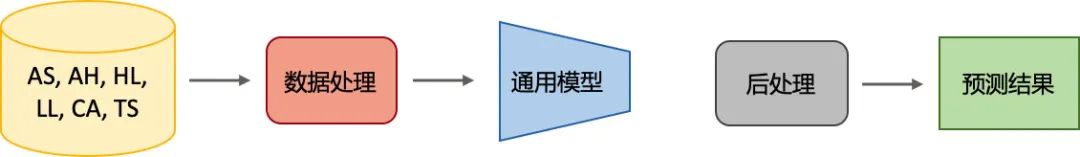
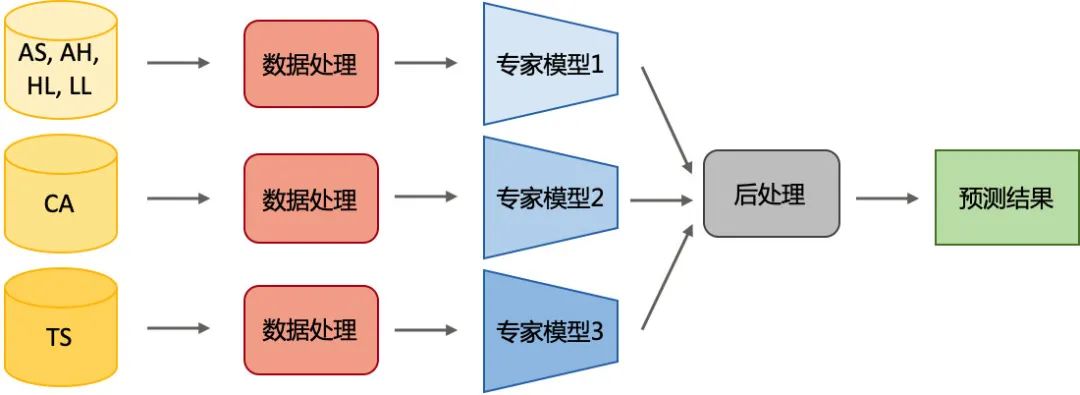
我们使用通用模型和专家模型结合的形式,通用模型(图2-11)对6种类型的细胞进行检测,专家模型(图2-12)针对特定的类别进行检测。
模型的具体组成发面,我们使用的是一些常见的经典结构:
检测框架:Faster-RCNN [2]; 基础网络:ResNet-50 [3]; 其它模块:FPN[4] / BiFPN[5], DCN[6], Cascade[7];
由于上述都是一些比较常见的结构,这里不进行详细介绍,下面主要介绍针对本次任务数据的特点,分享在训练过程中一些细节。
在训练通用模型的过程中:
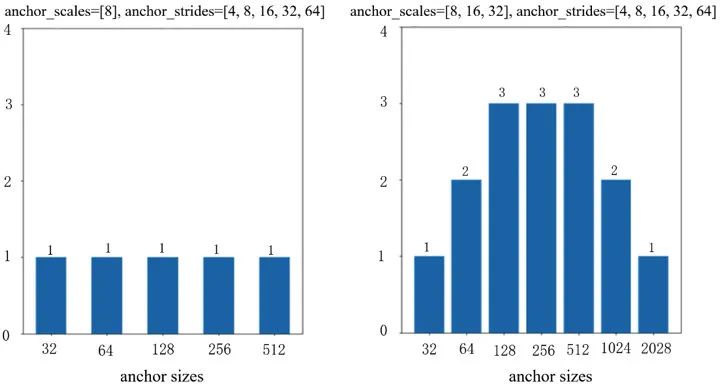
batchsize的大小会对模型性能产生影响。前面提到,此次数据存在不平衡问题,且TS以最少的ROI占据了最多的标注框,即TS在图片中密集出现。有234个ROI包含TS,而总共有3670个ROI,我们可以计算不同的batchsize下TS在一个batch中出现的概率,如图2-13所示,可见随着batchsize的增大,TS在某个batch中出现的概率增加。另外,当TS在某个batch中出现的时候,其梯度很可能会成为主导,最终导致模型对TS产生bias。所以我们最终采用较小的bathsize (1或2)进行实验; anchor scale会对模型的性能产生影响。因为6个类别的标注框尺度有较大的差异,一开始我们希望通过增加anchor scale来提升anchor对CA的覆盖程度,但是发现这样并不是很work。如图2-14所示,展示的是scales取[8]和[8, 16, 32]的时候anchor的尺寸和数量情况,可以看到,增加scales之后,anchor的尺寸变大了,能够覆盖到更大的CA (这是我们的本意),但是这会导致中等大小的anchor的占比升高了,而中等大小的anchor的尺寸主要是128-512,这一范围和阳性细胞的大小正好match,这会导致模型对四类阳性细胞产生bias,在我们的实验中,最终采用的是单一的scales; 在训练专家模型的过程中:考虑如何抑制假阳性预测。阳性细胞、CA、TS的ROI数量分别为2953、483、234,可见CA和TS中总的ROI (3670)的比例较小。训练专家模型的时候,以训练CA的专家模型为例,最常规的做法是仅使用483个含有CA的ROI进行训练,我们发现这样训练出来的模型在测试集上会有很多的假阳预测 (因为模型“见过”的背景较少,并没有见过四类阳性细胞以及TS,很容易会产生假阳预测)。因此,我们在实验中的做法是,训练某一种类型的专家模型的时候,将其它类型细胞的ROI数据当成背景数据使用,通过上述的随机背景替换数据增强方式进行利用。这样,模型的分类器就能“见到”其它类型的细胞,提升分类器的性能。

2.2.3 预测与后处理
预测阶段,我们采用交叠滑窗的形式 (步长为patch大小的一半),采用交叠滑窗的方式主要是为了保证patch边缘的检测质量,防止检测结果被patch“截断”。
在后处理方面,我们主要进行重叠抑制和异类不共存抑制两方面的处理:
重叠抑制: 模型内部抑制:对预测阶段的滑窗重叠部分的预测框进行融合,如图2-15所示。采用Box Voting[8],IoU阈值为0.2; 模型之间抑制:对不同模型的预测框进行融合,采用WBF (Weighted Box Fusion)[8]进行融合,IoU阈值为0.3; 类内抑制:对同种类别的重叠预测框进行融合 类间抑制:对不同类别的重叠预测框,抑制分数低的类别。采用跨类别的NMS,IoU阈值取0.3 (这种方法在比赛的时候收效并不明显,甚至有所下降,主要还是因为模型本身不是很强,以及评价指标mAP的计算方式); 异类不共存抑制: 利用CA过滤其它类型的框:当ROI中超过三个CA预测框的置信度大于0.85的时候,抑制该ROI上其它类别的预测框 (我们尝试过依据其它5类细胞的预测置信度进行抑制,但是没有收效,主要是因为我们的CA本身区分度比较高,且我们的CA模型比较强一些);


2.3 方案总结
在本次比赛中,我们首先根据病理学和数据统计得到一些先验和挑战:
先验:不共存,随机分布; 挑战:背景干扰,类别不平衡,标注框尺度变化;
我们提出的方法可以总结如下:
数据层面:随机背景替换,随机标注框移动(随机分布;背景干扰) 模型层面:通用模型和专家模型结合(不共存;类别不平衡) 训练层面:减小batchsize,均衡anchor scale(类别不平衡,标注框尺度变化) 后处理策略:不共存抑制,重叠抑制(不共存)
3. VNNI赛道
3.1 赛题概述
3.1.1 任务
对异常鳞状上皮细胞进行定位
VNNI赛道:在支持Intel VNNI的框架上进行量化推理
3.1.2 指标
性能scroe :mAP 时间score :QPS = slide数量 / inference总时间(总时间包括数据预处理、模型推理、后处理时间等) 最终score = 性能score + 时间score(两个指标分别计算,取第一名作为Gold standard,score=1.0, 其余的成绩与第一名的差距的百分比作为score的累加值,依次计算。如第一名0.3张/s,第二名0.1张/s,则第一名score=1.0,第二名score=1.667)。
3.1.3 数据
训练数据(初赛数据):宫颈癌细胞学图片800张,其中阳性数据500张(包含1212个ROI),阴性数据300张; 测试数据(复赛VNNI赛道新增):200张ROI区域图片(分辨率在几千到上万)
3.2 方案介绍
数据预处理在第2章算法赛道中已有详细介绍,VNNI赛道数据预处理方式与算法赛道基本一致,这里不再赘述。
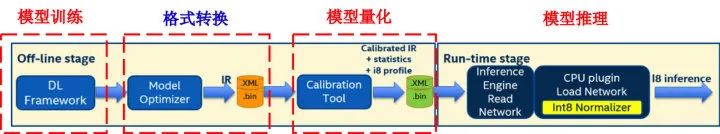
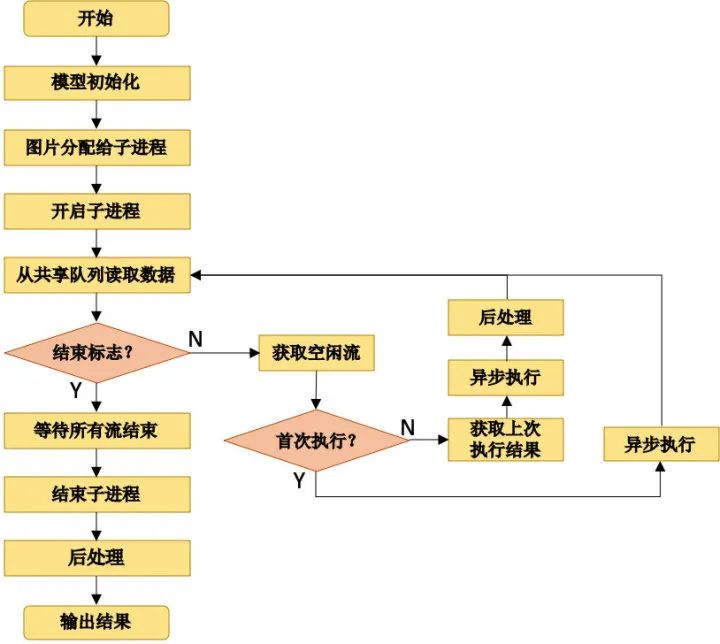
模型选择上,我们采用基于RetinaNet[9]的目标检测算法,在 pytorch 框架上进行模型训练,在 Openvino 上完成量化推理,流程图如下,包括以下四个步骤:模型训练、格式转换、模型量化、模型推理。
3.2.1 模型训练
为了提升推理速度,我们对 RetinaNet 各个模块重新设计。
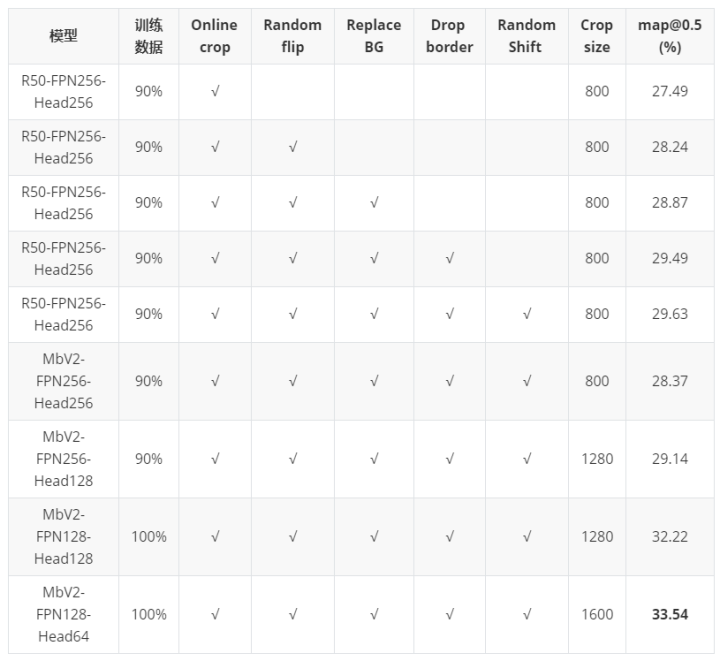
Backbone : 采用轻量化的 (ImageNet pretained) MobileNetV2 [10] ; 实验中,MobileNetV2性能与ResNet-50相当,ResNet-34、ResNet-18等精度下降; 使用本次比赛数据,预训练分类网络作为backbone初始化效果不如ImageNet; 减少 FPN 以及 Head 通道数: FPN-256 ---> FPN-128 Head-256 ---> Head-64

3.2.2 模型量化
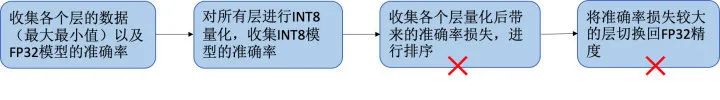
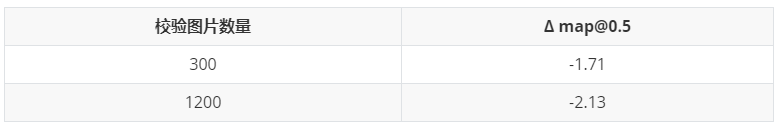
量化过程中,量化层选择及校验数据对量化结果会产生一定影响。**OpenVINO Calibration Tool **支持自动进行量化层选择,将准确率损失较大的层切换回fp32,但在实验中我们发现对我们模型并不是很work;实验中,我们不采用OpenVINO自动量化层选择,而是基于经验,FPN部分参数对量化比较敏感,对MbV2-FPN1287-Head64,FPN部分卷积层不量化,其余卷基层全部量化;关于校验数据,在我们的实验中增加校验数据并没有带来校验性能的提升,我们最终以300张训练图片作为校验数据;

3.2.3 模型推理
推理流程包括:图片读取、预处理、前向执行、后处理等步骤。
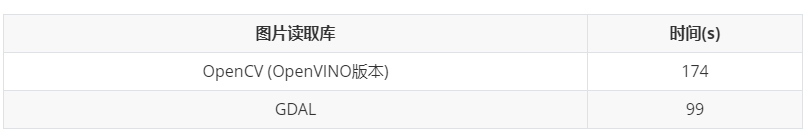
图片读取:实验中,由于比赛提供数据分辨率普遍较大,IO读取占据了相当大的一部分时间消耗,采取了两个优化策略:
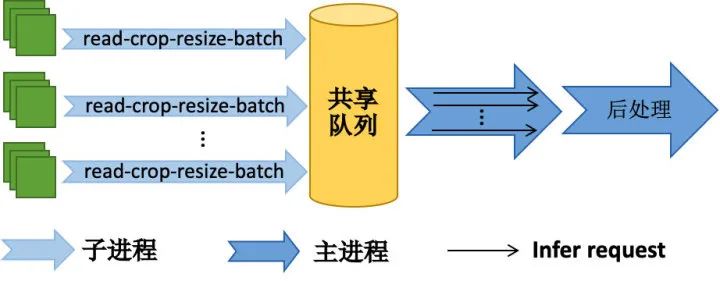
采用读取速度更快的GDAL库[12],性能优于opencv; 多个子进程同时读取,存放到共享队列中;
预处理:主要包括以下步骤:
采用无交叠滑窗策略; 丢弃边界像素,加速推理; 将裁减 1600x1600 区域缩放成 800x800;
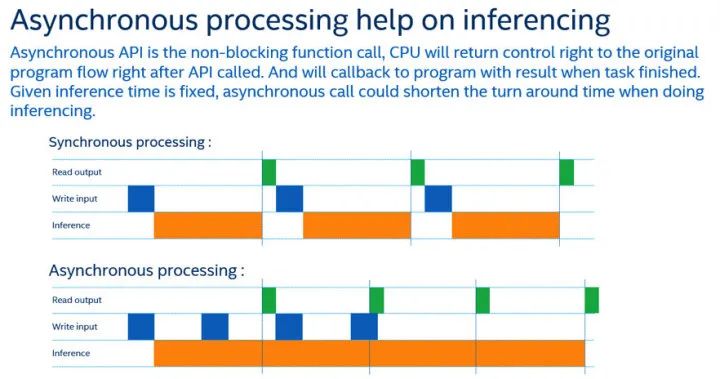
前向执行:采用 OpenVINO 的异步模式[11] :
发起执行请求后,控制权交还给主程序,分摊数据读取和后处理的时间; 执行完成后,通过回调函数通知主程序; ―可以发起多个infer request同时执行;

3.2.4 代码框架 & 流程图
子进程负责图片的读取、裁剪、缩放、拼batch等数据处理相关的操作,处理完的数据存放到共享队列中; 主进程从共享队列读取数据,负责模型推理、后处理操作;
3.3 实验结果
3.3.1 性能分析
数据增强带来了稳定的性能提升; 同个setting下 ResNet50 性能略高于 MobileNetV2,但本次比赛时间性能提升带来的分数收益更大,最终方案还是选择了MobileNet; 增大输入crop size也可以提升模型性能,可能是减少滑窗过程细胞被“截断”概率;
3.3.2 时间性能分析
对 Batch size 、 infer request 、 subProcess (io) 、 Drop border 等参数进行调优; 由网络结构从MbV2-FPN256-Head128调整为计算量更小的MbV2-FPN128-Head64,但推理时间不变(均为120s)可知,推理时间瓶颈不在模型,而是 读取,更换更快的gdal读取库之后,时间从120s --> 35s。

3.4 方案总结
本次VNNI比赛中,我们提出方法总结如下:
轻量化网络结构设计; 针对医疗数据场景样本较少的数据增强方法; 采用异步模式进行推理; 多进程、GDAL库等加速IO;
展望:
IO与推理的时间大约各占一半,更优化的IO库,更小的模型都能进一步提升性能; 推理时采用更大的裁剪区域或者整图,预计能进一步提升速度和精度; 采用量化损失更小的 backbone(Intel工程师建议,目前 OpenVINO 针对 ResNet 优化较好,量化损失较低);
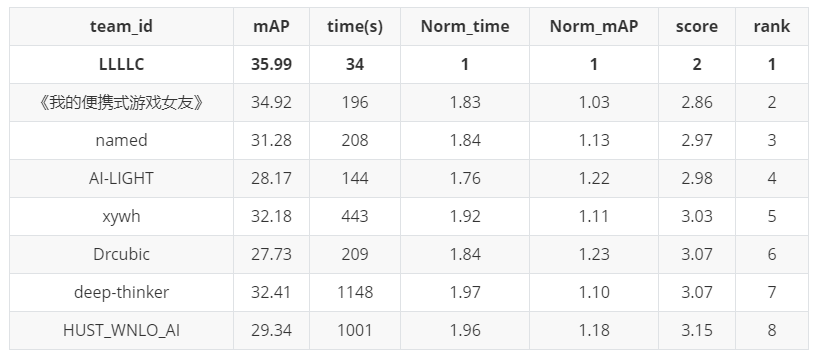
结果:
4. Reference
[1] https://github.com/wanghao14/Stain_Normalization
[2] Ren, Shaoqing, et al. “Faster r-cnn: Towards real-time object detection with region proposal networks.” Advances in neural information processing systems. 2015.
[3] He, Kaiming, et al. "Deep residual learning for image recognition." Proceedings of the IEEE conference on computer vision and pattern recognition. 2016.
[4] Lin, Tsung-Yi, et al. “Feature pyramid networks for object detection.” Proceedings of the IEEE conference on computer vision and pattern recognition. 2017.
[5] Tan, Mingxing, Ruoming Pang, and Quoc V. Le. "Efficientdet: Scalable and efficient object detection." Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. 2020.
[6] Dai, Jifeng, et al. "Deformable convolutional networks." Proceedings of the IEEE international conference on computer vision. 2017.
[7] Cai, Zhaowei, and Nuno Vasconcelos. "Cascade r-cnn: Delving into high quality object detection." Proceedings of the IEEE conference on computer vision and pattern recognition. 2018.
[8] Solovyev, Roman, and Weimin Wang. “Weighted Boxes Fusion: ensembling boxes for object detection models.” arXiv preprint arXiv:1910.13302 (2019).
[9] Lin, Tsung-Yi, et al. "Focal loss for dense object detection." Proceedings of the IEEE international conference on computer vision. 2017.
[10] Sandler, Mark, et al. "Mobilenetv2: Inverted residuals and linear bottlenecks." Proceedings of the IEEE conference on computer vision and pattern recognition. 2018.
[11] https://docs.openvinotoolkit.org/2019_R3.1/_inference_engine_tools_benchmark_tool_README.html
[12] https://gdal.org/
欢迎关注GiantPandaCV, 在这里你将看到独家的深度学习分享,坚持原创,每天分享我们学习到的新鲜知识。( • ̀ω•́ )✧
有对文章相关的问题,或者想要加入交流群,欢迎添加BBuf微信:
为了方便读者获取资料以及我们公众号的作者发布一些Github工程的更新,我们成立了一个QQ群,二维码如下,感兴趣可以加入。
