关于二代测序中duplication产生和占比问题的探讨
NGS系列文章 - 高通量测序原理


duplication是怎样产生的
在讨论上述问题之前,我们首先来看一下,duplication都在哪些环节中产生的呢?
要理解这个问题,必须首先要知道从样本准备到测序这一系列过程中都涉及了怎样的步骤。
以基因组测序为例简单概述,主要过程包含:
样本制备及核酸提取;
核酸序列的随机打断和特定长度片段的回收;
测序文库构建;
上机测序。
(1)核酸提取和序列的随机打断
Illumina测序要求短读长,因此在提取核酸后,首选需将它们打断为小片段,例如超声打断,并电泳回收一定长度(如350bp左右)的小片段。
考虑到偌大的背景集,经随机打断后,出现两条一摸一样(长度、碱基序列完全一致)小片段的概率应该就是0了。
因此,这一步理论上不会出现duplication。
(2)文库构建和PCR扩增
之后使用这些合适的短片段,构建测序文库。
建库过程
简略来说,就是在DNA片段两端添加测序接头(adapters)序列。(详细建库过程,可自行参阅各实际使用的建库试剂盒)
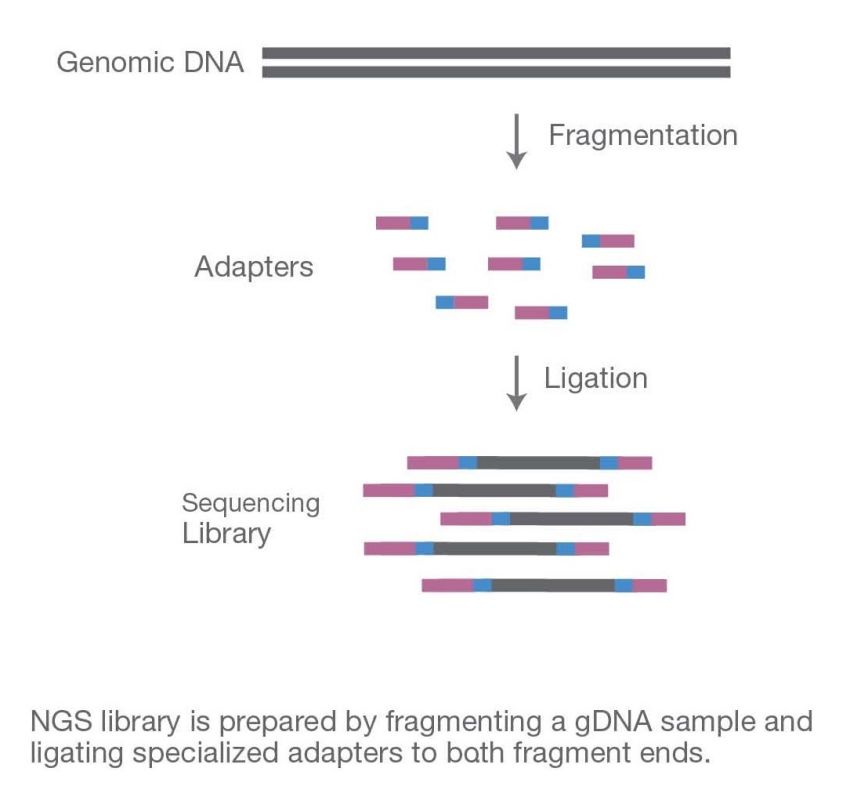
adapters添加后,会再进行数轮的PCR过程,以adapters上的序列为模板起始位置,进行PCR。
PCR的主要目的就是在含adapters DNA片段的两侧继续添加P5、P7接头,含P5、P7接头的DNA片段能够和测序仪Flow cell中的衔接子结合,使目标测序序列能够被测序仪捕获,以实现测序的目的(详见下文)。
另一方面,adapters和DNA片段的初始连接效率比较低下,在反应结束后,整个反应体系中含有adapters的DNA片段比例较低。如果直接用于上机测序,可能影响文库中DNA片段被测序仪捕获的效率。PCR扩增过程也可以使含adapters的DNA片段比例得到充分富集。
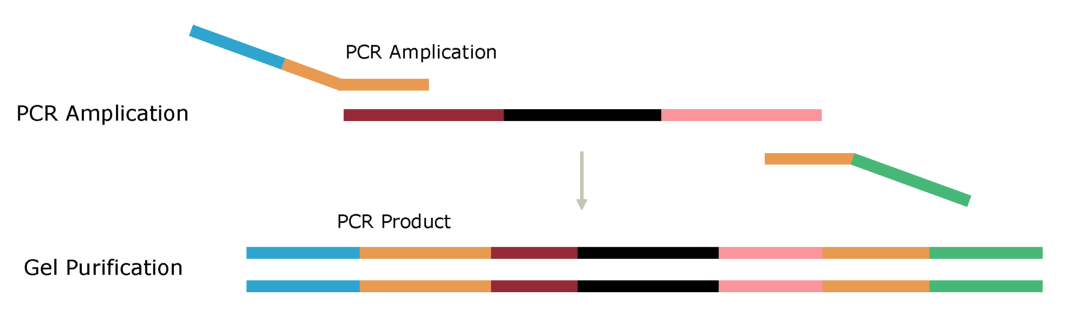
与建库有关的duplication产生
很轻易地就理解了,这个PCR扩增就是duplication的主要产生原因了。伴随循环次数的增加,会产生更多的多拷贝;且由于PCR扩增的偏好性问题,也并非所有的含adapters的DNA片段都是均匀扩增的,不过好在PCR循环次数一般不多,不均一性影响较微。
但是PCR少说也有数轮,含adapters的DNA片段被扩增了数十、数百次也有了,带来了duplication的丰富来源;多则更是产生数千的拷贝数。
(3)Illumina测序
DNA簇的形成
Illumina测序仪中实际进行的测序反应位于流动池(flow cell)中。

flow cell是吸附移动DNA片段的通道,它也是测序反应的核心容器,所有测序过程都在这里进行。每个flow cell有8个泳道,每个泳道的表面都附着有非常多的衔接子,可以与文库中DNA片段末端添加的adapters两侧的P5、P7接头互补以吸附目标DNA。测序文库中的DNA片段穿过flow cell时,它们将随机附着在flow cell表面的泳道上,并在这里以flow cell表面上的衔接子作为模板进行PCR扩增(即桥式PCR,可自行了解)。理论上,各泳道之间互不影响。在连续扩增循环后,每个DNA片段最终将在其各自位置成束聚集(cluster),每个cluster都代表了初始结合的单个DNA分子的多拷贝。

注意该过程的目的仅为放大碱基的信号强度,以满足测序的信号要求。cluster完成后,它们下一步用于测序。
测序反应
该测序方法基于SBS (sequencingbysynthesis, SBS)。在反应体系中加入DNA聚合酶、连接引物和4个带有碱基特异性荧光标记的dNTP。这些dNTP的3'-OH受化学保护,可确保在测序过程中一次只能延伸一个碱基。所有未使用的游离dNTP和DNA聚合酶在合成反应完成后洗脱。
然后加入荧光激发所需的缓冲溶液,通过激光激发荧光信号,并通过光学设备记录荧光信号。
最后,通过计算机分析将光信号转换为测序碱基。记录荧光信号后,添加化学试剂猝灭荧光信号并去除dNTP 3'-OH保护基团,从而可以进行下一轮测序反应。
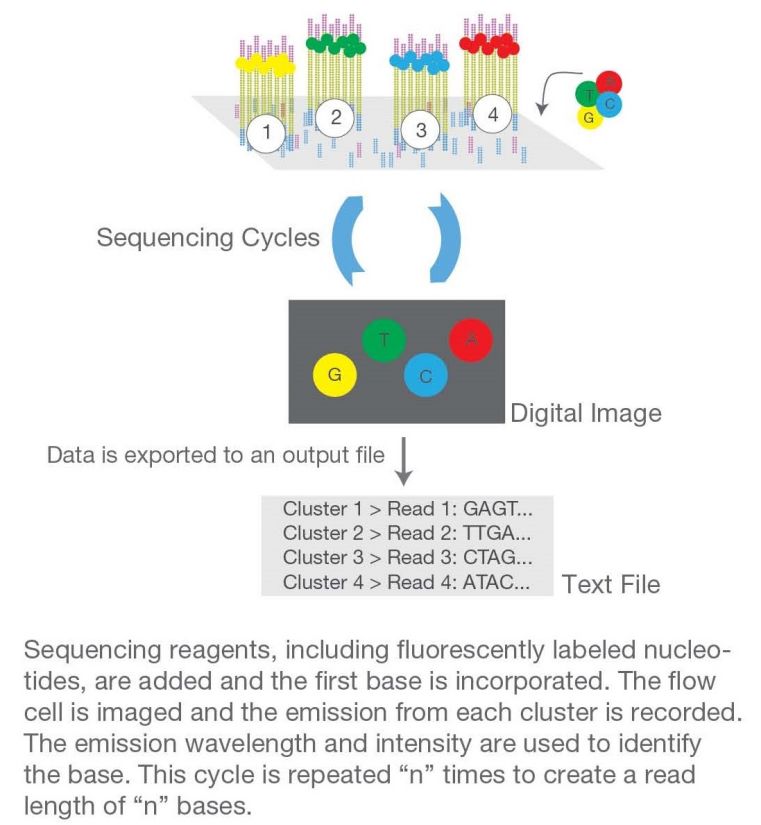
上一步进行桥式PCR将单分子DNA扩增成cluster,就可以保证这里测序时获得充分的荧光信号,并降低单个DNA分子扩增错误的影响。记录整个flow cell中所有结合位置的cluster的荧光信号,即可获得每一条DNA分子的序列碱基组成信息。cluster的位置被记录在fastq文件@seq-id这一行中。
对于每一组cluster,一次测序一个碱基,全部反应过程中即可逐步实现对整条(或部分长度,取决于测序最大读长)DNA片段的测序。

与测序有关的duplication产生
测序过程中产生duplication的原因主要有3点。
(1)在DNA片段与flow cell表面的吸附过程中,会出现相同DNA分子(由建库过程中的PCR产生)吸附在flow cell的不同位置,后续形成各自的cluster并测序后,得到了相同的reads序列。
(2)桥式PCR过程中,也可能会出现极少数已发生拷贝的DNA分子和flow cell的结合不稳定而脱离,之后吸附到另一结合区域并扩增成cluster,测序后得到相同的reads序列。
(3)荧光信号捕获时的问题,部分荧光不稳定导致识别碱基错误,有可能使本不相同(但差异很小)的两条序列的测序reads相似,后续它们比对至基因组的同一区域。
关于duplication比例的问题
好了,接下来回到最初的话题,关于测序数据中duplication的占比问题。
可以看到duplication产生的最多的一步,就是在建库过程中的PCR那一步环节,导致了几乎所有目标DNA片段都产生了多拷贝。就以目前常见的Illumina建库试剂盒来说,要求10-15轮PCR之间,以12轮PCR为例,平均每个目标DNA片段都获得212=4096个拷贝,多么大的一个数字。
然而最终的测序数据中,duplication的平均占比仅10-20%,并没有想象中的那么糟糕,想必很多同学都非常困惑这一点,这是为什么呢?
关键就在DNA片段与flow cell表面吸附的这一步。
因为不是文库中所有DNA片段都能吸附到flow cell表面,flow cell捕获的DNA片段仅为文库中带adapters的DNA片段的一个很小的子集。如上所述,该过程是随机捕获的,因此相当于从文库中所有的DNA片段中进行一次抽样,相较于背景集(文库)中的DNA片段数量,两者可以说不在一个数量级上。因此,同一目标DNA片段的多拷贝序列被捕获次数≥2次的情况,其实是很少的,所以结果中duplication的比例并不糟糕。
伴随的疑问,flow cell仅捕获文库中的一小部分DNA子集用于测序,那么这个子集能覆盖目标物种基因组码?这点倒是大可不必担心,这些被flow cell吸附的DNA片段数,也数以千万计了,以人基因组测序为例,最终获得的数据量覆盖深度几十×的覆盖度也有了;如果是人转录组或外显子测序,则覆盖度更高;换成其它小型基因组,更不用说。
至于其它可能影响duplication的原因,相较于这两种情况可以忽略不计。因此在二代测序中,duplication通常也被称为PCR duplication。
综上可知,duplication与建库过程中的PCR循环次数,以及flow cell对文库DNA片段的随机捕获直接相关。
在细节上,二者的关系是怎样的呢?
无意间翻到了Eric Vallabh Minikel博士曾为这个问题写的文档,描述的非常详细。
http://www.cureffi.org/2012/12/11/how-pcr-duplicates-arise-in-next-generation-sequencing/
下文内容主要参考该文部分,我在翻译的时候也稍作了修改。
我们假设建库过程中PCR扩增是均一的,共扩增了6轮循环,也即对每一个目标DNA片段产生了26=64个拷贝。上机测序时要求至少1μg DNA作为输入,那么可知其中所含unique DNA分子总量为1μg/64=15.6 ng DNA。
DNA的平均分子量约为660g/mol/bp,假设DNA文库中插入片段的平均长度为350bp,因此15.6 ng DNA中共包含15.6ng/(660*109ng/mol/bp*350bp)*(6.022*1023molecules/mol) ≈ 4.1*1010个unique DNA分子。
人基因组约3G,如果要以双端150bp的读数进行30×的全基因组测序,则需要产出30*3e9b/150b = 400M的reads,也就是flow cell需要捕获文库中的200M个DNA分子。
综上,测序时共输入4.1*1010个unique DNA分子(总DNA分子数量即64*4.1*1010 ≈ 2.6*1012),其中2*108个DNA分子由flow cell捕获,平均每个unique DNA分子被捕获的概率为(2*108)/(4.1*1010) ≈ 0.00488。
理论上,flow cell捕获的2*108个DNA分子中,unique DNA分子出现的次数(拷贝数,大于1即为duplication),将服从二项式分布。由于概率非常低且试验次数非常多,此时二项式的渐近极限-泊松分布可以更好地建模。
#R 语言中操作
x = seq(0,64,1)
x1 = seq(1,64,1)
x1lab = "unique DNA分子出现的次数"
ylab = "概率"
par(mfrow = c(2, 1))
barplot(dpois(x, lambda = 0.00488), names.arg = as.character(x), xlab = x1lab, ylab = ylab)
barplot(dpois(x1, lambda = 0.00488), names.arg = as.character(x1), xlab = x1lab, ylab = ylab)

可知,尽管上机文库中,平均每个unique DNA的拷贝数为64,但unique DNA分子被测序1次的概率仍然远大于2次及以上,对于2次及以上的情况,去除duplication后仍然还能保留一对unique reads。
然后统计duplication的比例,结果是0.0012,即0.12%。
1 - sum(dpois(x1, lambda = 0.00488)/x1)/(1-dpois(0, lambda = 0.00488))
也就是说,对于建库过程中执行6轮PCR的情形下,尽管平均每一个目标DNA片段产生了64个拷贝,但测序结果中理论上也仅出现0.12%的duplication率。
如上提到,目前市面上的建库试剂盒差不多都需要10-15轮PCR。然后我们再看建库过程中,PCR扩增了12轮的情形,这看起来是相当多的循环次数,因为理论上每一个目标DNA片段将产生212=4096个拷贝。
在上机文库的DNA总量仍为1μg DNA,不考虑PCR偏好性的情况下,共包含6.4*108个unique DNA分子。同样为人基因组的双端150bp的30×全基因组测序,平均每个unique DNA分子被flow cell捕获的概率为(2*108)/(6.4*108) ≈ 0.3125。
此时模拟测序结果中,理论上的unique DNA分子出现的次数以及duplication的比例。
x1 = seq(1,64,1)
x1lab = "unique DNA分子出现的次数"
ylab = "概率"
barplot(dpois(x1, lambda = 0.3125), names.arg = as.character(x1), xlab = x1lab, ylab = ylab)
1 - sum(dpois(x1, lambda = 0.3125)/x1)/(1-dpois(0, lambda = 0.3125))

结果约为0.0767,即7.67%。
也即表明,尽管上机文库中平均每个unique DNA的拷贝数为4096,最终测序结果中也仅仅不到8%的duplication比例。
这回就能够理解,为什么二代测序通常不会产生糟糕的duplication比例的原因的吧。
参考资料
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
(请备注姓名-学校/企业-职务等)



























