让Python告诉你,最近疯传的SIR传染病模型是什么?

最近看到在网上传的一张SIR传染病模型的图,很多人应该对这个模型不是很了解,今天就讲一下这个模型。这一篇只讲学术,不讨论别的。
SIR模型是传染病模型中最经典的一个,类似的还有SI和SIS两种。SIR是三个单词首字母的缩写,其中S是Susceptible的缩写,表示易感者;I是Infective的缩写,表示感染者;R是Removal的缩写,表示移除者。这个模型本身是在研究这三者的关系。在病毒最开始的时候,所有人都是易感者,也就是所有人都有可能中病毒;当一部分人在接触到病毒以后中病毒了,变成了感染者;感染者会接受各种治疗,最后变成了移除者。这三者的关系如下图所示:
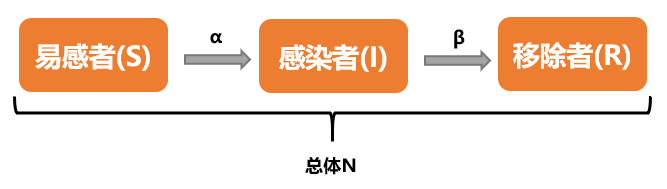
在病毒最开始的时候S=N,然后S以每天α的速度变到I,I又以每天β的速度变到R,不同时刻t下这三者的关系为:
N(t) = S(t) + I(t) + R(t)S(t+1) = S(t) - αS(t)I(t+1) = I(t) - βI(t)R(t+1) = R(t) + βI(t)
如果我们想要获取某个时间t对应的S、I、R人数,需要知道α和β值,以及S0和I0值。这个模型可以利用Python实现,具体代码如下:
%matplotlib inlineimport scipy.integrate as spiimport numpy as npimport pylab as plalpha=1.4247beta=0.14286TS=1.0 #观察间隔ND=70.0 #观察结束日期S0=1-1e-6 #初始易感人数I0=1e-6 #初始感染人数INPUT = (S0, I0, 0.0)def diff_eqs(INP,t):'''The main set of equations'''Y=np.zeros((3))V = INPY[0] = - alpha * V[0] * V[1]Y[1] = alpha * V[0] * V[1] - beta * V[1]Y[2] = beta * V[1]return Yt_start = 0.0t_end = NDt_inc = TSt_range = np.arange(t_start, t_end+t_inc, t_inc) #生成日期范围RES = spi.odeint(diff_eqs,INPUT,t_range)pl.subplot(111)pl.plot(RES[:,0], '-g', label='Susceptible')pl.plot(RES[:,1], '-r', label='Infective')pl.plot(RES[:,2], '-k', label='Removal')pl.legend(loc=0)pl.title('SIR_Model')pl.xlabel('Time')pl.ylabel('Numbers')pl.xlabel('Time')
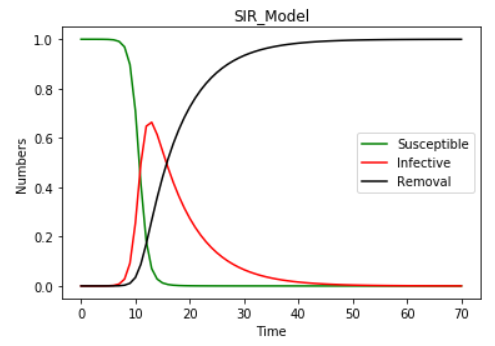
最后运行上面的代码可以得到如下结果图,上面这张图表示了随着时间t的变化S、I、R人数的一个变化趋势。
这个模型有两个假设条件:
1.一段时间内总人数N是不变的,也就是不考虑新生以及自然死亡的人数
2.从S到I的变化速度α、从I到R的变化速度β也是保持不变的
在实际环境中上面的两个假设一般是不太容易满足的,所以得出来的结果会和实际数据有偏差。
评论
