The Innovation | clusterProfiler:聚焦海量组学数据核心生物学意义
高通量组学数据功能解读中,功能富集分析是至关重要的一步,相关软件繁多但大多数仅针对极少量的模式生物开发,无法支持大量非模式生物的分析诉求。功能分析依赖准确的功能注释,但许多软件在发表文章之后并未及时更新内置的功能注释。2016年,Nature Methods文章指出,高达42%的相关工具内置注释超过五年未更新,用户基于此类工具的数据挖掘,结论反应的仅是学界五年前的生物学知识积累,颇有时光倒流的感觉。尤为重要的是,基于旧有注释,大约只能捕获到最新数据库中26%的生物学过程或通路。我们开发初版clusterProfiler时,就致力解决这些困扰领域的普遍问题,并在十年间持续进行工具的更新和维护后,推出 clusterProfiler 4.0版本(https://bioconductor.org/packages/clusterProfiler/)。
同行评论
为满足包括模式生物和非模式生物在内的几千个物种的GO分析,clusterProfiler4.0衔接支持最新完整的OrgDb(全基因组注释,可以使用AnnotationHub在线获取),同时在线实时衔接KEGG数据库,从而实现基于最新的KEGG注释,分析几千个物种的通路。为方便用户同时进行过表征分析(ORA)和基因集富集分析(GSEA),升级版提供了通用的接口支持不同来源的注释数据,尤其是新物种GO注释、COVID-19等新的功能注释,进行富集分析。针对日益复杂的实验设计,clusterProfiler4.0可对比分析多组数据,允许用户针对功能谱进行比较分析,完整提供对比分析中涉及的关键辅助工具,包括ID转换(如ENTREZ, ENSEMBL, SYMBOL等)、结果操作(如条件过滤,排序、计算衍生变量)等,并对应实现可视化展示,让结果解读更为便捷高效。
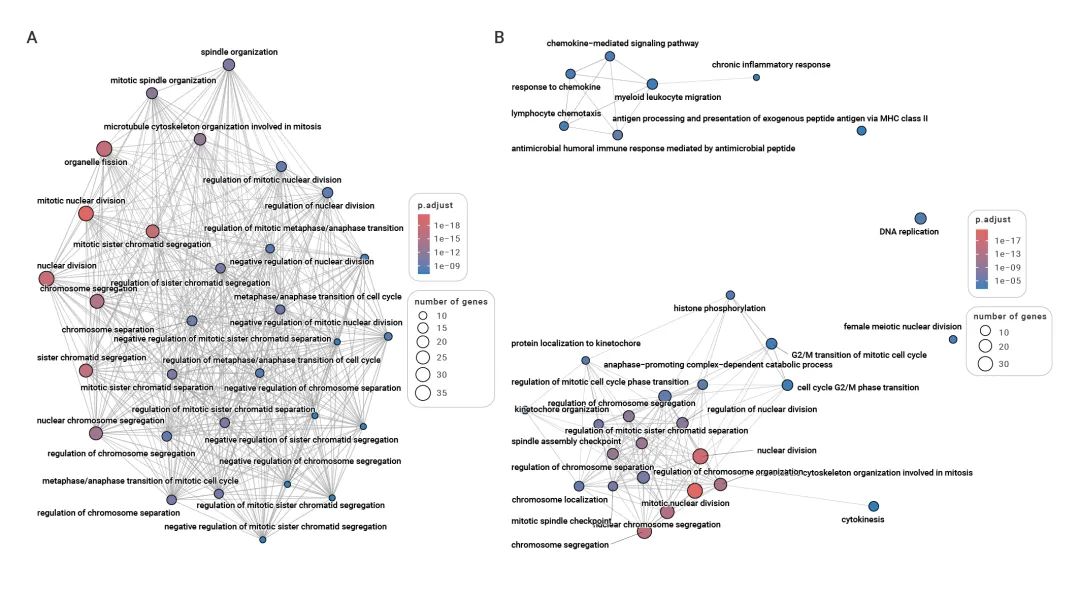
图1 GO富集分析
富集分析中,GO术语以有向无环图的形式组织,具有冗余性,一个节点显著富集,其父节点可能只因包含这个子节点而被富集,但其他子节点并无贡献,这会影响对关键节点的分辨。然而在最常用的GO富集结果柱状图展示时,此种关联信息缺失,让上述问题不易察觉,造成解读偏差。举例而言,图1A使用Enrichment Map可视化30个最显著的基因集,看似形成一个紧密连接的复杂功能模块,其实是因上述冗余问题造成的假象。利用clusterProfiler4.0提供的去冗余功能,关键网络(图1B)可以给我们更为凝练的全局信息,明确地显示核心的功能模块。
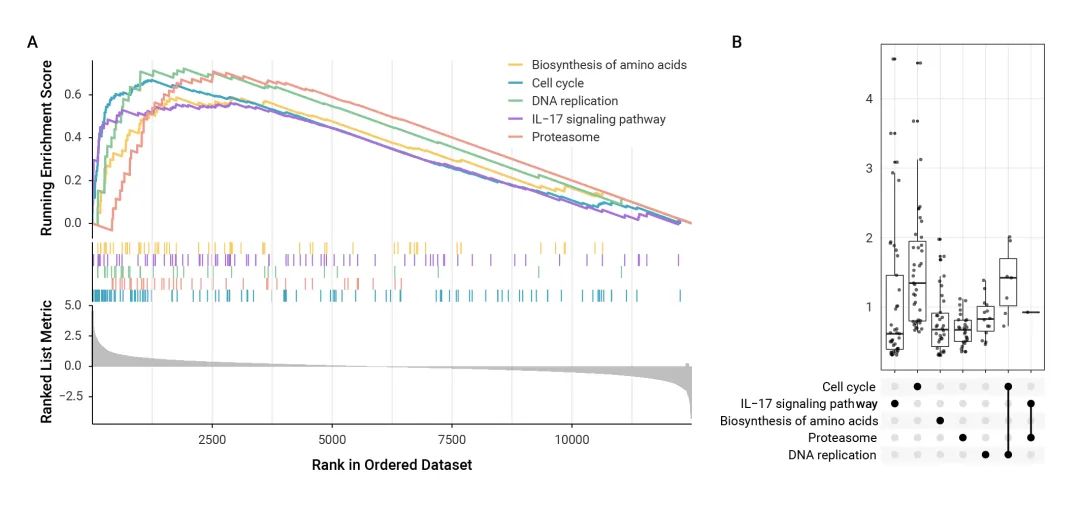
图2 KEGG富集分析
类似的,KEGG通路分析中,不同通路间存在基因交集,形成串扰(crosstalk)。特定生物学过程中,串扰基因发生活性改变,往往影响多个通路,因而无法将该生物现象背后的生物通路简单归结为某一条的作用。我们对一份乳腺癌表达数据进行GSEA分析,依据NES(归一化的富集分数)作为通路被干扰程度的指标,选取NES绝对值最大的五条通路包括Biosynthesis of amino acids, Cell cycle, DNA replication, IL-17 signaling pathway 和Proteasome进行展示。图2A作为经典的GSEA富集分析呈现方式,主要传递的信息是:分析涉及的属于某一通路的基因,在所有基因排序中是否倾向聚集于曲线头部(代表通路激活)或尾部(代表通路抑制)。图中间的色块分布虽然可以显示相应通路的基因在五条通路曲线相应的位置,但无法展现通路间的基因交集,交集基因的上下调是否一致,对了解通路之间的串扰大有帮助。图2B将这些信息呈现出来,可以看出各通路中特有基因(图2B下单个黑点)以及不同通路之间交集基因的上下调表达情况(fold change),有助于我们审视通路间串扰以及这些串扰基因在通路交互调控中的作用。
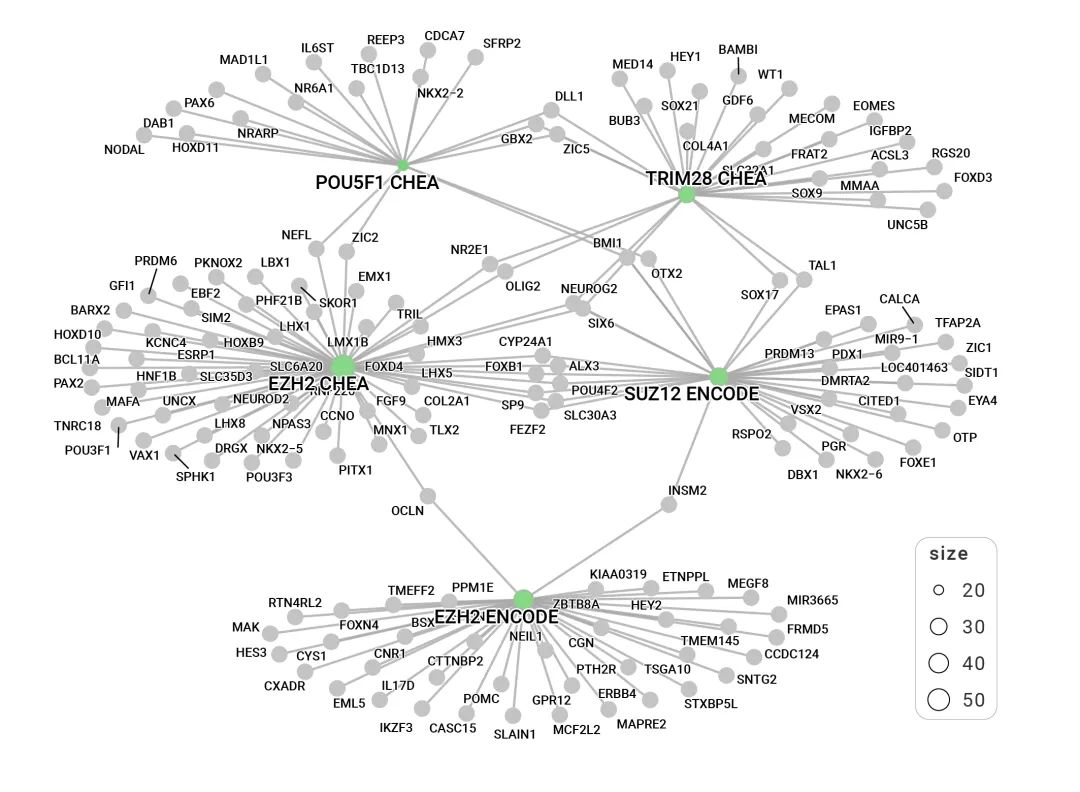
图3 基因组坐标的通路分析
在通路分析中,当下关注的靶标通常是调控关系、相互作用等功能注释比较透彻的蛋白编码基因。随着数据的不断积累,一些非编码基因也出现了功能分析的需求。clusterProfiler4.0提供的通用策略可以实现对此类基因的功能分析。首先为缓解当前大量非编码区域尚欠缺功能注释的问题,我们课题组开发了ChIPseeker,分析测序数据中非编码RNA、转录因子结合位点、转座子等的基因组坐标,将该坐标和相邻的基因关联起来,然后利用clusterProfiler4.0完成邻近基因的功能分析,为洞察非编码基因生物学功能提供依据。联合ChIPseeker和clusterProfiler可以对ChIP-seq, DNase-seq, ATAC-seq等各种染色体开放区域测序、RRBS等各种表观遗传测序数据获得的基因组坐标进行功能分析。以图3举例展示,我们选取一个转录因子CBX6的ChIP-seq数据集,得到其结合序列的基因组坐标,注释其邻近基因,确定其在哪些转录因子已知的互作基因集中富集,成功鉴定四个关键转录因子(图3.绿色节点),以往文献报道它们均与CBX6有功能的关联,提示可能共同参与转录调控。这类ChIPseeker和clusterProfiler联用策略,可以拓展到任意的基因区域集合,探索它们在各类调控因子靶基因中的富集情况,从而发现其关键的调控因子以及调控因子间的相互作用。也可以探索候选基因区域在GO,KEGG,Reactome, WikiPathways等注释信息中的富集,帮助发现参与调控的此类通路。
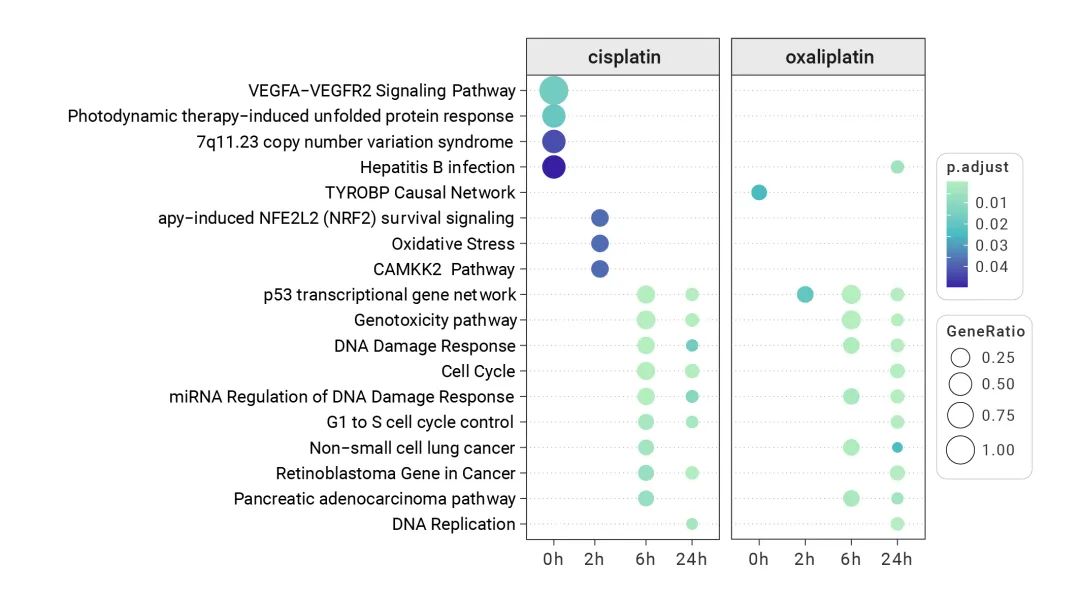
图4 比较多组数据功能谱
多处理实验设计为全面理解分子机制、药物作用等提供方法,但也给数据可视化带来挑战。如图4显示,该研究有2种药物处理、4个时间点,共8组数据,clusterProfiler4.0中我们加入了formula的支持,实现对多组数据对比分析。通过指定formula, 如Gene~time+treatment, 把基因按照时间点和不同的处理进行分组,分别对8组数据进行富集分析,最后以x轴为时间点,分面为不同的处理组,实现8组数据功能谱的清晰呈现和对比,明显看出不同的药物处理虽然起始效应存在显著差异(0h,2h),但后期效应却有很高的一致性(6h,24h)。
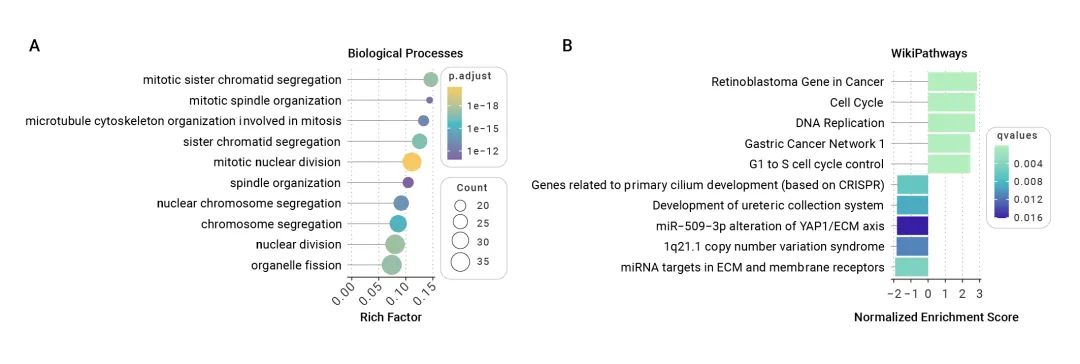
图5 使用ggplot2可视化分析结果
上述图1-4使用我们课题组开发的enrichplot包进行可视化,考虑到兼容用户常用的数据操作软件,clusterProfiler4.0同时实现了tidy接口,允许用户使用dplyr动词进行数据的操作和ggplot2进行数据的可视化(图5)。例如,图5A展示了使用dplyr中的mutate为结果增加rich factor,然后使用ggplot2对富集结果以棒棒糖图的形式呈现;图5B则通过arrange,group_by和slice对GSEA富集结果进行排序、分组和切割,最终分别获得上、下调通路中NES(归一化富集分数)最大的五条通路,并使用柱状图进行呈现。衔接dplyr和ggplot2可以方便用户更为灵活自主地对富集结果进行探索。
总结和展望
综上,clusterProfiler4.0充分考虑目前富集分析中的核心诉求,使用最新的功能注释以保证结论及时反映最新共识,应对不同的组学数据整合分析需求, clusterProfiler4.0允许衔接外部功能数据库、支持多组数据的比较分析以及提供tidy接口助力结果解读。目前,clusterProfiler已被整合进超过30个的同行分析软件中,助力不同场景下的功能分析,相信clusterProfiler4.0未来将发挥更大的作用,助力研究者更高效地解读生物医学数据及建立更可靠的机制假说。
扫二维码|查看原文
原文链接:https://www.cell.com/the-innovation/fulltext/S2666-6758(21)00066-7
本文内容来自Cell Press合作期刊The Innovation第二卷第三期以Article发表的“clusterProfiler 4.0: A universal enrichment tool for interpreting omics data” (投稿: 2021-05-08;接收: 2021-06-29;在线刊出: 2021-07-01)。
DOI: https://doi.org/10.1016/j.xinn.2021.100141
引用格式:Wu T., Hu E., Xu S., et al. (2021). clusterProfiler 4.0: A universal enrichment tool for interpreting omics data. The Innovation. 2(3),100141.
余光创,博士,教授,博士生导师。南方医科大学基础医学院生物信息学系主任。研究方向为多组学数据整合分析、数据可视化。开发多款生物信息学分析工具,包括clusterProfiler, GOSemSim, ChIPseeker, ggtree等,以第一作者或通讯作者发表6篇ESI高被引论文,入选爱思唯尔2020年中国高被引学者。
个人网页:http://yulab-smu.top
期刊简介
扫二维码 | 关注期刊官微
The Innovation 是一本由青年科学家与Cell Press于2020年共同创办的综合性英文学术期刊:向科学界展示鼓舞人心的跨学科发现,鼓励研究人员专注于科学的本质和自由探索的初心。作者们(Volume 1 Issue 1 -- Volume 2 Issue 2)来自全球24个国家;每期1/3-1/4通讯作者来自海外。目前有183位编委会成员,来自21个国家;51%编委来自海外;包含1位诺贝尔奖获得者,26位各国院士;领域覆盖全部自然科学。The Innovation已被DOAJ,ADS,Scopus等数据库收录。
期刊官网1(Owner):
www.the-innovation.org
期刊官网2(Publisher):
www.cell.com/the-innovation/home
期刊投稿(Submission):
www.editorialmanager.com/the-innovation
商务合作(Marketing):
marketing@the-innovation.org
Logo|期刊标识

See the unseen & change the unchanged
创新是一扇门,我们探索未知;
创新是一道光,我们脑洞大开;
创新是一本书,我们期待惊喜;
创新是一个“1”,我们从此走起。

第2卷第2期




往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集