iMeta封面 | 宏蛋白质组学分析一站式工具集iMetaLab Suite(加拿大渥太华大学Figeys组)
iMetaLab Suite:宏蛋白质组学分析一站式工具集

https://doi.org/10.1002/imt2.25
SHORT COMMUNICATION

●2022年5月21日,加拿大渥太华大学医学部药学院Daniel Figeys团队在iMeta在线发表了题为“iMetaLab Suite: A one-stop toolset for metaproteomics”的文章。
● 该研究开发了一套名为iMetaLab Suite的免费一站式分析工具集,涵盖了宏蛋白组学中最常用的功能、分类和统计分析。
● 第一作者:李乐园、宁志斌
● 通讯作者:Daniel Figeys (dfigeys@uottawa.ca);
Caitlin M.A. Simopoulos
宏蛋白质组学是一个相对新兴的领域,主要研究人类、动物、植物和环境的复杂微生物组中的蛋白质组成。宏蛋白质组学研究所需的生物信息学工作流程包含蛋白质鉴定(数据库搜索)、蛋白质定量到下游的功能和分类分析。由于这其中蕴含的难度和挑战性,一定程度上限制了其他领域微生物组研究人员对宏蛋白组学的使用和深入探索。为了克服这些挑战,我们开发了一套名为iMetaLab Suite的工具集。iMetaLab Suite包括以下部分。1)MetaLab Desktop自动化数据库搜索软件,为微生物组的蛋白质鉴定和定量提供便利;2)iMetaReport自动化报告,在数据库搜索后自动展示搜索结果和数据集概况;3)iMetaShiny交互式在线工具集,涵盖了宏蛋白组学中最常用的功能、分类和统计分析。
关键词:微生物组,宏蛋白质组学,生物信息学,数据库搜索,统计分析,可视化

● 为研究者提供一站式宏蛋白质组学解决方案
● 提供数据库搜索及包含物种分类和功能信息的自动化报告
● 包含常用宏蛋白质组学分析的交互式在线工具
Bilibili:https://www.bilibili.com/video/BV1gT4y1B75Y/
Youtube:https://youtu.be/FwUB3zzMEKQ
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
引 言
在微生物细胞中,约占细胞干重50%的蛋白质在微生物生命活动中发挥着多样的作用。因此,使用适当的技术来理解蛋白质的组成和微生物组的功能非常重要。宏蛋白组学即为这样一种技术。在常规宏蛋白质组学流程中,从微生物组样本中提取的总蛋白经过酶解成为肽段,采用液相色谱-串联质谱法(LC-MS/MS)对这些肽段进行分析。将得到的MS/MS谱图与数据库中序列的理论谱图进行比对,获得序列的鉴定结果。上述过程对于单一物种的蛋白质组学分析相对容易,这是由于数据库来源于单一物种、数据库大小通常合理。例如,UniProt数据库中E. coli K-12菌株在的FASTA序列数据库包含4375个蛋白质序列(文件大小1845 KB)。然而,复杂微生物组的参考蛋白质目录由于涵盖了尽可能多的物种,其数据库规模急剧增加。例如人类肠道微生物组的综合基因目录(Integrated Gene Catalog,IGC)的蛋白质数据库包含近990万条序列,文件大小3.17 GB[1]。也就是说,IGC数据库与E. coli K-12数据库相比增加了约两千倍。采用此类大规模蛋白质目录作为数据库,不仅对运算能力构成挑战,而且对目标-诱饵搜索策略的错误发现率(FDR)估计模型效率产生负面影响。为了克服上述挑战,我们此前研发了采用迭代式数据库搜索策略的MetaPro-IQ流程[2],为MaxQuant搜索生成一个缩小的数据集特异性数据库。传统MaxQuant搜索的输出产生定量的肽段和蛋白质表格。对于复杂的微生物组样本,从这些搜索结果中得出准确的生物分类学匹配和功能注释也具有挑战性。此外,由于蛋白质同时包含生物分类学和功能信息,宏蛋白质组学的下游数据分析与传统蛋白质组学相比也增加了一维难度。上述挑战的累加效应导致非生物信息专家应用宏蛋白质组学技术较为不便。
为解决上述难题,我们开发了iMetaLab工具集,包含完整的数据库搜索-蛋白质鉴定和定量流程(MetaLab桌面软件)[3],搜库后自动生成的网页报告(iMetaReport),以及一系列交互式数据分析和可视化工具(iMetaShiny)。iMetaLab数据库搜索实现了 MetaPro-IQ流程[2]的自动化。早期MetaPro-IQ流程需要使用者具有一定的生物信息技能,为了迎合科研人员对该流程日益增长的使用需求,我们将该流程写入MetaLab桌面软件,并逐步增加了谱图聚类缩库方法[3],翻译后修饰分析[4],以及软件内置的 iMetaReport自动化报告模块。我们致力于将iMetaLab工具集分享给微生物组学研究社群,本工具集目前已有来自全世界160余不同研究单位的注册用户 。iMetaLab是一个面向科研应用免费的一站式宏蛋白质组学工具集,我们将持续动态更新工具集的内容。
结 果
iMetaLab Suite概览
iMetaLab工具集(图1)可通过https://iMetaLab.ca 网站获取。MetaLab桌面版本可免费由该网站下载,也可直接通过邮件联系技术支持团队(techteam.metalab@gmail.com)获取最新版本下载链接。MetaLab桌面软件的输入为用户提供的LC-MS/MS原始文件,实验设计对照表(可选),以及用户设置的搜库参数。详细的MetaLab桌面软件使用说明请见https://wiki.imetalab.ca/。在默认设置下,MetaLab将执行数据库搜索流程生成一系列结果表格,主要包括鉴定总结,肽段,蛋白质小组,分类学和功能注释表格。这些表格涵盖宏蛋白质组学下游数据分析最常用的数据。其中,我们为分类学和功能注释结果生成了不同格式的表格,以满足各种数据可视化需求。
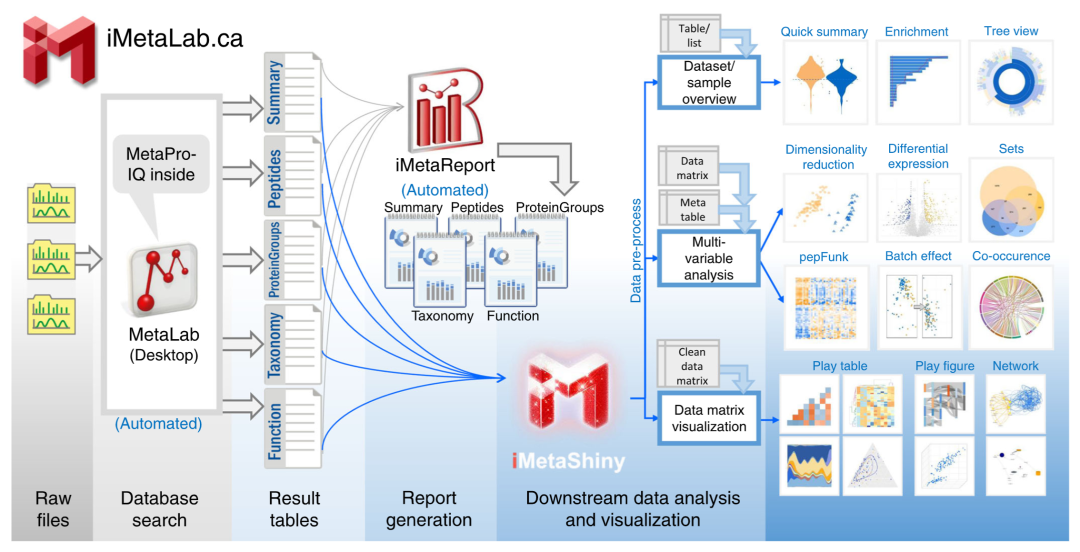
图1. iMetaLab工具集内容概览
用户将原始质谱文件加载入MetaLab桌面软件以进行自动化宏蛋白质组学数据库搜索。搜库完成后将产生一系列结果表格。软件将基于搜库结果表格自动生成iMetaReport网页报告,涵盖鉴定总结,肽段,蛋白质小组,生物分类学和功能注释报告。用户可使用结果表格在iMetaShiny网页进行各种数据统计分析和可视化。
MetaLab数据库搜索结束后,iMetaReport自动化报告将由软件生成,并向用户发送弹窗提示查看网页报告。iMetaReport包含五个主要的页面,分别展示鉴定总结,肽段,蛋白质,生物分类学和功能注释的数据概况。当搜库设置时的用户输入包含实验设计(meta信息)时,可视化报告可帮助用户预览不同实验分组间样本宏蛋白质组组成是否具有差异。iMetaReport的示例可在https://report.imetalab.ca查看。
iMetaShiny包含一系列宏蛋白质组学常用数据分析和可视化的在线交互式Shiny应用 。该系列应用可分为三个主要类型。第一类应用提供数据的概览,包括LC-MS/MS鉴定的快速结果报告,包含生物分类学和功能注释的富集分析,以及基于用户输入NCBI ID的生物分类学可视化。第二类应用为多元统计分析工具,包括降维分析工具(PCA, PLS-DA,t-SNE),差异蛋白表达分析,组间对比分析,pepFunk[5](肽段为中心的分析方法),批次效应分析/矫正,共现网络分析等。第三类工具为绘图类,基于用户输入的特定格式表格进行数据的可视化。在每个Shiny应用中我们都提供可以下载的示例数据,以便于用户参考和准备数据表格。除上述工具外,我们也提供诸如96孔板随机化工具、样本随机化工具等协助用户进行前期实验设计。我们还在不断纳入更多的分析应用。
案例一:数据库搜索和自动化报告
我们应用前期发表的一组实验数据,该实验将体外培养的个体微生物组样本分为双氯芬酸(非甾体抗炎药物)处理组和对照组,每组三个技术重复[6]。蛋白质经酶解后采用Orbitrap Q-Exactive 进行1.5小时梯度的质谱分析。所获得的六个原始质谱文件采用MetaLab桌面软件(V2.2版本)进行搜库,采用IGC数据库和软件默认参数进行搜库。在这里我们使用了一台Windows服务器(两个Intel Xeon E5649处理器, 96 GB RAM)中的四个线程,搜库过程用时14小时。数据库搜索结束后产生了一系列结果文件,包括summary.txt, peptides.txt,proteinGroups.txt,BuiltIn.taxa.all.csv,functions.tsv等,并自动创建了iMetaReport报告html页面。其中,鉴定总结页面由summary.txt生成。在本案例中,报告显示鉴定到21600条肽段序列和6601个蛋白质小组,平均MS/MS鉴定率为44.9% (图2A-B)。肽段和蛋白质报告分别使用了peptides.txt和proteinGroups.txt作为输入,两个报告包含显示数据整体质量的系列重要的参数,例如离子带电荷状态,得分分布 (图2C), 强度等。肽段和蛋白质报告中也提供热图和主成分分析得分图以直观展示样本间对比,这些分析分别采用log10 转化的肽段强度或蛋白质小组(proteinGroup)的无标记定量(LFQ)强度,应用稳健序列算法进行了插值以去除可能的数据稀疏性。在本示例中,处理组和对照组在PC1上显示出明显的区分(图2D)。如果用户在数据库搜索中设置了实验设计信息,蛋白小组报告将自动根据LFQ强度在用户输入的实验组之间进行方差分析(ANOVA),并给出矩阵和配对比较的FDR调整后的p值(图2E)。在生物分类学报告页面中,显示各个分类级别的鉴定数量,α和β多样性,以及微生物组成的堆积条形图。在本案例中,可以清楚地观察到双氯芬酸处理组合对照组在属水平上蛋白质生物量贡献的差异(图2F)。功能报告包含各种功能数据库的注释结果可视化,例如Clusters of Orthologous Groups(COGs)(图2G),也提供功能水平的热图和PCA。如果用户在搜库前没有设置实验设计信息,在搜库结束后用户可以在保留其他搜库结果文件的前提下,仅删除报告文件并在搜库界面重新设置信息并运行。MetaLab软件会检测已有的文件并跳过已经完成的搜库步骤,直接运行报告生成。https://report.imetalab.ca网页提供了一个完整的iMetaReport报告示例。需要注意的是iMetaReport的初衷是便于用户快速检查数据质量和实验效果,我们推荐用户使用iMetaShiny进行后续数据分析。
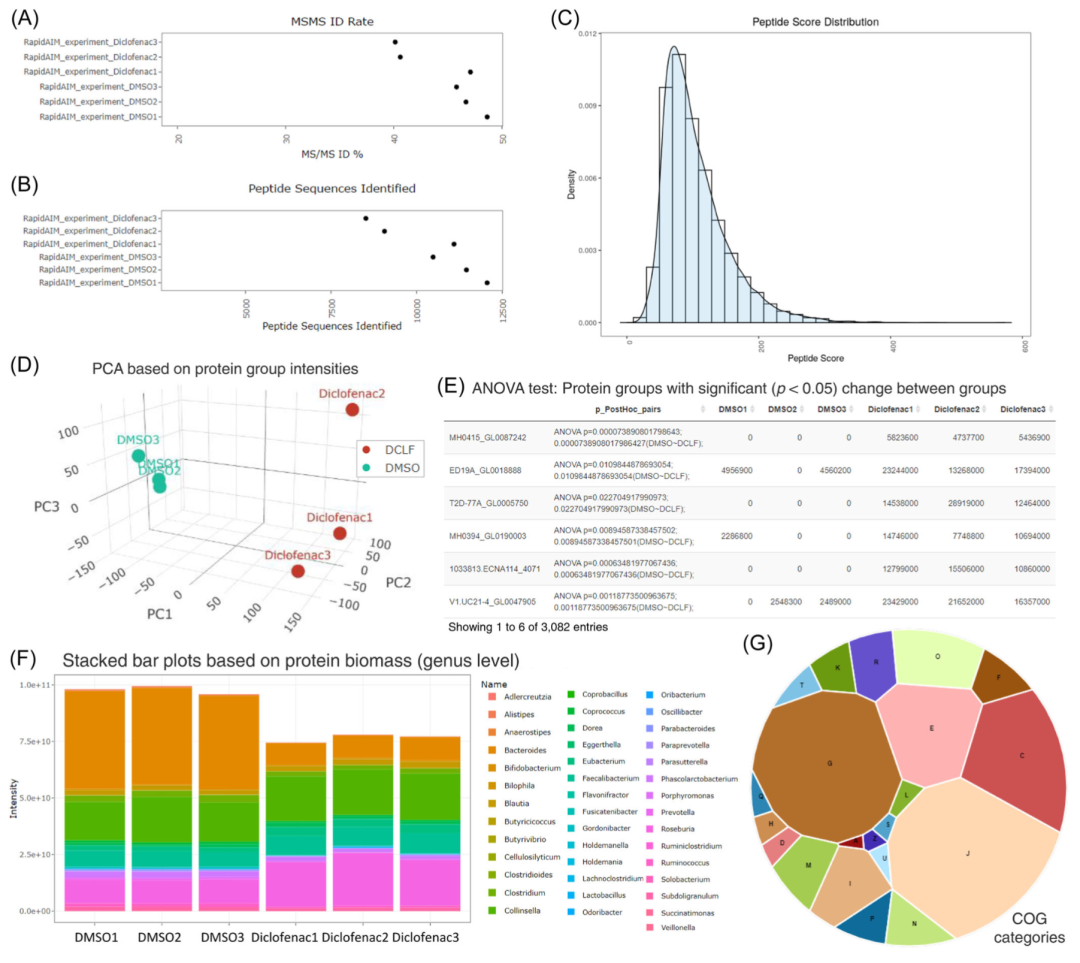
图2. iMetaReport示例
A. 鉴定总结报告:每个样本中的MS/MS 鉴定率。B. 鉴定总结报告:每个样本中的肽段鉴定数量。C. 肽段报告:数据集中的肽段得分分布。D. 蛋白质小组报告:基于蛋白质小组强度的主成分分析。E. 蛋白质小组报告:基于蛋白质小组强度的方差分析。F.生物分类学报告:基于属水平蛋白质生物量的堆积条形图。G. 功能报告:数据集中COG分类的组成
案例二:差异表达的蛋白质组及其生物分类学和功能分析
我们继续使用案例一的搜库结果进行案例二的演示。采用蛋白质小组(LFQ)强度进行差异蛋白质表达分析,在分析界面https://shiny.imetalab.ca/Volcano_plot/上传数据表格后,打开数据预处理选项进入数据预处理界面,过滤掉有75%以上缺失值的行,并按列进行了标准化。用户也可以直接输入预处理过的蛋白表达表格,并且跳过数据预处理选项直接进入差异蛋白质表达分析。进入分析后,在本案例中使用默认的统计参数和平滑曲线阈值来确定显著变化的蛋白质小组。页面右侧显示自动生成火山图,并随用户参数调整而实时更新,如图3A所示。在双氯芬酸作用下,该微生物组样本呈现95个强度显著增加和117个强度显著降低的蛋白质小组。用户可在结果下载选项卡中下载分析结果表格。下面我们对这些差异表达的蛋白质小组进行富集分析(https://shiny.imetalab.ca/metaproteomics_enrichment/)。将蛋白质ID输入到该工具中,本示例选择了功能和生物分类学关联分析,以COG为功能注释类型。该工具将通过内置的数据库为蛋白质ID分配功能和生物分类学信息,并进入可视化界面。我们选择Circos可视化分析,如图3B和C所示,显著增加的功能大部分来自Enterobacterales,而显著下降的功能多来自Bacteroides。
案例三:以肽段为中心的功能富集分析
使用LFQ蛋白质小组强度之外,还可以应用我们前期开发的pepFunk工具(https://shiny.imetalab.ca/pepFunk/)可进行以肽段为中心的功能分析。在本案例中,我们上传peptides.txt结果表格,将DMSO组设置为对照组,将双氯芬酸组设置为处理组。我们将基因集变异分析(GSVA)迁移适配于肽段强度数据分析,生成显著富集的KEGG通路。图3D显示KEGG富集在处理组和对照组之间具有明显差别。
讨 论
我们力求通过开发和共享iMetaLab工具集,使得宏蛋白质组学生物信息分析流程的便捷性最大化,使微生物组研究领域中具有不同生物信息水平的科学家、以及传统蛋白质组学/系统生物学领域的科学家都能够更加容易地使用宏蛋白质组学这一工具。我们保持活跃地开发新的数据库搜索工作流程和策略,以及开发更多的宏蛋白组学功能、分类学和生态学分析工具,并将实时更新到iMetaLab工具集中。我们也随时欢迎用户的反馈和建议,将有助于改善用户体验和迎合多样需求。
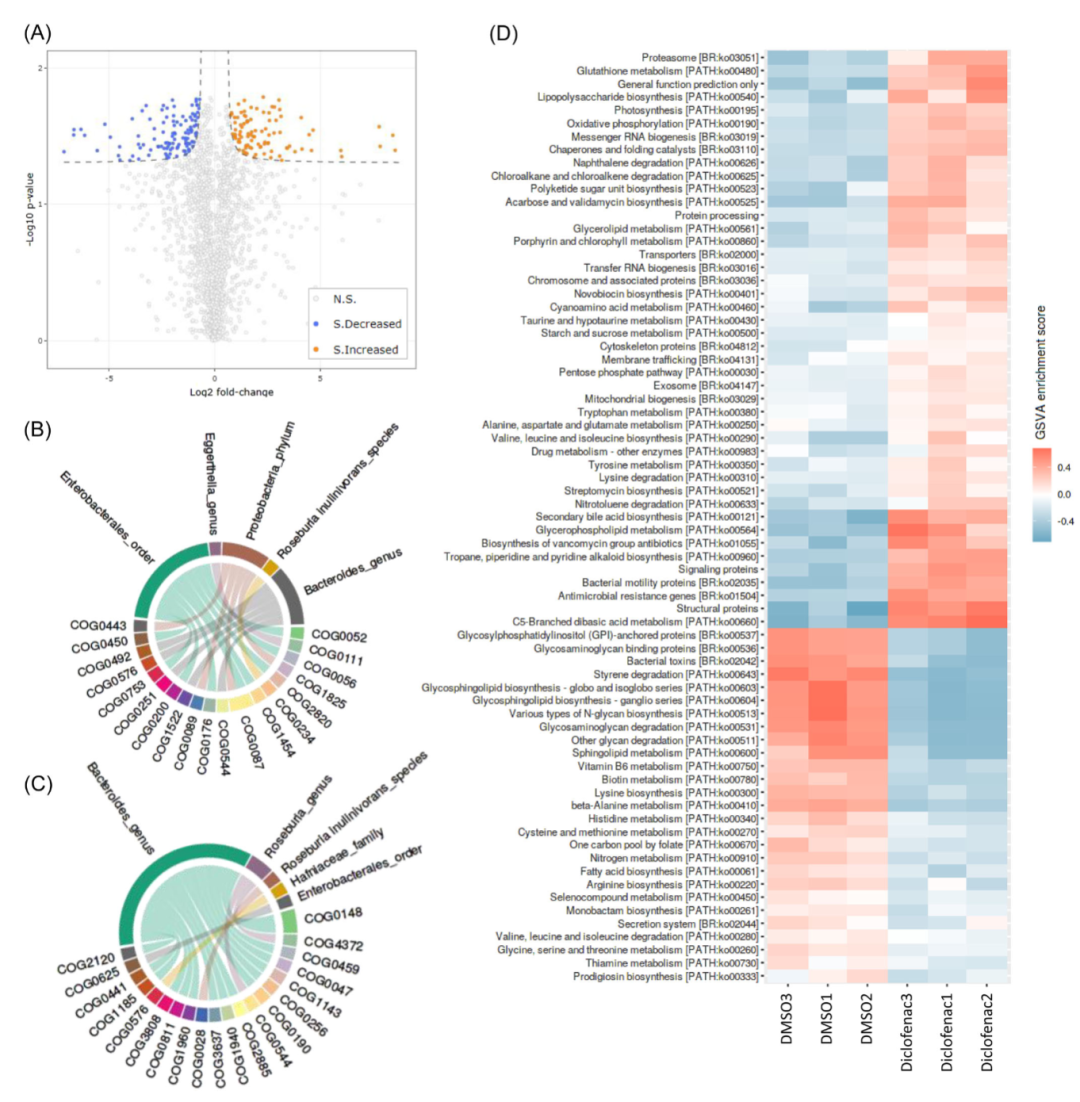
图3. iMetaShiny应用示例
A. 双氯芬酸作用下的差异蛋白质表达分析结果。橘色点代表显著增加的蛋白质小组,蓝色点代表显著降低的蛋白质小组。B-C. 差异表达的蛋白质的生物分类学-功能富集分析(采用每个蛋白质小组的第一个蛋白质,P < 0.05)。D. 热图显示双氯芬酸数据集的GSVA得分。
方法
MetaLab软件采用JAVA开发,并整合了开源第三方包/工具MzJava[7],PRIDE Cluster[8],X!Tandem[9],MaxQuant[10]和Msconvert。iMetaReport基于R Markdown[11]开发,使用了包括ggplot2[12],plotly[13],tidyverse[14],vegan[15],ggdendro,d3heatmap,pheatmap等包。服务器通过openCPU[16] 实现公开访问。用户数据库搜索结果通过MetaLab上传到openCPU服务器以生成报告。iMetaShiny应用采用R和Shiny包实现[13],使用的R包包括DT,data.table,shinyBS,htmlwidgets等,由Shiny服务器支持。这些服务器通过亚马逊云AWS托管。
致谢
本研究工作由加拿大政府通过基因加拿大-安大略基因组研究所(OGI-114),加拿大健康研究所项目(ECD-144627),加拿大自然科学与工程研究委员会(NSERC,210034),安大略省经济发展和创新部(REG1-4450)支持。D.F.致谢渥太华大学授予杰出研究主席。C.M.A.S.获得NSERC CREATE 微生物组科学与工程技术(TECHNOMISE)计划的资助。
利益冲突
D.F.是MedBiome临床微生物组学公司共同创始人。其他作者声明没有利益冲突。
作者贡献
D.F.和Z.N.设计了iMetaLab Suite工具集的框架。Z.N.为iMetaLab.ca、iMetaReport和iMetaShiny构建网络服务器和生物信息学框架。Z.N和L.L开发了iMetaReports和iMetaShiny工具。L.L.撰写了文章。K.C.开发并维护MetaLab Desktop软件。X.Z.开发了MetaPro-IQ流程。C.M.A.S.开发了iMetaShiny中的pepFunk工具。所有作者都测试了工具集,修改、阅读和认可了文章最终稿。
数据可用性声明
LC-MS/MS 测序数据通过PRIDE合作伙伴库存入ProteomeXchange Consortium,提交编号为PXD033624。数据库检索结果和报告保存在GitHub(https://github.com/northomics/iMetaLab_paper)。补充材料(图片摘要、幻灯片、视频、中文翻译版本和更新材料)可通过DOI或iMeta网站http://www.imeta.science/获取
李乐园(第一作者)
● 加拿大渥太华大学医学部副研究员(research associate)、iMeta期刊青年编委
● 本科和博士毕业于北京航空航天大学。博士后期间于Daniel Figeys教授研究团队开展药物-微生物组相互作用及宏蛋白质组学研究。在iMeta、Nature Communications、Microbiome、Gut Microbes等期刊发表文章30余篇
Daniel Figeys(通讯作者)
● 博士、教授,加拿大渥太华大学医学部药学院院长、iMeta期刊编委
● 研究方向为微生物组、宏蛋白质组学、宏基因组学、生物信息学和药物-微生物组相互作用。已在Nature,Nature Biotechnology,Gut,iMeta, Nature Communications等期刊发表文章200余篇,累计引用超过18000次,h-index 64,i-10 index 198
(▼ 点击跳转)
iMeta文章中文翻译+视频解读
iMeta|南医大孔祥清团队创建前瞻性多组学纵向原发高血压队列eHypertension
▸▸▸▸
iMeta | 深圳先进院马迎飞组开发基于神经网络分析肠道菌群的方法
▸▸▸▸
iMeta | 南医大陈连民等综述从基因组功能角度揭示肠菌对复杂疾病的潜在影响
▸▸▸▸
iMeta | 北大陈峰组综述口腔微生物组的标准化研究:从技术驱动到假说驱动
▸▸▸▸
iMeta | 电子科大林昊组开发蛋白质赖氨酸乳酸化位点预测工具DeepKla
▸▸▸▸
iMeta | 南昌大学丁霞等-水产养殖系统对中华鳖微生物组和肠道代谢组的影响
▸▸▸▸
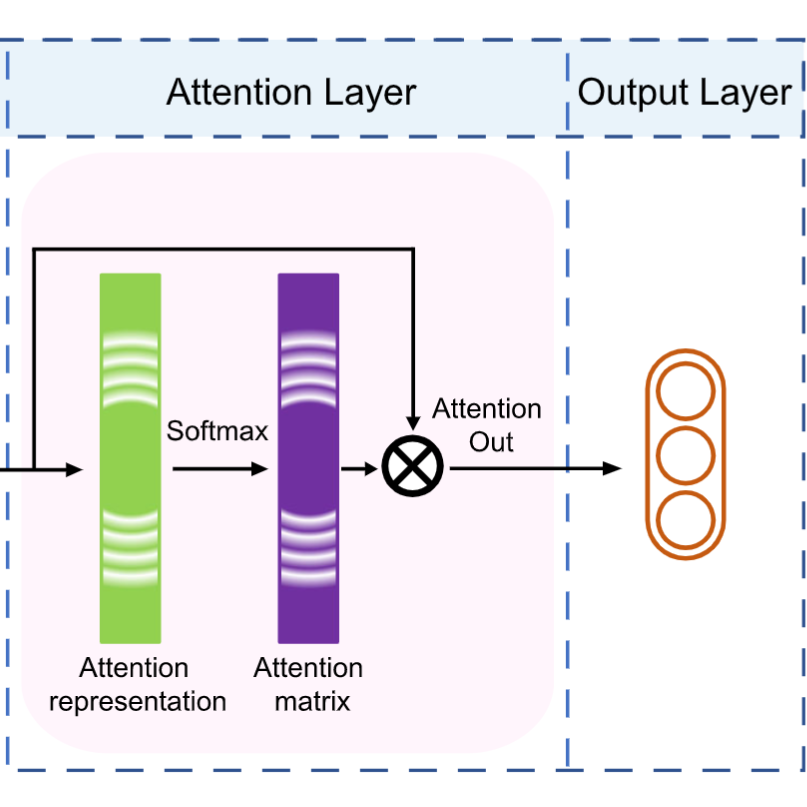
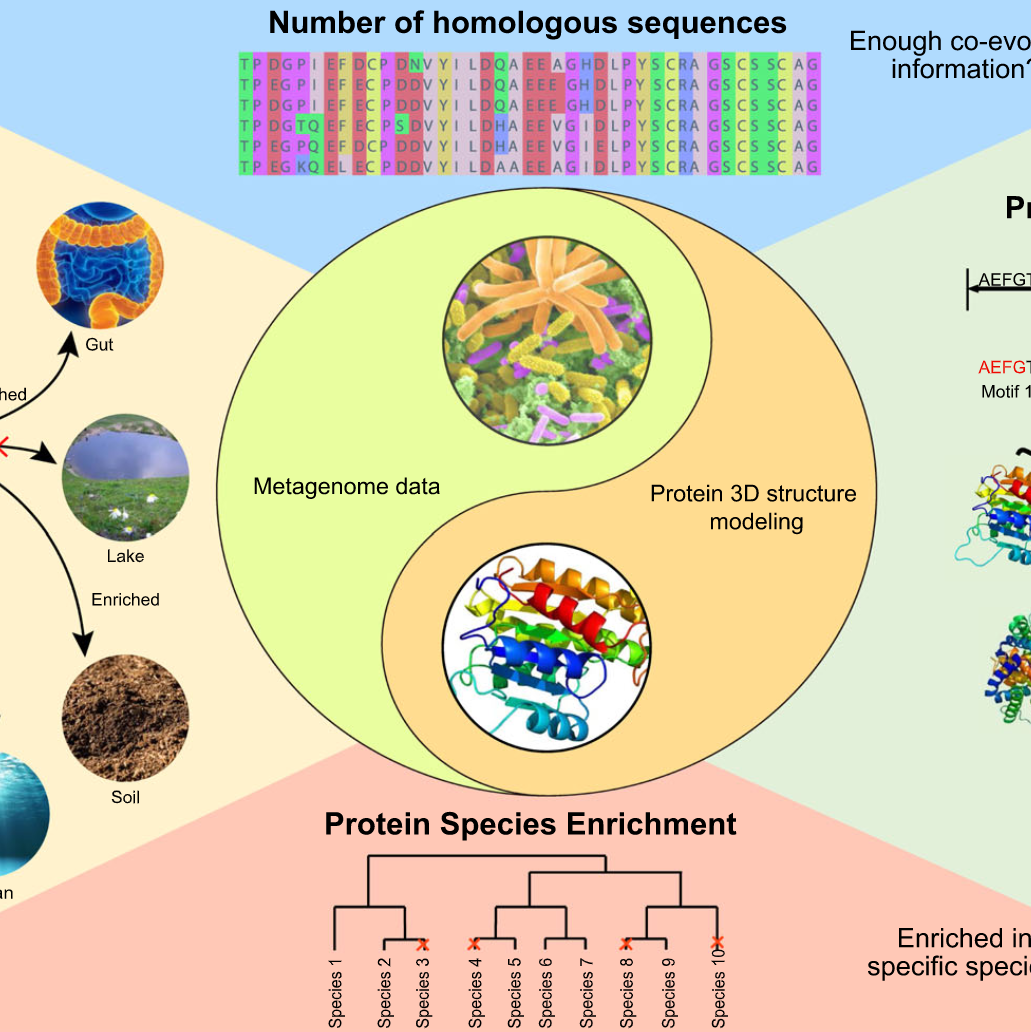
iMeta | 华中科大宁康组综述用于蛋白质结构预测的宏基因组定量分析
▸▸▸▸
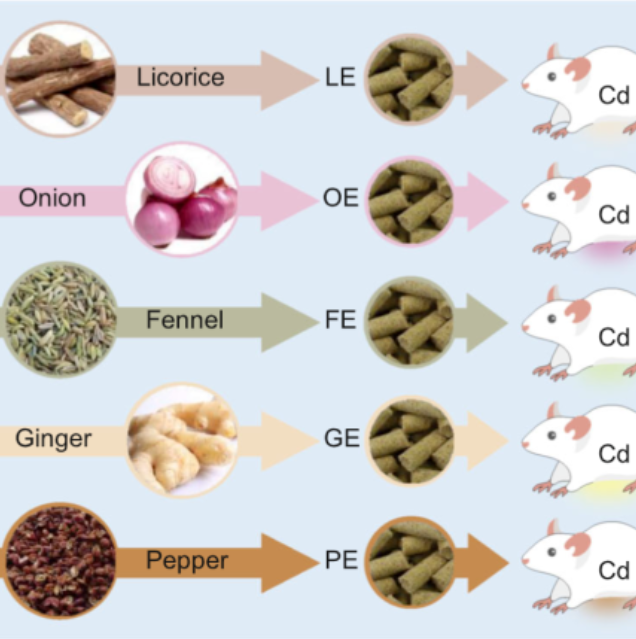
iMeta | 中科院李小方等膳食甘草促进小鼠镉解毒并调节肠道菌群代谢
▸▸▸▸
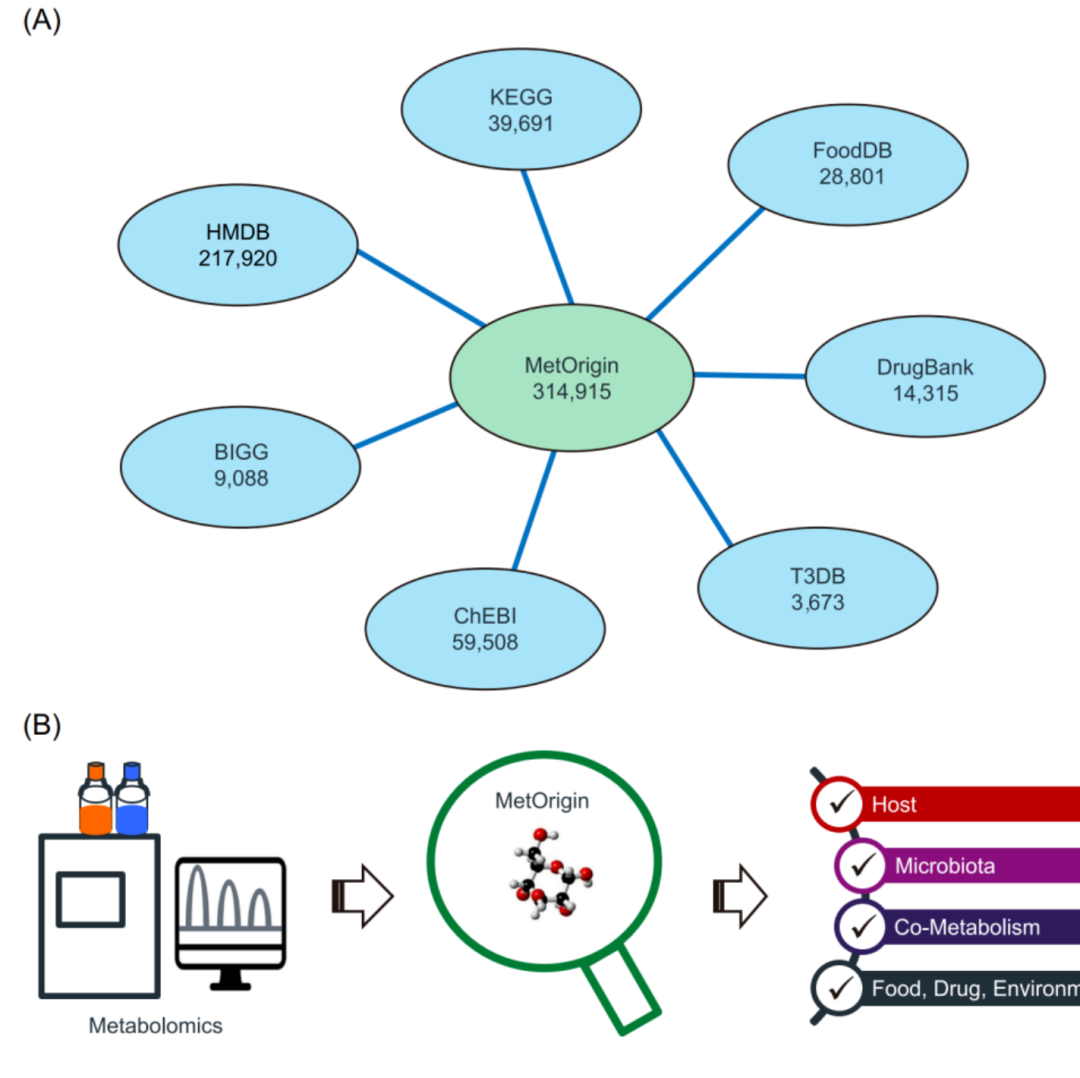
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
▸▸▸▸
iMeta | 南科大宋毅组综述逆境胁迫下植物向微生物组求救的遗传基础(附招聘)
▸▸▸▸
iMeta:高颜值高被引绘图网站imageGP

“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
微信公众号
iMeta
责任编辑
微微