走出 scipy 的深坑:用 numpy 实现散列数据网格化
手头有中国区900百帕高度上的某时刻温度数据集,这个数据集包括3个数组:经度数组、纬度数组、温度数组。各数组值域范围如下:东经28.644232° ~ 177.3808°,北纬3.2973719° ~ 59.27192°,温度205.01515°K ~ 366.69788°K,共计18952个数据点,散列在一个倒三角区域内。
data = np.load(r'D:\XufiveGit\CSDN\data\giirse.npz')lon = data['lon']lat = data['lat']tmp = data['tmp']lon.shape, lat.shape, tmp.shape((18952,), (18952,), (18952,))lon.min(), lon.max(), lat.min(), lat.max()(28.644232, 177.3808, 3.2973719, 59.27192)tmp.min(), tmp.max()(205.01515, 366.69788)
将这些数据绘制在等经纬平面上,看起来是这样的:
import matplotlib.pyplot as pltplt.scatter(lon, lat, s=1, c=tmp, cmap=plt.cm.hsv)plt.show()
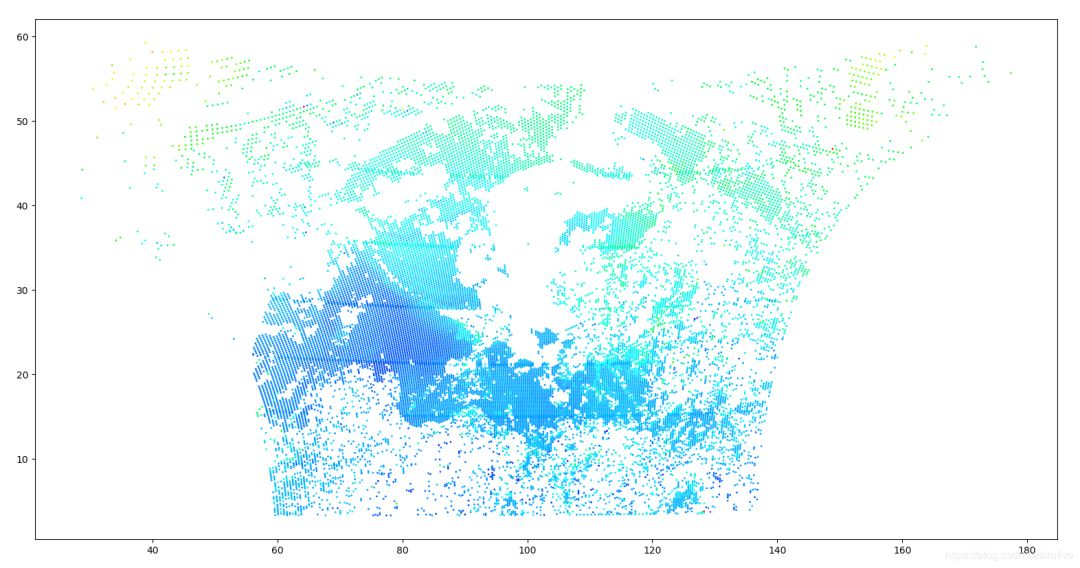
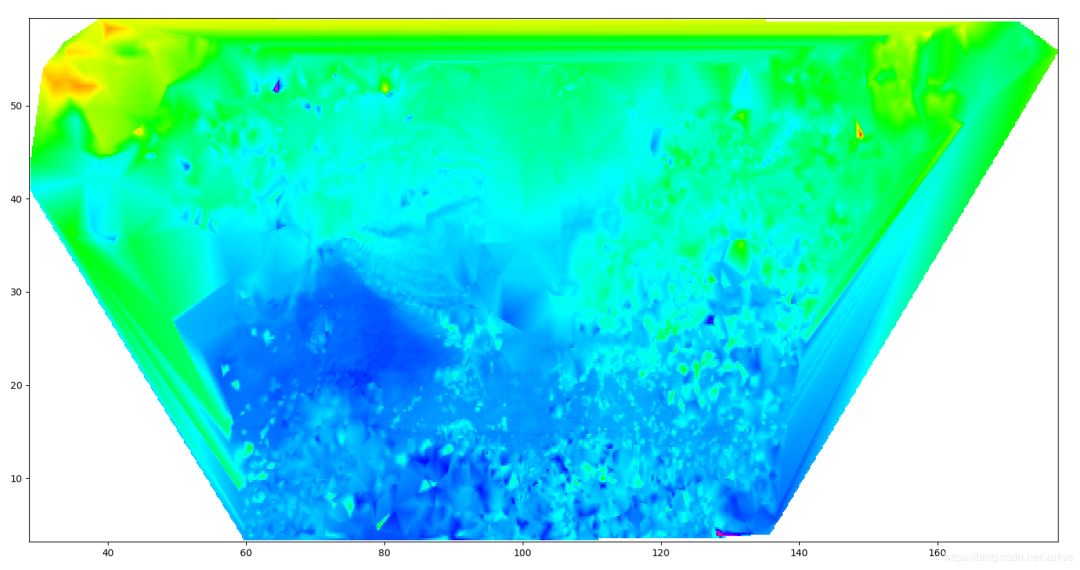
客户要求将整个区域网格化,并将散列数据对应到临近的格点上,网格精度为0.2°(经度纬度相同)。最初,我们使用 scipy 的插值模块 interpolate 提供的格点插值函数 griddata 来实现,但效果不符合客户要求,且速度也慢得无法接受。griddata 的method 参数有3个选项:临近点、线性、三阶样条,下图是 griddata 分别使用线性插值(linear)和三阶样条插值(cubic)实现的效果(临近点插值效果更差)。
import numpy as np> from scipy.interpolate import griddata> import matplotlib.pyplot as plt> lats, lons = np.mgrid[3.2:59.6:0.2, 28.6:177.6:0.2]> z = griddata((lon,lat), tmp, (lons, lats), method='linear') # 可以替换为‘cubic’> plt.pcolor(lons, lats, z, cmap=plt.cm.hsv)> plt.show()

尝试了各种手段,均以失败告终。不得已,只好用c代码写了一个dll,再用 python 调用,虽然代码结构复杂了不少,好在效果、速度都可以满足用户需求。之后的几年中,很多项目里面,凡是需要散列数据网格化的地方,无一例外,都调用了这个动态连接库。
近期又遇到了同样的需求,正好时间也比较宽裕,就花了几天的心思,终于用 numpy 实现了散列数据的网格化。还是以刚才的数据集为例,完整代码如下:
> lats, lons = np.mgrid[3.2:59.6:0.2, 28.6:177.6:0.2]> lon_1 = np.floor((lon-28.6)/0.2).astype(np.int)> lat_1 = np.floor((lat-3.2)/0.2).astype(np.int)> z = np.empty(lons.shape)> z.fill(np.nan)> z[(lat_1, lon_1)] = tmp
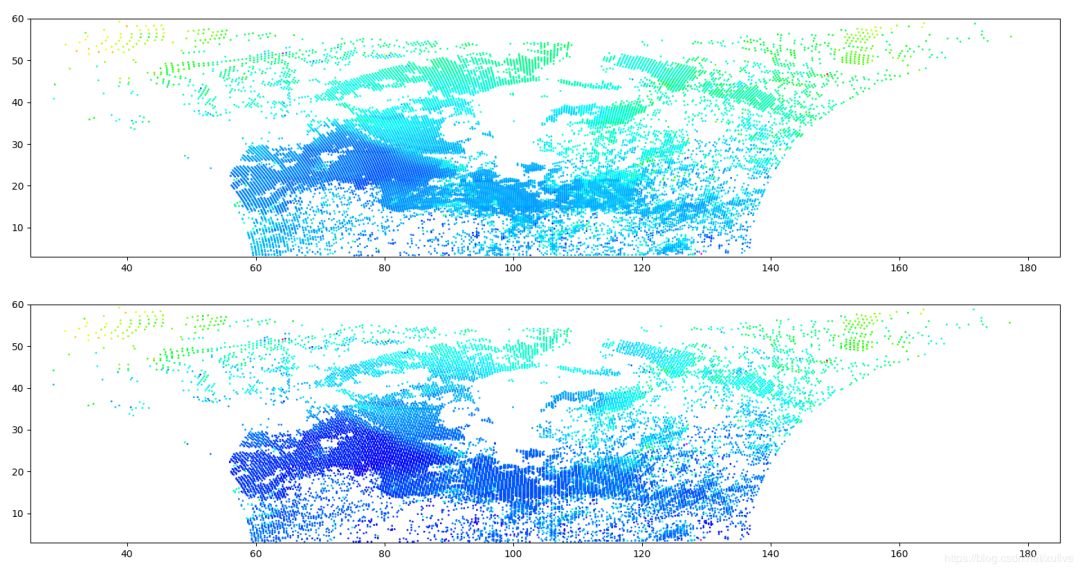
够简单吧?重要的是,速度快到匪夷所思的地步!我把散列数据和格点数据用用样的方法画在一张图上,效果如下:
> plt.subplot(211)> plt.scatter(lon, lat, s=1, c=tmp, cmap=plt.cm.hsv)> plt.axis([25, 185, 3, 60])> plt.subplot(212)> plt.scatter(lons.ravel(), lats.ravel(), s=1, c=z.ravel(), cmap=plt.cm.hsv)> plt.axis([25, 185, 3, 60])> plt.show()
让我知道你“在看”
评论
