为什么Alpha多样性的输入数据会是它?
生物信息学习的正确姿势
NGS系列文章包括NGS基础、在线绘图、转录组分析 (Nature重磅综述|关于RNA-seq你想知道的全在这)、ChIP-seq分析 (ChIP-seq基本分析流程)、单细胞测序分析 (重磅综述:三万字长文读懂单细胞RNA测序分析的最佳实践教程)、DNA甲基化分析、重测序分析、GEO数据挖掘(典型医学设计实验GEO数据分析 (step-by-step))、批次效应处理等内容。
在扩增子培训过程中,总会被问到一个问题:抽平后的OTU表和计算相对丰度后的OTU表差别是什么?
抽平和计算相对丰度都是对OTU表进行标准化的方式,而抽平后的OTU表一般就会用于计算Alpha多样性,两者都可用于计算样本间的差异OTU。但是为什么不统一使用抽平后的OTU表或计算相对丰度的OTU表呢?
我们在扩增子培训中学到了两种以上的方式计算Alpha多样性,比如用vegan包去计算6种Alpha多样性、usearch计算14种Alpha多样性等。下面我们可以尝试使用抽平后的OTU表(otutab_rare.txt)和计算相对丰度后的OTU表(otutab.freq.txt)分别来计算Alpha多样性。
方法一:vegan包计算Alpha多样性
suppressWarnings(suppressMessages(library(vegan)))
otu_rare=read.table("otutab_rare.txt", header=T, sep="\t", quote = "", row.names=1, comment.char="")
otu_freq=read.table("otutab.freq.txt", header=T, sep="\t", quote = "", row.names=1, comment.char="")
# 计算抽平后的OTU表的richness, chao1和ACE指数
estimateR_rare = t(estimateR(t(otu_rare)))[,c(1,2,4)]
head(estimateR_rare)
## S.obs S.chao1 S.ACE
## KO1 2148 2442.130 2442.174
## KO2 2097 2376.372 2387.770
## KO3 1792 2169.230 2147.837
## KO4 1853 2174.955 2199.956
## KO5 1657 2061.449 2091.146
## KO6 1976 2303.736 2294.931
# 计算相对丰度后的OTU表的richness, chao1和ACE指数
## 由于代码运行报错所以就注释掉了,报错内容在注释中进行展示
#estimateR_freq = t(estimateR(t(otu_freq)))[,c(1,2,4)]
## Error in estimateR.default(newX[, i], ...) : function accepts only integers (counts)方法二:usearch计算Alpha多样性
# input rare table to calculate the alpha diversty
usearch -alpha_div otutab_rare.txt -output rare.alpha.txt
## usearch v10.0.240_win32, 2.0Gb RAM (8.4Gb total), 8 cores
## (C) Copyright 2013-17 Robert C. Edgar, all rights reserved.
## http://drive5.com/usearch
##
## License: woodcorpse@163.com
##
## 00:00 4.5Mb 0.1% Reading otutab_rare.txt00:00 5.2Mb 100.0% Reading otutab_rare.txt
# input otu rare table to calculate the alpha diversty
#usearch -alpha_div otutab.freq.txt -output freq.alpha.txt
## ---Fatal error---
##alphadiv.cpp(192) assert failed: m_TotalCount > 0可以看到当输入数据是抽平后的OTU表(otutab_rare.txt)时,无论用哪种方法计算Alpha多样性都没有问题,而输入数据是计算相对丰度后的OTU表(otutab.freq.txt)时,代码纷纷报错。
由于usearch并未开源,所以我们主要来看vegan的estimateR函数的报错原因function accepts only integers (counts),表明该函数只接受整数,并且函数的文档也指明Function estimateR is based on abundances (counts) on single sample site,说明输入数据需要是丰度的counts值,但更具体的原因是什么呢?这就和Alpha多样性的不同指标的计算方式有关了。
estimateR函数计算了三种Alpha多样性指标,分别是Richness、Chao1、ACE。
Richness是最好计算的Alpha多样性指数,其计算方式是
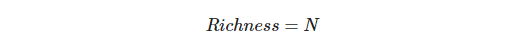
其中N就是该样本的物种数目;
Chao1是常用的Alpha多样性指数之一,其计算方式是
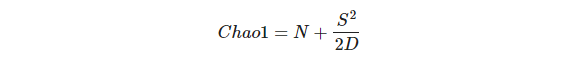
其中N是该样本的物种数目;S是该样本中丰度为1的物种数目;D是该样本中丰度为2的物种数目;
ACE是常用的Alpha多样性指数之一,其计算方式是
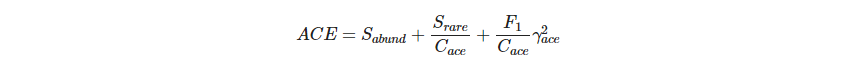

可以看到计算Chao1指数和ACE指数都依据丰度的counts值来计算,而输入计算了相对丰度的OTU表时就丢失了丰度的counts值信息,故而只能使用抽平后的OTU表。
那你还知道哪些基于丰度的counts值计算的Alpha多样性指标或者Alpha多样性其他容易被忽视又很重要的知识点呢?欢迎在评论区讨论。
视频演示Alpha多样性图形绘制
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集