分子结构模拟工具UCSF Chimera的安装及基本操作
UCSF Chimera是一个用于分子结构和相关数据的交互式可视化和分析工具。主要包括:密度图,超分子组合,顺序排列,对接结果,轨迹和构象整合。也可以生成高质量图像和动画。
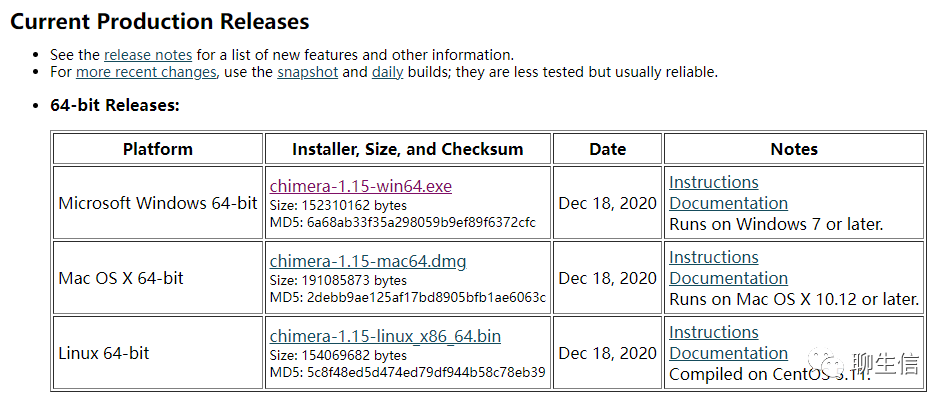
软件下载
基本操作
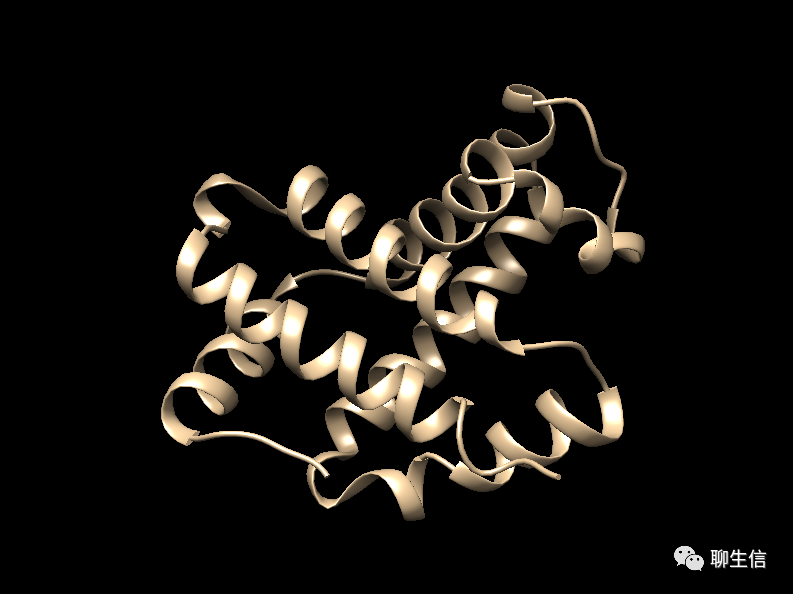
UCSF Chimera的基本操作与PyMOL大致相同,首先准备好一个PDB文件。我们以HBB蛋白为例,使用UCSF Chimera打开,默认结构为ribbon。
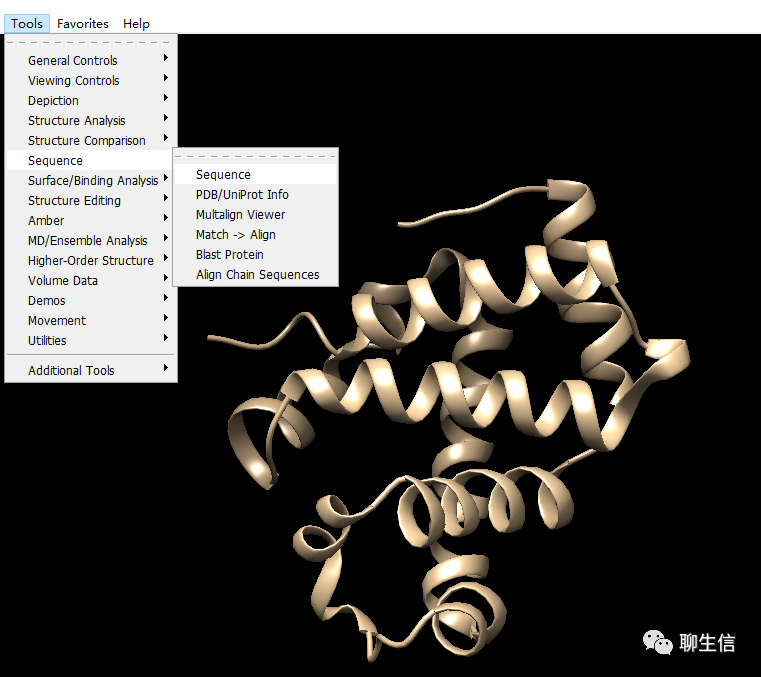

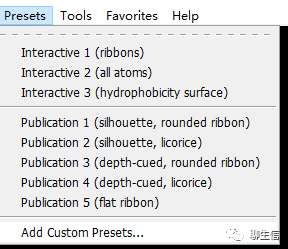
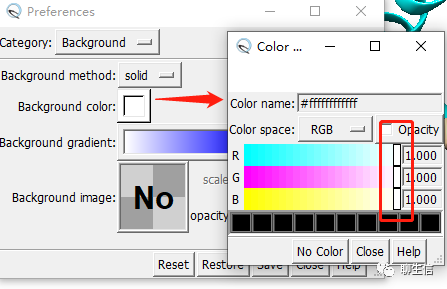
设置背景颜色
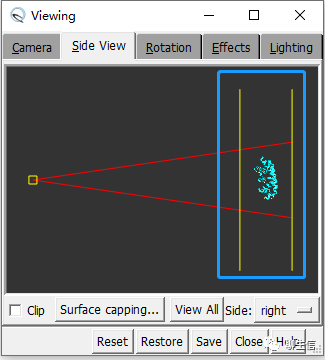
显示/隐藏区域:通过调整蓝框中的两条黄线,显示或隐藏蛋白质的一部分区域
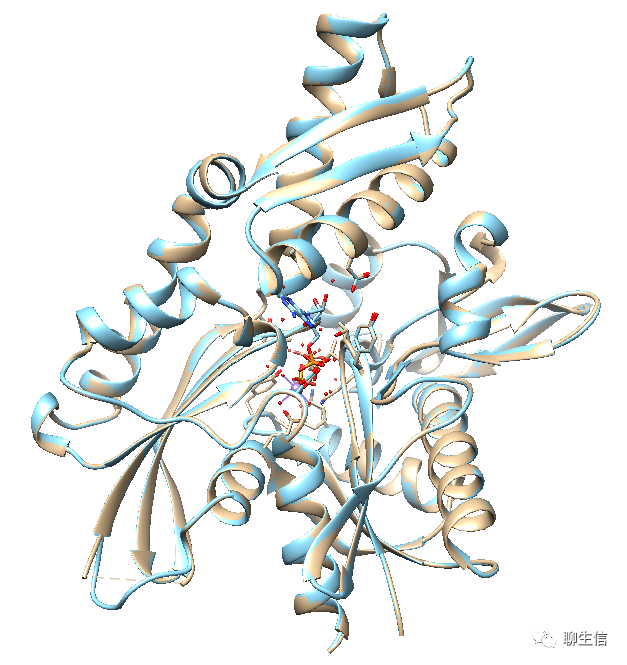
蛋白质结构比对
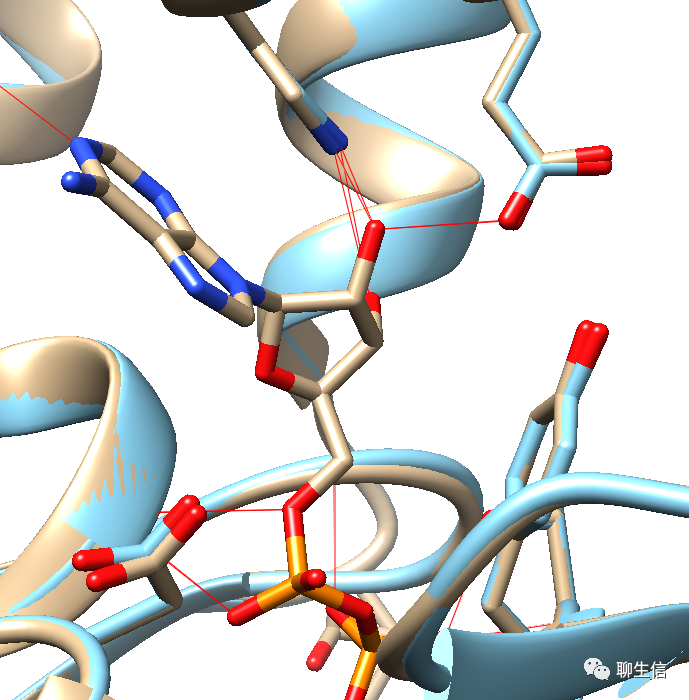
显示氢键
首先我们需要隐藏周围的水分子,点击"Select→Residue→HOH"。按住键盘的“Ctrl”,选择配体中的一个原子,按键盘的“↑”键,选中完整配体,点击“Tools→Structure Analysis→FindHBond,勾选1“Only find H-bonds”,显示配体与周围氨基酸的氢键作用。
测量两个原子之间的距离
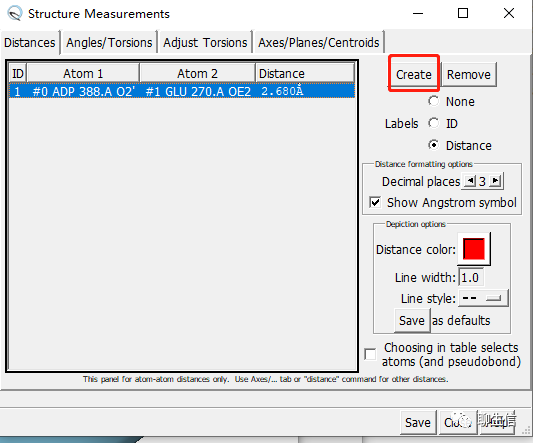
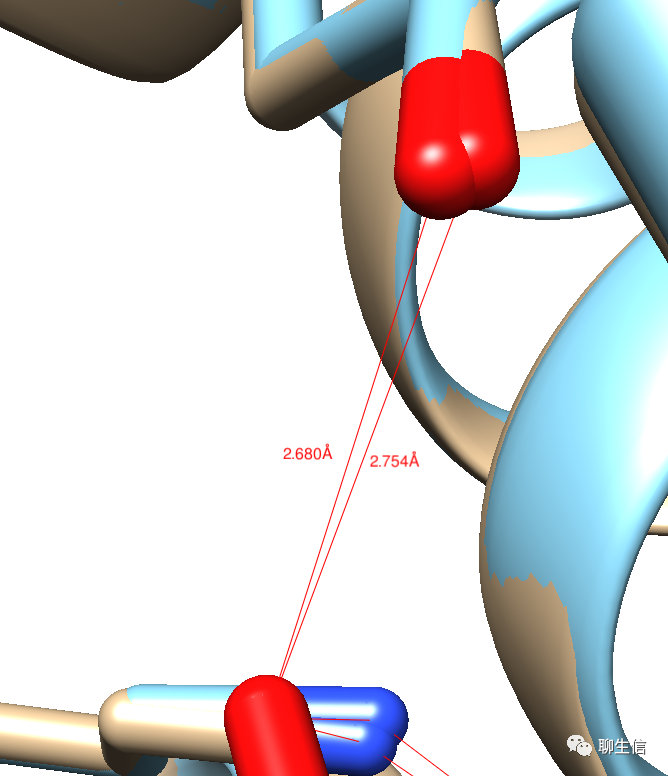
输出图片
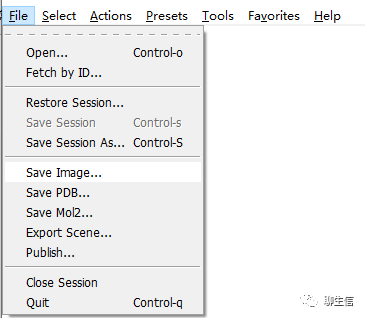
UCSF Chimera下载免费,安装简单,输出图片较为美观。与PyMOL相比操作较为复杂(如选择残基时需要同时操作键盘和鼠标),但UCSF Chimera做点突变时相比 PyMOL功能更强大,可以自动调整点突变后周围其他氨基酸结构的变化。UCSF Chimera的资助于2018年结束,不再处于积极的开发中,只在关键维护时才进行更新。
北京、广州同时开课 | 临床基因组学数据分析实战助力解析Case,快速发表文章
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
评论



























