Nature 子刊 | 超算+AI为天然产物生物合成路线规划提供导航

为了解决这一问题,中山大学药学院巫瑞波教授课题组与国家超级计算广州中心杨跃东教授联合星药科技、麻省理工学院和佐治亚理工学院,基于超算的强大计算能力,提出一种深度学习驱动的生物逆合成路径导航工具BioNavi-NP。该工作近日发表于Nat Commun杂志,论文的共同第一作者为郑双佳、曾涛两位博士生。如图1所示,该框架中,研究者们首先训练一个生物变压器神经网络来进行单步生物逆合成预测,即生成任意目标天然产物的候选前体。在训练好的单步生物反应预测模型的基础上,研究者们进一步开发了一个自动逆向生物合成路线规划系统BioNavi-NP。该系统通过深度学习驱动的基于与或树(AND-OR tree)的搜索算法来解决合成途径潜在大量分支反应造成的组合爆炸问题,从而能够高效且准确地规划出合理的天然产物生物合成途径。给定目标天然产物,BioNavi-NP可在几分钟内给出其到指定分子砌块(building block)的多条路径,并根据已知反应或酶对路径进行打分排序。大量的实验表明,相较于目前最先进的生物合成路径预测方法,BioNavi-NP在内部测试集和外部独立测试集中都表现出了明显的竞争优势。
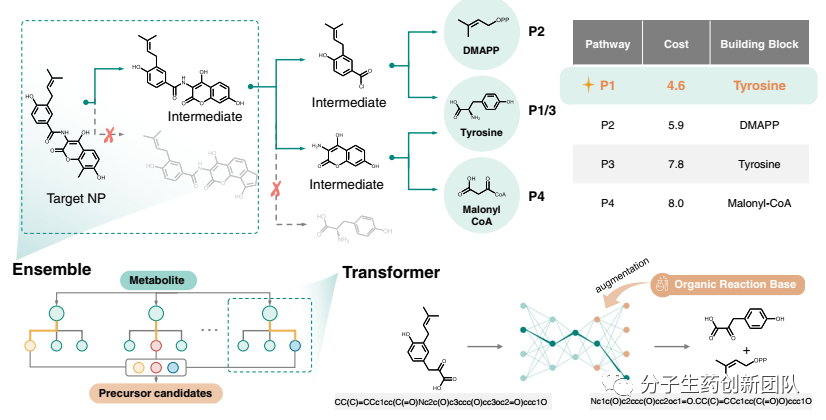
图1. BioNavi-NP模型示意图
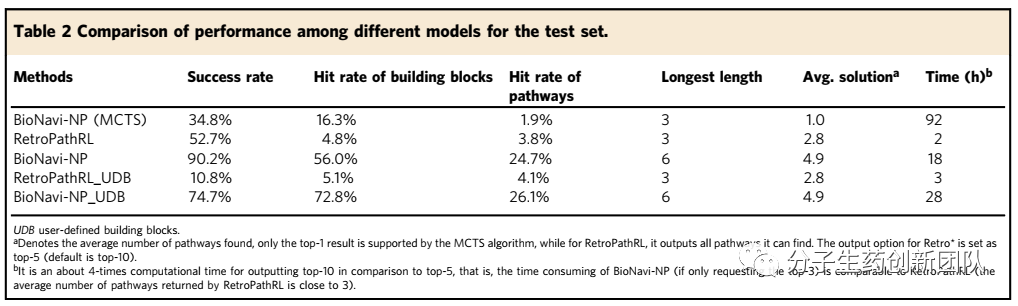
图2. BioNavi-NP在Metacyc天然产物测试集上与最新预测方法的比较结果。
研究者们利用该模型构建了一个用户友好的互动网站(http://biopathnavi.qmclab.com/)。用户仅需要输入感兴趣的天然产物分子,配合基本的参数,便可在数分钟内预测得到其多条可能的生物合成途径。对于多步骤生物反合成路线中的每个生物合成步骤,研究者们可通过网站预先嵌入的酶预测工具Selenzyme和E-zyme进一步评估合成所需要的酶。预测的反应途径将按照计算成本、长度和生物体特定的酶进行分类。
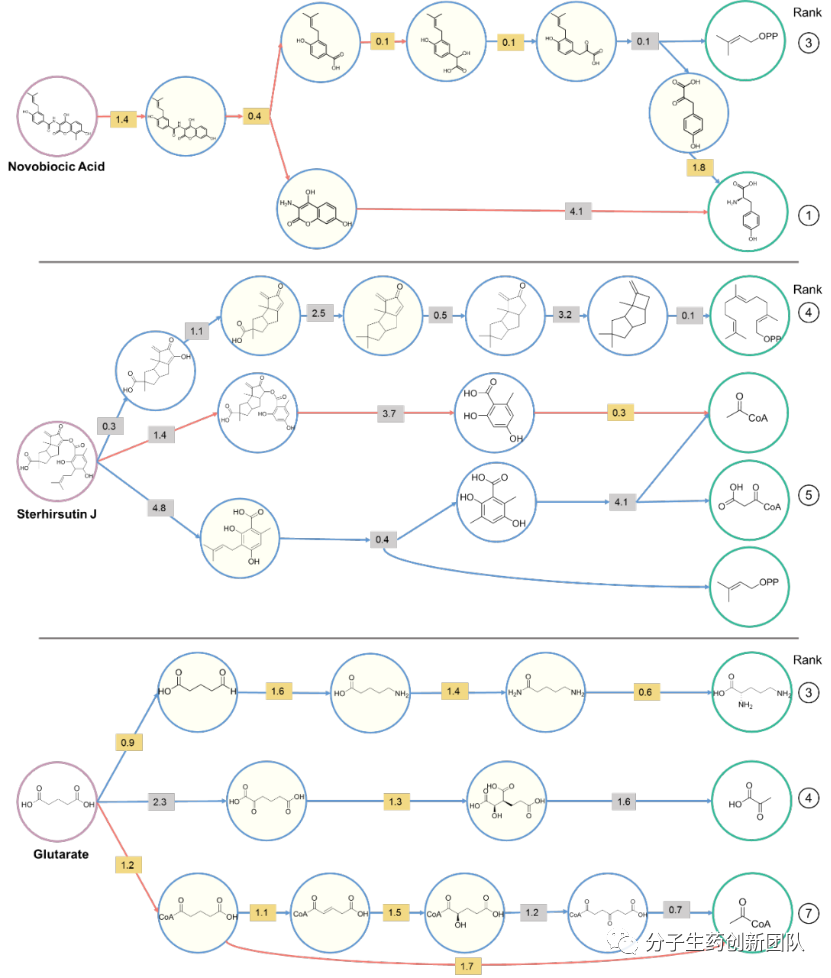
图3:BioNavi-NP网站输出结果示意图
BioNavi-NP网站对目标化合物的预测结果如图3所示,对于来自多种合成途径的复杂分子(如novobiocic acid和sterhirsutin J),BioNavi-NP能够正确对其结构进行拆分并分别找到其对应的分子砌块。在对戊二酸(glutarate)的生物合成途径预测结果中,BioNavi-NP不仅顺利预测出了已知的两条路径(分别排序第三和第七),并且结果中排序第四的路径与不久前Wang等人(ACS Synth. Biol. 2017, 6, 1922-1930)在大肠杆菌构建的新颖路径基本一致(只缺少部分中间步骤)。这说明BioNavi-NP不仅能对天然产物未知的生源途径做出预测,还能对其异源生物合成途径的重构提供重要参考。
本研究提出了一种新颖的端到端深度学习框架BioNavi-NP,利用生物变压神经网络(BioTransformer)及与或树(And-Or-Tree)驱动的深度搜索算法预测天然产物的生物合成途径。在已知的天然产物生物合成途径理论预测工具中,这是第一个无需模板的逆生物合成全路径预测模型。实验评估表明,BioNavi-NP模型在内部测试集及多个外部独立公共测试集上都显示出了优异的性能。网站工具的搭建以及酶预测工具的嵌入,使得研究者们能够便利地使用该工具进行合成生物学的研究。文末,作者对当前BioNavi-NP模型的一些局限性也作了讨论。未来,该框架将嵌入多种路径打分排序方法以及原子映射技术,以进一步提高模型的可用性及可解释性,进而推动天然产物生物合成新途径的发现。
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集