cola,一个做consensus clustering的R包
今天和大家分享是我这个月发表的一个Bioconductor工具,叫做cola。它提供一个普遍的框架,用来做consensus clustering。Bioconductor链接为https://bioconductor.org/packages/cola/,论文链接为https://doi.org/10.1093/nar/gkaa1146。
在cola框架中,consensus clustering被标准化为若干个步骤,其中某些关键步骤中,用户可以自定义自己的方法。如下图所示:
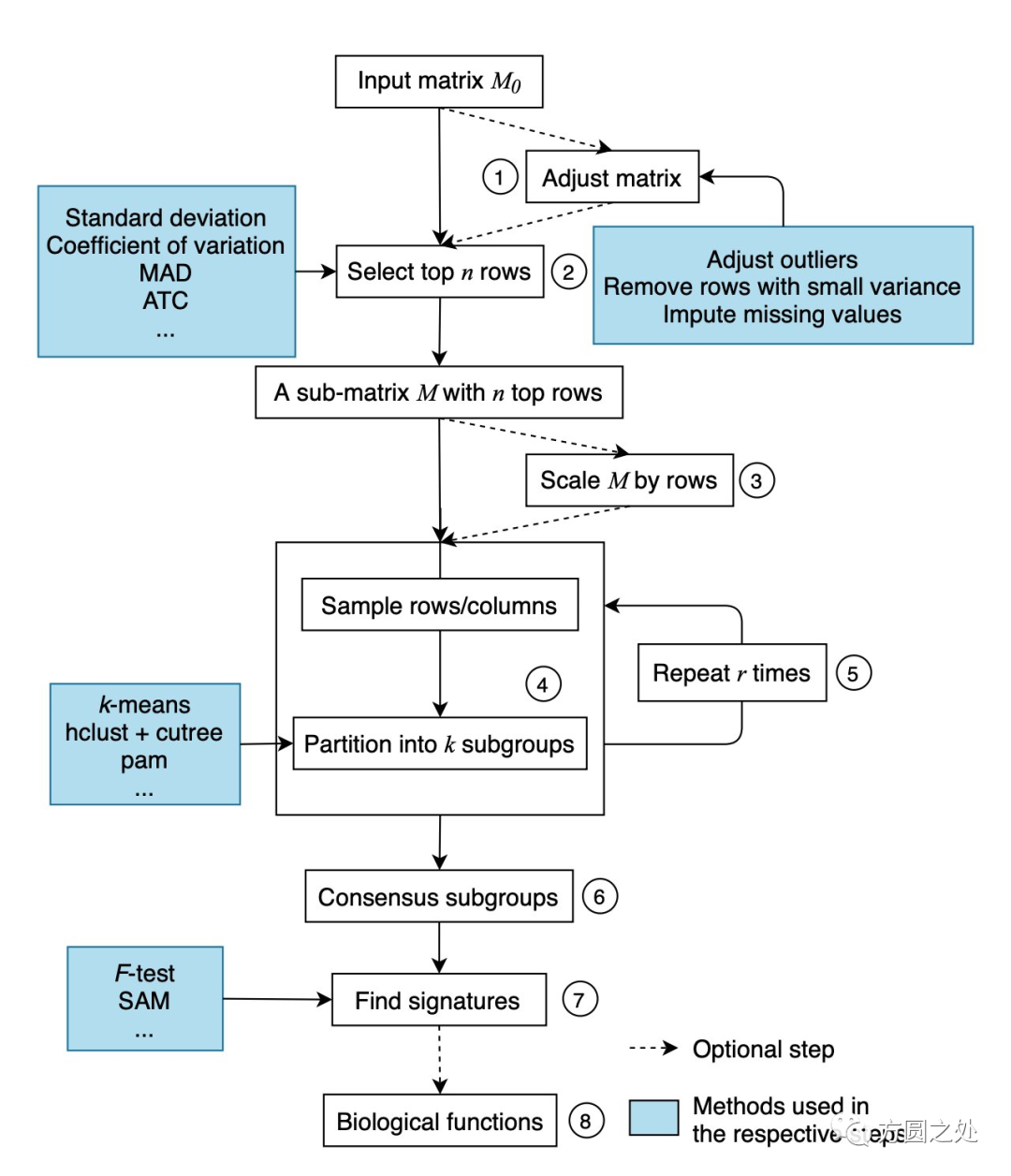
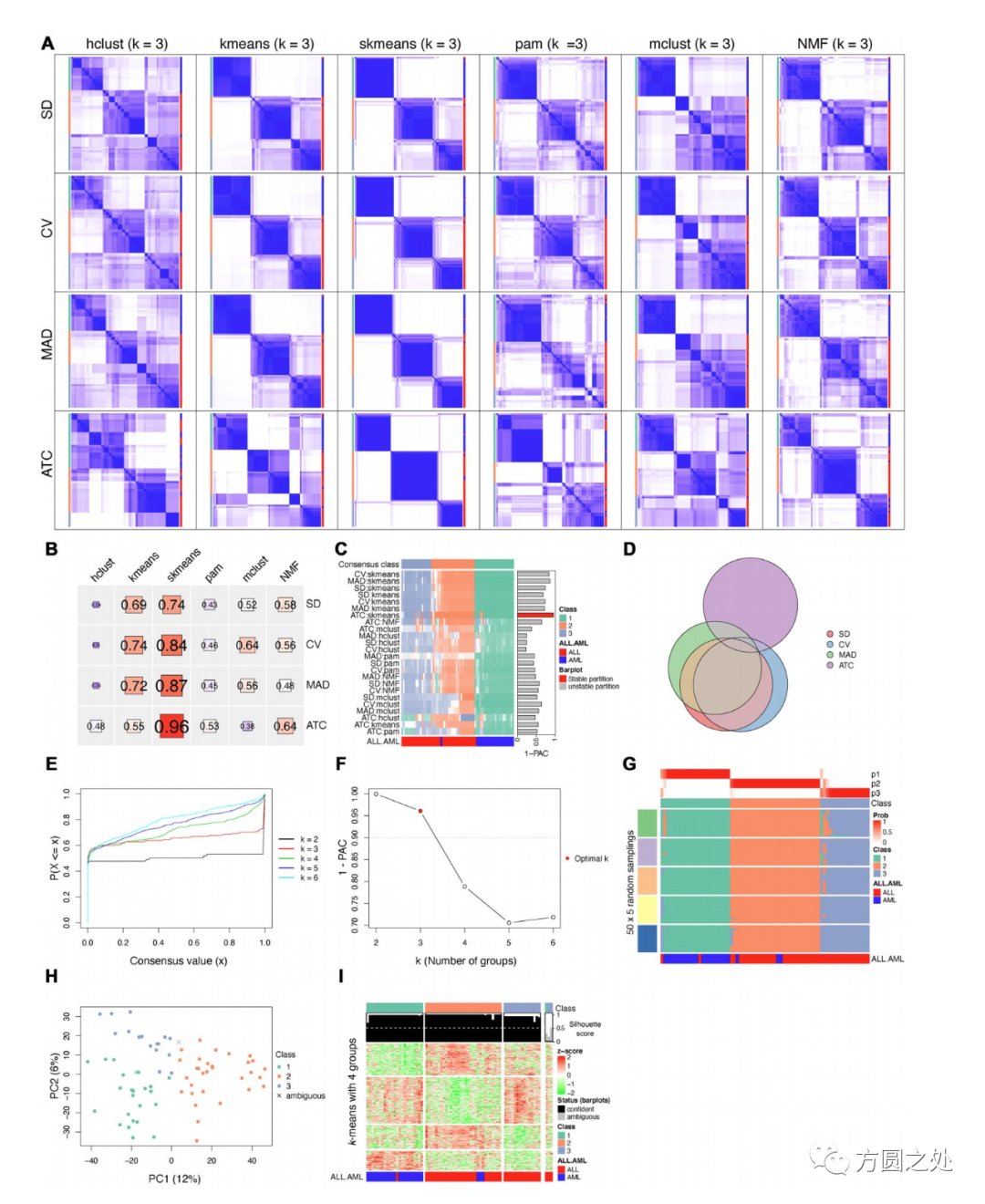
我提出了一个简单但是有效的方法,称作为ATC方法,用来提取有用的feature,用以clustering。这个ATC方法是基于输入矩阵的全局相关系。你可以看到在下图中(第一行的四个热图),四个不同方法所提取的top features,ATC方法提取的top feature能够更有效的生成稳定的clustering(第四个热图)。

我同样提出建议使用shperical k-means clustering (skmeans)对ATC所提取的features进行聚类,我展示了skmeans一般来说能够揭示更多的subgroups,并且分类具有更高的稳定性。
在论文中,我使用了超过400个公共数据集对不同的聚类方法进行了比较。我也对consesus clustering中的关键参数的选择进行了系统性的比较,例如是对行进行随机抽取还是对列进行随机抽取,和随机抽取的次数对结果的影响。
在论文中,我应用cola在基因表达数据和DNA甲基化数据上。结果建议对不同类型的数据应该选择不同的参数和方法,不能宽泛的使用相同的数据(例如缺省的参数)。
cola包支持同时运行多个聚类方法,并且cola提供了大量的函数用以对结果进行比较。下图是cola所生成一些图:
cola提供了强大的函数集,但是同时也提供了一个简单易用的用户接口。也就是说,在大多数情况下,用户只需要执行下面两行代码,然后cola会自动运行所有分析,并且将所有结果生成到一个HTML报告中,并且这个HTML报告中也包含了代码,是完全可以reproducable的。
rl = run all consensus partition methods(matrix, ...)
cola report(rl, ...)
最后,欢迎大家使用!
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
(请备注姓名-学校/企业-职务等)



























