前文回顾
1. GATK官方教程 / 概述及工作前的布置
2. GATK教程 / 体细胞短变异检测 (SNV+InDel)流程概览
3. GATK教程 / 变异检测前的数据预处理
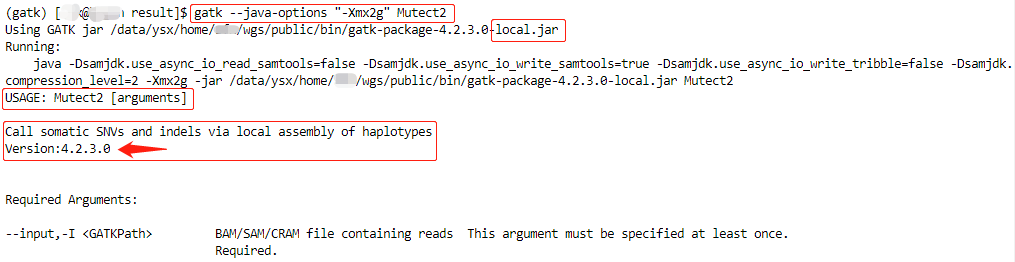
(如何) 使用GATK4 Mutect2调用体细胞突变
(How to) Call somatic mutations using GATK4 Mutect2· 本教程适用于Mutect2版本4.1.1.0,及更高版本,版本确认:· 建议和问题反馈:https://gatk.broadinstitute.org/hc/en-us/community/topics GATK (v4.1.1.0)包含了Mutect2管道的几个主要进步,这是好的,此新版Mutect2不得不在一些地方更改命令行,尽管是开发者们通常尽量避免的。以下内容是主要的改动(也包含主要的分析流程):
错误修复(Bug Fixes)
修复了几个错误,即关于以下内容的报错信息:无效的对数概率/Invalid log probabilities、无穷大/Infinities、NA值/NaNs。这些错误是由于忽略了有限精度(Finite precision)错误而引入的。 新版还解决了CalculateContamination在非常小的基因面板/Panels上工作不佳的问题。
FilterMutectCalls的新的必需输入
1) FilterMutectCalls现在需要一个参考基因组,它应该是与Mutect2相同的fasta文件输入。
2) FilterMutectCalls还需要一个来自Mutect2的stats文件。例如,当运行Mutect2时输出-O unfiltered.vcf,则会生成一个名为unfiltered.vcf.stats的文件。FilterMutectCalls会自动查找这个文件,所以如果你在本地运行,不需要更改。
也就是说:
gatk Mutect2 -R ref.fasta -I tumor.bam -O unfiltered.vcf 紧接着,是Mutect算法过滤/FilterMutectCalls,生成:filtered.vcfgatk FilterMutectCalls -R ref.fasta -V unfiltered.vcf -O filtered.vcf 因为Mutect2在后台创建了unfiltered.vcf.stats,并且FilterMutectCalls也知道去寻找它。 然而,如果在集群或云(Cluster/Cloud)上运行,则需要跟踪stats文件。例如,你需要将它从VM中去本地化(Delocalize),就像在Mutect2 WDL中所做的那样。你可以在FilterMutectCalls中用-stats参数显式地输入stats文件。 如果你将Mutect2分散到多个节点上(Scattering Mutect2 over multiple nodes),则必须用MergeMutectStats合并stats文件: -stats unfiltered1.vcf.stats \
-stats unfiltered2.vcf.stats \
-O merged.stats
而后将merged.stats文件提交给FilterMutectCalls。
FilterMutectCalls中的新的过滤策略
FilterMutectCalls现在基于单个数值进行过滤,此数值即一个变异是体细胞突变的概率。
以前,“为什么这可能不是这种情况”的每个不同的原因,都有自己的阈值(Previously, different reasons why this might not be the case each had their own thresholds)。但目前已经删除了一些参数,如:-normal-artifact-lod, -max-germline-posterior, -max-strand-artifact-probability, 及-max-contamination-probability。甚至之前最基本的-tumor-lod也消失了。FilterMutectCalls并没有用一个错误概率阈值来代替它们,而是将它们全部替换为“空”。FilterMutectCalls将自动决定优化“F score”(即:灵敏度和精度的调和平均值)的阈值。
此阈值的用法:① 为了调整结果以支持更高的灵敏度,用户可以将-f-score-beta设置为大于其默认值1的值(beta是调和平均值中灵敏度versus/vs.精度的相对权重);② 将其设置地更低,会使结果偏向更高的精度。 你可以把这些变化,看作是完成了两件事:① 过滤阈值的统一,会将所有过滤器置于灵敏度vs.精度的ROC曲线上的同一位置。之前的方法是,一个阈值可能会牺牲大量的灵敏度,来获得精度上的、较小的增加;而另一个阈值可能会做相反的事,结果是:灵敏度和精度都很低,低于潜在的ROC曲线。② 一旦进入ROC曲线,我们就可以通过优化F分数(即“F score”),在灵敏度和精确度之间实现良好的平衡。FilterMutectCalls中的新的体细胞聚类模型(New Somatic Clustering Model)
长期以来,该程序的开发者一直怀疑对亚克隆等位基因分数的频谱进行建模(Modeling the spectrum of subclonal allele fractions),将有助于从错误中区分体细胞变异。例如,如果肿瘤中的每1个体细胞变异都是在40%的细胞中发生的“杂合(Het)”,我们就会知道拒绝任何等位基因分数显著不同于20%的情形(Reject anything with an allele fraction significantly different from 20%)(此处不是40%可能是因为人类是二倍体生物,此例中也说明了是“Het”)。 在Mutect2的贝叶斯框架(Bayesian framework)中,这意味着体细胞变异的可能性,是由二项式分布给出的。开发者用狄利克雷过程二项混合模型(Dirichlet process binomial mixture model)来解释未知数量的亚克隆。这仍然是一种过度简化(Oversimplification),因为(此时)CNVs、小亚克隆和乘客突变的遗传漂变(Genetic drift of passenger mutations)均会产生一些与离散值不匹配的等位基因分数。因此,开发者进一步在混合模型中加入了少数几个β二项式,来解释等位基因分数的背景扩散,且仍受益于聚类。
最后,开发者使用这些二项式和β二项式可能性(Binomial and beta-binomial likelihoods),来改进由Mutect2计算的肿瘤对数几率(Tumor log odds),其中Mutect2假设等位基因分数具有均匀分布(Uniform distribution)。
除了聚类等位基因分数外,Mutect2还学习了体细胞SNV和InDel的整体先验概率(Overall prior probabilities),这样就可以对静态肿瘤(Quiet tumor)中的明显变异,更加怀疑(Skeptical)。开发者用随机EM(Stochastic EM)方法,学习该模型的参数,其中E步骤(E steps)包括对等位基因分数簇的中式餐馆过程采样(Chinese Restaurant Process sampling of the allele fraction clusters)。如果你想知道的话,开发者已在Mutect2的一些去标签及非癌症用途(Off-label, non-cancer uses,例如线粒体/Mitochondria)上测试了这种新方法,效果非常好。
FilterMutectCalls在一个新的.filtering_stats.tsv中报告了体细胞聚类的学习参数。这个文件还包含了为优化F分数而选择的概率阈值,以及期望从该选择中获得的、每个过滤器的假阳性和假阴性数值。
新的Mutect2读定向工作流指南(A step-by-step guide to the new Mutect2 Read Orientation Artifacts Workflow)
在测序前(即不是测序仪产生的错误),如果你怀疑你有任何的、发生在单链上的(碱基)替换错误(Substitution errors)的样本,你绝对应该使用Mutect2的定向偏差过滤器(Orientation bias filter)。这适用于所有FFPE肿瘤样本,以及在Illumina Novaseq上测序的样本。
福尔马林固定石蜡包埋(Formalin-Fixed Paraffin-Embedded,FFPE)组织样本,临床上常用,适合做IHC、组化或基因组原位分析;特点是常温下也很稳定,但长时间保存后,DNA也会碎片化或降解。此外,常温下FFPE可能产生微量的SNV/InDel随机突变,需高深度测序,如>80X,纠正之,或存在偶发的尿嘧啶损伤,需在DNA提取与扩增时用尿嘧啶-DNA糖基化酶处理,以减少假突变。参考:https://www.mybeckman.cn/resources/technologies/next-generation-sequencing/challenges-with-ffpe-tissue-samples
Panel of Normals (PON)
Panel of Normal(PON)是一种用于体细胞变异分析的资源。根据你要寻找的变体类型,PON的生成方式有所不同。所有PON的共同点是:① 它们由正常样本制成(在本文中,“正常/Normal”是指来自被认为没有任何体细胞改变的健康组织);② 它们的主要目的是捕获反复出现的技术工件(Technical artifacts),以改进变异调用(Variant calling)分析的结果。
因此,选择要包含在任何PON中的正常样本,最重要的选择标准是数据生成方式的技术属性。使用在技术上与肿瘤尽可能相似的正常样本非常重要(It's very important to use normals that are as technically similar as possible to the tumor. 相同的外显子组或基因组制备方法、测序技术等)。 此外,正常样本应该来自年轻且健康的受试者,以尽量减少使用来自患有未确诊肿瘤病人的样本作为正常样本的机会。正常样本通常来自血液样本。 因此,正常样本可以不来自肿瘤患者的未受累组织(即“自身配对”),但是需要与肿瘤组织在建库、测序等过程中保持技术上的完全平行。 对于应该使用多少样本来制作PON,没有明确的规则(即使是小型PON,也比没有PON要好),但在实践中,我们建议至少以40个为目标。在Broad研究所,我们通常为给定版本的管道制作标准PON(对应于生产中用于生成序列数据的所有协议的组合,从样品制备开始,包括分析软件),并使用它来处理通过该版本管道的所有肿瘤样本。因为我们以相同的方式处理许多样本,所以我们能够制作由数百个样本组成的PON。下面给出了特定于变异类型的建议。 对于短变异发现,PON是通过在一组正常样本上单独运行变异调用工具Mutect2,并将生成的变体调用与最佳实践文档中定义的一些标准(例如,排除至少在2个正常样本中不存在的任何位点)相结合,来创建的。这将生成一个仅限位点(Sites-only)的VCF文件,可以用作Mutect2的PON。 对于CNV发现,PON是通过在一组正常样本上单独运行初始覆盖率收集工具(Initial coverage collection tools),并结合使用专用PON创建工具(Dedicated PON creation tool)生成的拷贝比率数据(Copy ratio data),来创建的。这将生成一个可用作PON的二进制文件。公共GATK正常样本Panel
公共GATK正常样本Panel可作为GATK资源包(GATK resource bundle)的一部分下载。
gs://gatk-best-practices/somatic-hg38/1000g_pon.hg38.vcf.gzgs://gatk-best-practices/somatic-b37/Mutect2-exome-panel.vcfgs://gatk-best-practices/somatic-b37/Mutect2-WGS-panel-b37.vcfhttps://console.cloud.google.com/storage/browser/gatk-best-practiceshg38人类基因组GATK官方下载地址:
https://console.cloud.google.com/storage/browser/genomics-public-data
有关公共可用资源的更多信息,请参见此处:
https://gatk.broadinstitute.org/hc/en-us/articles/360035890811-Resource-bundle
问题反馈
列出的PON是vcf格式,但GATK拷贝数管道需要hdf5文件。在使用这些文件之前,是否还需要执行其它处理步骤?
我使用Mutect2从MMRF数据中识别体细胞突变,该数据包含来自5个不同种族的多发性骨髓瘤(MM)样本。我有唯一的肿瘤,对应匹配的正常样本是1004 MM左右的WES样本。根据PON文件,PON应该由健康的正常人创建,他们有一个未诊断的肿瘤,而我没有。其次,MM是一种非常异质的疾病,每个种族都有独特的突变特征和克隆进化史。所以,使用PON进行体细胞突变鉴定,还是只依赖肿瘤和匹配的正常样本? PON是否也有助于去除其它胚系变异,这些胚系变异可能因匹配的正常样本的低覆盖率,或缺少匹配的正常样本,而在样本中遗漏?或者你认为以1000genome或dbSNP的vcf的形式提供的胚系资源足以去除胚系变异吗?我这样问是因为我想知道我们是否应该用来自不同种族的正常样本创建一个PON,以有效地去除胚系变异。https://gatk.broadinstitute.org/hc/en-us/articles/360035890631-Panel-of-Normals-PON-#:~:text=A%20Panel%20of%20Normal%20or%20PON%20is%20a,looking%20for%2C%20the%20PON%20will%20be%20generated%20differently.
事实上,通过Mutect2 v4.1.1.0的优化,你甚至可以在无需怀疑的情况下运行过滤器。这不会影响准确性/Accuracy,且现在CPU的成本相当低。
过滤有三个步骤。首先,使用--f1r2-tar-gz参数运行Mutect2,将创建一个带有原始数据(Raw data)的输出,用于学习定向偏差模型。之前这是由CollectF1R2Counts完成的。通过将其引入Mutect2中,消除了CollectF1R2Counts的成本,且对Mutect2的执行时间几乎没有影响。 当指定多个肿瘤样本时,你只需要单个--f1r2-tar-gz输出,它包含每个肿瘤样本的数据。gatk Mutect2 -R ref.fasta \ -L intervals.interval_list \ -germline-resource af-only-gnomad.vcf \ -pon panel_of_normals.vcf \ --f1r2-tar-gz f1r2.tar.gz \ 接下来,将这个原始数据(Raw data)传递给LearnReadOrientationModel:gatk LearnReadOrientationModel -I f1r2.tar.gz -O read-orientation-model.tar.gz 运行GetPileupSummaries来汇总一组已知的变异位点数的读支持(Read support for a set number of known variant sites):
gatk GetPileupSummaries \ -V chr17_small_exac_common_3_grch38.vcf.gz \ -L chr17_small_exac_common_3_grch38.vcf.gz \ -O getpileupsummaries.table 使用CalculateContamination估计污染:gatk CalculateContamination \ -I getpileupsummaries.table \ -tumor-segmentation segments.table \ -O calculatecontamination.table 最后,将学习到的Read定向模型传递给带有-ob-priors参数的FilterMutectCalls:gatk FilterMutectCalls -V unfiltered.vcf \
[--tumor-segmentation segments.table] \ [--contamination-table contamination.table] \ --ob-priors read-orientation-model.tar.gz \ 更需注意:如果要将Mutect2分散在集群或云中的节点上,则必须输入每个Mutect2分散的 --f1r2-tar-gz输出,提交给LearnReadOrientationModel。这是在gatk repo和Terra中的Mutect2 wdl中自动完成的。例如:for chromosome in {1..22}; do
gatk Mutect2 -R ref.fasta -I tumor.bam -L $chromosome --f1r2-tar-gz ${chromosome}-f1r2.tar.gz -O ${chromosome}-unfiltered.vcf all_f1r2_input=`for chromosome in {1..22}; do printf -- "-I ${chromosome}-f1r2.tar.gz "; done` LearnReadOrientationModel $all_f1_r2_input -O read-orientation-model.tar.gz新版Mutect2正常样本Panel工作流程指南(A step-by-step guide to the new Mutect2 Panel of Normals Workflow)
开发者重写了CreateSomaticPanelOfNormals,以使用GenomicsDB来实现可伸缩性。
此外,将INFO字段中FRACTION (带有Artifact的正常样本的分数/Fraction)和BETA (在带有Artifact的样本中的Artifact等位基因分数,其beta分布的形状/Shape参数),添加到正常样本的Panle。 FilterMutectCalls目前还没有使用这些,但希望在不久的将来可以用其进行测试。此外,开发者从不喜欢胚系变异(此类变异是通过种系过滤器/Germline filter以更具原则性/条理性的方式处理)是如何最后变成了被硬过滤的PON位点(Hard-filtered pon sites),所以现在正常样本的Panel工作流,可以选择性地从其输出结果中排除胚系事件(Germline events),(最终)只保留了技术Artifacts。创建一个正常样本的Panel的3个步骤是:
步骤1:对每个正常样本,在只有肿瘤模式(Tumor-only mode)下,运行Mutect2:gatk Mutect2 -R reference.fasta -I normal1.bam --max-mnp-distance 0 -O normal1.vcf.gz gatk Mutect2 -R reference.fasta -I normal2.bam --max-mnp-distance 0 -O normal2.vcf.gz 步骤2:从正常样本的Mutect2调用(Normal Mutect2 calls)中,创建一个GenomicsDB:
gatk GenomicsDBImport -R reference.fasta \ -L intervals.interval_list \
--genomicsdb-workspace-path pon_db \
-V normal1.vcf.gz \
-V normal2.vcf.gz \
-V normal3.vcf.gz
步骤3:使用CreateSomaticPanelOfNormals合并正常调用:gatk CreateSomaticPanelOfNormals -R reference.fasta \ --germline-resource af-only-gnomad.vcf.gz \ 在第三步中,只有af的gnomAD资源是Mutect2使用的相同的公共GATK资源(在调用肿瘤样本时,但在上面的步骤1中没有)。
一些问题反馈
我能否确认这是当前PoN创建步骤中使用的逻辑:任何候选PON位点必须出现在至少2个样本(按照默认的--min-sample-count)。如果该位点不在gnomad中(或在可忽略的频率),它会自动被视为PON包含的候选位点。如果一个位点以不可忽略的频率在gnomad中存在,那么它就会计算它是胚系变异的概率。如果p(胚系)< 0.5,那么它就是候选者。这个0.5是手动可调的通过CMD行参数: --max-germline-probabilityhttps://github.com/broadinstitute/gatk/blob/master/src/main/java/org/broadinstitute/hellbender/tools/walkers/mutect/CreateSomaticPanelOfNormals.java 另外,能否确认一下,这个正常样本Panel是排除了基于染色体位置的变异,不需要等位基因特异性匹配的?例如如果正常样本Panel中该染色体位置有G->T进入,则可以对肿瘤样本中的一个变体进行筛选(去掉)。我看过这样的例子,我想确认一下这就是预期的行为。 我刚刚运行了GenomicsDBImport到我的外显子组样本(17个种系样本)。它已经运行了3天,并创建了一些tmp文件占用1.5 Tb,它仍然在chr4!有什么解决方案吗 当我运行GetPileupSummaries,我得到这个错误:Exception in thread "main" java.lang.OutOfMemoryError: GC overhead limit exceeded at java.util.BitSet.initWords(BitSet.java:166) at java.util.BitSet.<init>(BitSet.java:161) at htsjdk.samtools.GenomicIndexUtil.regionToBins(GenomicIndexUtil.java:164) at htsjdk.samtools.BinningIndexContent.getChunksOverlapping(BinningIndexContent.java:121) at htsjdk.samtools.CachingBAMFileIndex.getSpanOverlapping(CachingBAMFileIndex.java:75) at htsjdk.samtools.BAMFileReader.getFileSpan(BAMFileReader.java:935) at htsjdk.samtools.BAMFileReader.createIndexIterator(BAMFileReader.java:952) at htsjdk.samtools.BAMFileReader.query(BAMFileReader.java:612) at htsjdk.samtools.SamReader$PrimitiveSamReaderToSamReaderAdapter.query(SamReader.java:533) at htsjdk.samtools.SamReader$PrimitiveSamReaderToSamReaderAdapter.queryOverlapping(SamReader.java:405) at org.broadinstitute.hellbender.utils.iterators.SamReaderQueryingIterator.loadNextIterator(SamReaderQueryingIterator.java:125) at org.broadinstitute.hellbender.utils.iterators.SamReaderQueryingIterator.<init>(SamReaderQueryingIterator.java:66) at org.broadinstitute.hellbender.engine.ReadsDataSource.prepareIteratorsForTraversal(ReadsDataSource.java:416) at org.broadinstitute.hellbender.engine.ReadsDataSource.iterator(ReadsDataSource.java:342) at java.lang.Iterable.spliterator(Iterable.java:101) 我也有同样的问题,我发现这基本上与如何从BED文件传递contigs有关。如果BED(或interval_list,因为他们叫它)有很多不同的小间隔在每个contig/chr你正在分析,你会看到在GenomicsDBImport输出的第一行关于那个的警告。解决方案是在GenomicsDBImport命令中插入 --merge-input-interval 你好,我有一个关于CalculateContamination中的“-tumor-segmentation”选项的问题:gatk CalculateContamination \ -I getpileupsummaries.table \ -tumor-segmentation segments.table \ -O calculatecontamination.table - gatk网站上说它“输出表包含按小等位基因分数分割肿瘤” 在上一个版本(https://gatk.broadinstitute.org/hc/en-us/articles/360035889791?id=11136)的教程中没有这样做,下面是命令:gatk CalculateContamination \ -I 7_tumor_getpileupsummaries.table \ -O 8_tumor_calculatecontamination.table -现在“-tumor-segmentation”是一个推荐的选项吗?它如何帮助计算污染?另一个相关的问题,这里的[](方括号)是什么意思?他们是可选的吗? gatk FilterMutectCalls -V unfiltered.vcf \ [--tumor-segmentation segments.table] \ [--contamination-table contamination.table] \ --ob-priors read-orientation-model.tar.gz \ -如果我运行上面的FilterMutectCalls,它会兼顾*交叉样本污染*过滤和*方向偏差*过滤吗? 你是对的。当我添加参数 --merge-input-interval时,它就会产生效果。 gatk什么时候会支持调用复杂的变体,比如EGFR中的变体,不知道在gatk更新了这么多版本之后,这个要求还不满足吗 Panel-of-Normals文件生成,GenomicsDBImport文件合并,我多次得到如下错误信息:[E::bcf_get_info_values] Trying to get 32-bit int data from a field which contains 64 bit values 你好,你能解释一下-tumor-segmentation片段的具体作用吗?表格,以及我们应该如何生成这样的表格? 当我为GenomicsDBImport添加超过2个vcf文件时,我得到了这个错误。- v HDF1_TA1_S4_normal.vcf.gz \- v HDF1_TA2_S5_normal.vcf.gz \- v HDF1_TA3_S6_normal.vcf.gz \- v HDF1_UT1_S1_normal.vcf.gzA USER ERROR has occurred: Duplicate sample: 1. Sample was found in both file:///gpfs/research/medicine/sequencer/NovaSeq/Outputs_fastq/2020_Outputs/Akash_Gunjan_05-19-2020_Yuna-samples/GATK_Bamfiles/Preprocessed_Bam/PON/HDF1_TA2_S5_normal.vcf.gz and HDF1_TA1_S4_normal.vcf.gzFilterMutectCalls需要一个引用参数。但是在上面的命令中省略了这个参数。是故意的还是可以添加的。我无法理解Mutect2(4.1.1.0,或更新)的这个新管道。我的样本是肿瘤,匹配正常组织,WGS。裁判是hg38。组织类型为FFPE。我的理解是这样的(假设我有3对样本)看起来很连线:gatk Mutect2 -R ref.fasta \-L intervals.interval_list \-normal normal_sample_name1-normal normal_sample_name2-normal normal_sample_name3-germline-resource af-only-gnomad.vcf \ -pon panel_of_normals.vcf \ --f1r2-tar-gz f1r2.tar.gz \ -O unfiltered.vcf 我使用这个我使用这个命令生成pon.vcf.gz,"gatk CreateSomaticPanelOfNormals -R /home/dlin/reference/bwa-ref-hg19/Homo_sapiens.GRCh37.75.dna.primary_assembly.fa --germline-resource af-only-gnomad.vcf.gz -V gendb://pon_db -O pon.vcf.gz" 但是我找不到“af-only-gnomad.vcf.”Gz”文件的hg19,谁能告诉我怎么下载这个文件? 我对PoN生成步骤2有一个问题:GenomicsDBImport步骤从Mutect2创建名为VCFs的EMPTY数据库。
https://gatk.broadinstitute.org/hc/en-us/articles/360035531132--How-to-Call-somatic-mutations-using-GATK4-Mutect2
https://gatk.broadinstitute.org/hc/en-us/categories/360002302312
https://gatk.broadinstitute.org/hc/en-us/sections/360007226651-Best-Practices-Workflows
https://gatk.broadinstitute.org/hc/en-us/articles/360036194592-Getting-started-with-GATK4
https://gatk.broadinstitute.org/hc/en-us/articles/360035535912-Data-pre-processing-for-variant-discovery
https://gatk.broadinstitute.org/hc/en-us/articles/360037498992--How-to-Map-reads-to-a-reference-with-alternate-contigs-like-GRCH38
https://gatk.broadinstitute.org/hc/en-us/articles/360035894731-Somatic-short-variant-discovery-SNVs-Indels-
https://gatk.broadinstitute.org/hc/en-us/articles/360035535892-Somatic-copy-number-variant-discovery-CNVs-
https://www.bioinfo-scrounger.com/archives/666/
往期精品(点击图片直达文字对应教程)
机器学习
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集