学习1010种热图绘制方法
转载自Epigenetics表观遗传学,略有修改和补充。
题目有感于德国天才数学家莱布尼兹对二进制的阐述,和其传教士好友布维对易经等汉学的传播。有兴趣的可以阅读莱布尼兹文章英文版:http://www.leibniz-translations.com/binary.htm (二进制和伏羲八卦)
In [3]: a = int('1010', 2)
In [4]: a
Out[4]: 10 想学Python的可以约起
In [5]: bin(10)
Out[5]: '0b1010'
In [7]: bin (16)
Out[7]: '0b10000'什么是 R 语言?R 语言,一种自由软件编程语言与操作环境,主要用于统计分析、绘图、数据挖掘。我之所以学 R 语言,一方面是希望能够利用 R 语言将原始数据转化为可放入论文中的精美图形,另一方面,大数据时代已经到来,每个人都应该懂一些大数据的处理手段,R 语言可以胜任。R 语言下载地址为:https://cran.r-project.org/,通常我们使用页面更加优雅的 RStudio,下载地址为:https://www.rstudio.com/。学习R可参考之前发布的帖子 R语言学习 - 入门环境Rstudio,R语言学习 - 基础概念和矩阵操作和在R中赞扬下努力工作的你,奖励一份CheetShet。
可以把 R 语言理解为一款性能强大的软件,R 里面有很多可处理纷繁复杂任务的包(packages),包里面又有若干执行具体功能的函数(functions),而每一个函数里面又有大量可调节的参数(parameters)。R 语言是开源的,世界各地的开发者们不断地添砖加瓦,分享自己的智慧,截至到目前,R 里面可用的包达到 11987 个,这几乎让 R 语言强大到不可思议。值得注意的是,开发一个 R 包就可以发表一篇不错的文章了。
什么是热图?热图是矩阵中的数值以颜色来显示的图形化表示。热图因其丰富的色彩变化和生动饱满的信息表达被广泛应用于各种大数据分析场景。之前生信宝典出过三篇文章介绍热图的绘制、美化、简化:R语言学习 - 热图绘制 (heatmap), R语言学习 - 热图美化, R语言学习 - 热图简化。另外还有在线绘制工具 最简单漂亮的免费在线生信绘图工具。
R 语言里面可以用来绘制热图的主要包括:
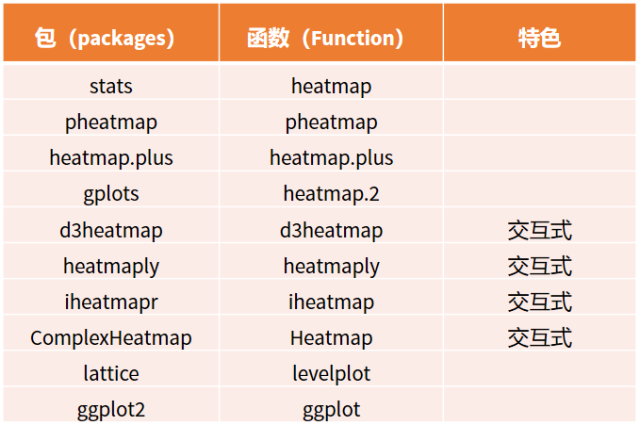
今天将按照这个顺序依次为大家分享它们的绘图方法。
一、基础安装里的 heatmap 函数
所谓基础安装,即下载安装 R 语言后即可使用的包。
heatmap 的使用格式如下:
heatmap(x, Rowv = NULL, Colv = if(symm)"Rowv" else NULL,
distfun = dist, hclustfun = hclust,
reorderfun = function(d, w) reorder(d, w),
add.expr, symm = FALSE, revC = identical(Colv, "Rowv"),
scale = c("row", "column", "none"), na.rm = TRUE,
margins = c(5, 5), ColSideColors, RowSideColors,
cexRow = 0.2 + 1/log10(nr), cexCol = 0.2 + 1/log10(nc),
labRow = NULL, labCol = NULL, main = NULL,
xlab = NULL, ylab = NULL,
keep.dendro = FALSE, verbose = getOption("verbose"), ...)
其中括号中的都是可调整的参数,初步统计了一下,至少包含 23 项参数,但是每一次绘制热图时,其实只需要部分参数即可完成绘图。参数中比较重要的参数是:
x, 需要绘图的矩阵
Rowv 决定“行层级聚类树图”是否以及如何被计算和重新排序,其默认值为空;
Colv 决定“列层级聚类树图”是否或如何被从排序。如果x是一个对称矩阵(行列数相同),那么
Colv=Rowv表示着列与行的处理方式相同。scale = c("row", "column", "none"),按照行或列进行归一化na.rm = TRUE,移除缺失值
另外,以上没有提到的参数是颜色,可用参数 col=。
以下我们通过一个简单的案例来尝试一下。
Case: 将 50 名 NBA 球星的数据绘制成热图
案例来源:http://flowingdata.com/2010/01/21/how-to-make-a-heatmap-a-quick-and-easy-solution/
#首先要获得数据,直接用read.csv导入具有逗号分隔符的表格
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
#R语言会保存一个nba的数据框,键入nba,可查看数据框的内容
nba
#将nba这个数据框按照PTS(points, 分数)由低到高排序,注意nba$PTS后面的逗号是必须的,表示按行排序
nba <- nba[order(nba$PTS),]
#目前导入的数据框默认以数值命名行,重新对行进行命名
row.names(nba) <- nba$Name
#导入第2~20列的数据,并将数据由数据框格式转变为矩阵格式
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
#利用heatmap绘图,按列进行标准化,颜色使用cm.colors函数,使用100种颜色
nba_heatmap <- heatmap(nba_matrix, col = cm.colors(100), scale="column")
注意:(1)#后面一行的代码都不被执行,因此可以用来做标注;(2)在数据整理过程中,如果你好奇数据整理后究竟发生了什么变化,只需要键入相应的数据框或矩阵名,比如“nba”或“nba_matrix”,就可以查看变化;(3)热图绘制时一般输入的是矩阵,而 R 默认的输入格式是数据框,因此需要转化。矩阵和数据框的差异请参照R语言的相关教程。
对于一个热图而言,有三个参数至关重要:1. 用来绘图的矩阵是必须的;2. 热图最令人称赞的就是它绚丽的颜色了,因此颜色参数不可或缺;3. 为了让颜色的区分度更好,需要对数据按照一定的方式进行归一化 (选择什么预处理方式需要慎重,具体看上面的热图美化)。小伙伴们可以自行尝试不进行归一化的糟糕效果。
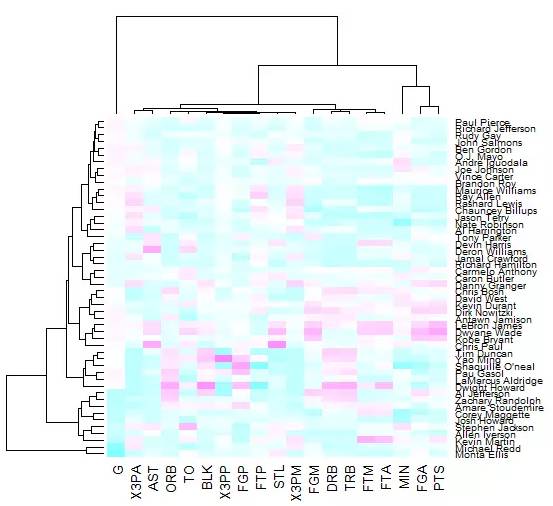
图 1. heatmap 绘制热图
我们发现,在绘图的同时也绘制了层级聚类图,对于 NBA 这一数据,层级聚类图是没有太大意义的,而且颜色其实并不是我们平时所见的红色和黄色。现在我们进行调整
nba_heatmap <- heatmap(nba_matrix, Rowv=NA, Colv=NA,
col = heat.colors(100), scale="column")我们进行了三点调整:1. 将行层级聚类图 Rowv 设置为 NA,即不显示;2. 将列层级聚类图设置为NA,即不显示;3. cm.colors 修改为 heat.colors,即调整了颜色的模式。
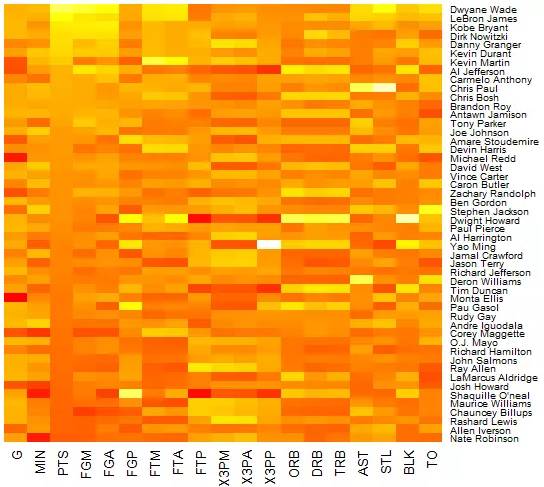
图 2. 调整参数后用 heatmap 函数绘图
二、pheatmap 包里的 pheatmap 函数
pheatmap 实际上是 Pretty Heatmaps 的缩写,新的命名充满了对原始 heatmap 的嫌弃。要使用非基础安装里面的包,就需要安装并加载这个包,代码如下:
install.packages(“pheatmap”) #安装pheatmap包
library(pheatmap) #加载pheatmap包
要查看一个包或者一个包里面函数的详细介绍,代码为:
?pheatmap #查看pheatmap包里面的详细介绍
?pheatmap::pheatmap #查看pheatmap包里pheatmap函数的具体参数
与 heatmap 类似的是,pheatmap 也可以同时绘制热图和系统树图,同样需要矩阵格式的原始输入,需要用 scale 进行标准化,需要颜色;但不同的是,是否需要行或列的系统树图的表达方式不同,前者是Rowv=NULL ,而后者是cluster_rows = TRUE。利用上面的 NBA 数据,我们只是在绘制热图的那一步利用 pheatmap 函数,输入以下代码:
nba_heatmap <- pheatmap(nba_matrix,
cluster_rows = TRUE, cluster_cols = TRUE,
col = heat.colors(100), scale="column")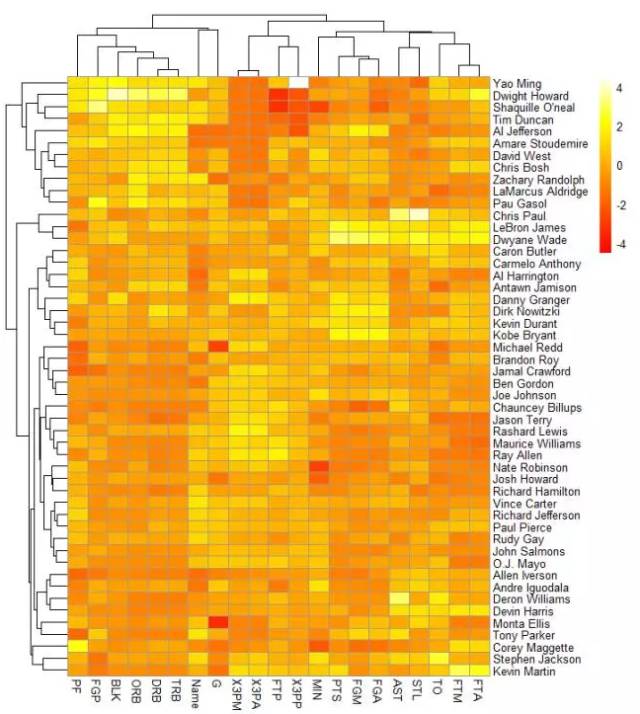
图 3. 用 pheatmap 绘制热图
果然,pheatmap 一出手就不同凡响,信息要比 heatmap 更多。根据这幅图,我们再倒退回去看 pheatmap 函数中的一些关键参数。
热图中的色块以小格子呈现,与小格子相关的默认参数有:border_color = "grey60", cellwidth = NA, cellheight = NA;如果我们不想让每个格子都具有灰色的边框,只需要设置 border_color = NA。
heatmap 函数做的热图非常大的问题在于我们虽然看到了颜色的差异,但是每一种颜色代表的具体数值又是多少呢?我们不得而知。但是 pheatmap 完美地解决了这一问题,看图 3 的右上角,我们可以看到一个颜色和相对数值的对应关系。与这一呈现相关的参数是:legend = TRUE, 默认设置是保留 legend。
如果想获取pheatmap的结果,可以str(nba_heatmap),然后抽丝剥茧的取出每一部分内容。
三、heatmap.plus 包里面的 heatmap.plus 函数
heatmap.plus 与 base 安装里面的 heatmap 很像,只有两个参数 RowSideColors 和 ColSideColors 不一样,在 heatmap 中 两者的输入格式是向量,而在 heatmap.plus 中输入格式是矩阵。关于 R 语言中数据的格式请参照相关书籍。
要理解 RowSideColors 和 ColSideColors 的含义,尝试以下代码:
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
nba <- nba[order(nba$PTS),]
row.names(nba) <- nba$Name
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
rc <- rainbow(nrow(nba_matrix), start = 0, end = .3)
cc <- rainbow(ncol(nba_matrix), start = 0, end = .3)
nba_heatmap <- heatmap(nba_matrix, Rowv=NA, Colv=NA,
col = heat.colors(100),
RowSideColors = rc,
ColSideColors = cc,scale="column")
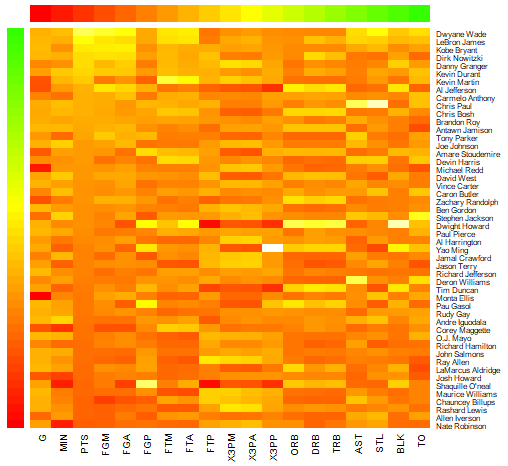
图5. 用 heatmap 绘制带有测边的热图
这里 RowSideColors = rc,ColSideColors = cc,而 rc <- rainbow(nrow(nba_matrix),start = 0, end = .3),cc <- rainbow(ncol(nba_matrix), start = 0, end = .3)。
侧边就是一个颜色梯度条,这里用到 rainbow 函数,其使用格式是 rainbow(n, s = 1, v = 1, start = 0, end = max(1, n - 1)/n, alpha = 1),这里 n= nrow(nba_matrix), 而 s 和 v 分别代表饱和度(Saturation)和纯度(Value),start 和 end 分别为色度(Hue)的起始点。
但是这段代码对 heatmap.plus 是不行的,首先需要将 rc 和 cc 转变为矩阵,试着运行下面这段代码:
rc <- rainbow(nrow(nba_matrix), start = 0, end = .3)
cc <- rainbow(ncol(nba_matrix), start = 0, end = .3)
rc <- matrix(as.character(rc),nrow = 50,ncol = 19)
cc <- matrix(as.character(cc),nrow = 19,ncol = 50)
nba_heatmap <- heatmap.plus(nba_matrix,Rowv=NA, Colv=NA,
col = heat.colors(100),
RowSideColors = rc, ColSideColors = cc,
scale="column")
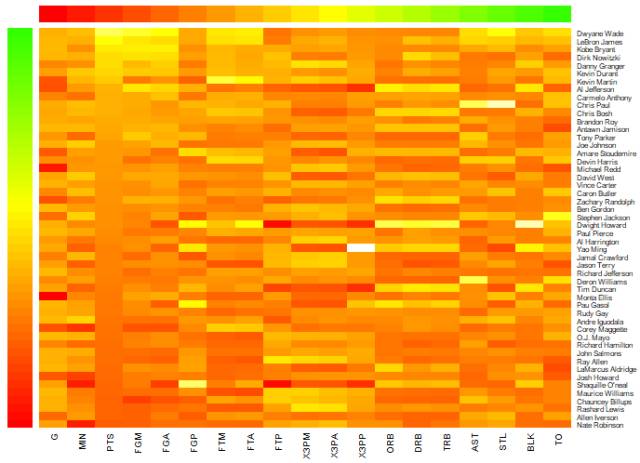
图 6. 用 heatmap.plus 绘制带有测边的热图
pheatmap绘制带有侧边的更合适,具体见 R语言学习 - 热图简化, 最简单漂亮的免费在线生信绘图工具。
四、gplots 包里面的 heatmap.2 包
按照惯例,我们还是试一试下面的代码:
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
nba <- nba[order(nba$PTS),]
row.names(nba) <- nba$Name
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
heatmap.2(nba_matrix, Rowv=NA, Colv=NA, col = heat.colors(100), scale="column")
得到如下图:
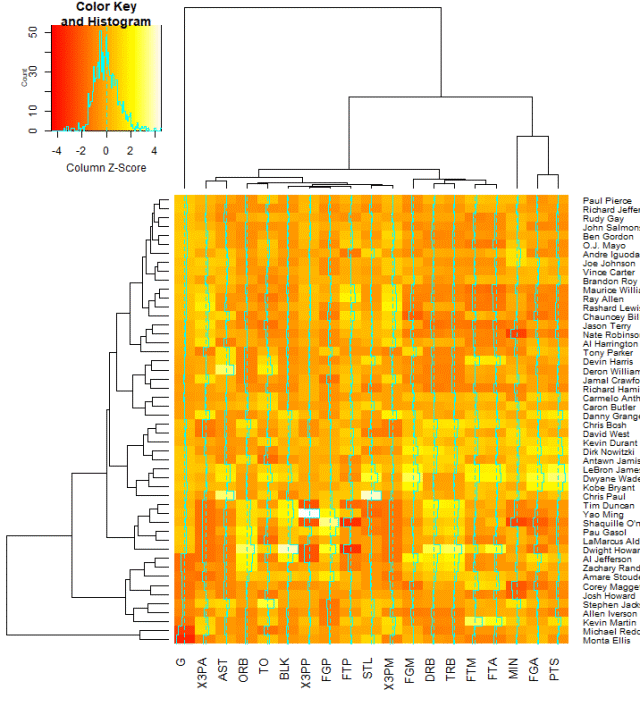
图 7. 用 heatmap.2 绘制的热图
我们发现,图中多了很多绿色的线,这是什么呢?查看说明才知道这是 "trace" line,与之相关的代码为:trace=c("column","row","both","none"), tracecol="cyan",hline=median(breaks),vline=median(breaks),linecol=tracecol。hline 和 vline 分别表示水平和数值的虚线是否需要画出,其默认值均为中位数的转折点。
五、d3heatmap 包中的 d3heatmap 函数
d3heatmap 里面一共包含三个函数:d3heatmap、d3heatmapOutput、renderD3heatmap,后两者是在 shiny 中使用的包裹函数。(shiny 是 RStudio 开发的一款用于构建交互式网页 app 的 R 包,shiny 的构建有点复杂,这里暂且略过,因此只讲解 d3heatmap)
按照惯例,我们还是试一试下面的代码:
d3heatmap(nba_matrix, Rowv=NA, Colv=NA,
col = heat.colors(100), scale="column")
你会得到以下这幅图,这幅图看起来普普通通,但是神奇之处是当你鼠标落到任一色块时,可以显示当前色块的数值。
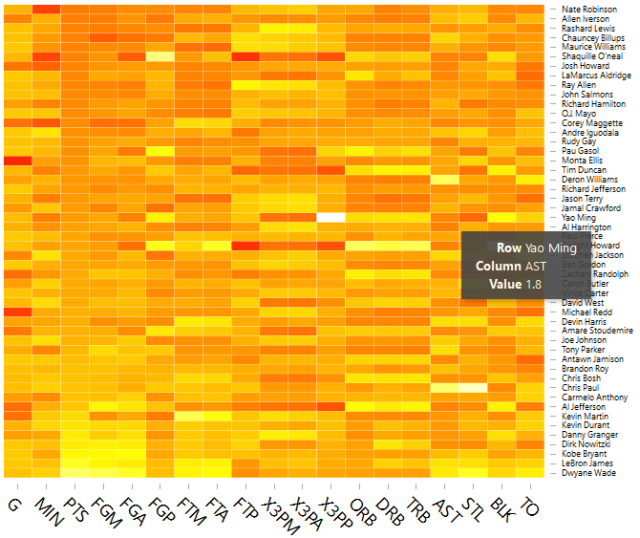
图 8. d3heatmap 绘制的交互式热图
六、heatmaply 包里面的 heatmaply 函数
heatmaply 也是交互式的。尝试以下代码:
install.packages("heatmaply")
library(heatmaply)
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
nba <- nba[order(nba$PTS),]
row.names(nba) <- nba$Name
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
heatmaply(nba_matrix, col = heat.colors(100),
fontsize_row=7, fontsize_col=7, scale="column",
margins = c(50,120,NA,0))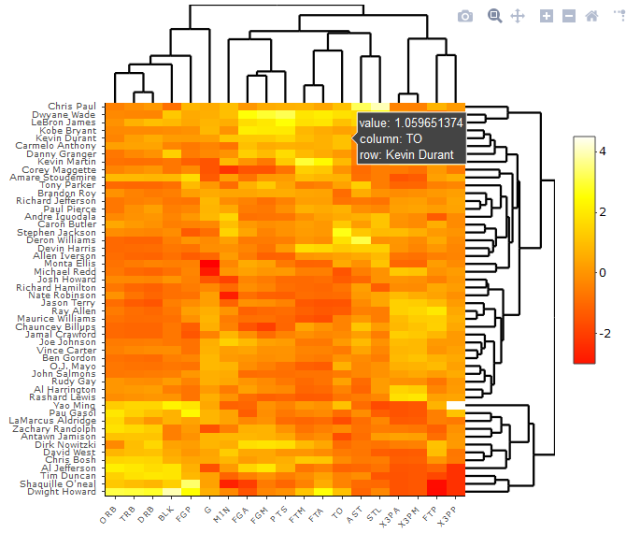
图 9. 用 heatmaply 绘制的热图
这里新出现的 fontsize_row、fontsize_col 和 margins 参数分别表示行标签字体大小、列标签字体大小以及边界(下、左、上、右)。
七、iheatmapr 包里的 iheatmap 函数
iheatmap 绘制的也是交互式的热图,这意味着你用鼠标掠过热图上的色块时,会显示该点的数值。同时,在右上方可以看到一系列可以调整的小图标,这些都是可以调节的参数。
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
nba <- nba[order(nba$PTS),]
row.names(nba) <- nba$Name
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
iheatmap(nba_matrix,colors = heat.colors(100), cluster_rows ="kmeans",
cluster_cols ="hclust",row_k=10,scale="cols")
注意这里的书写方式,已经改为 “colors=heat.colors(100)” 和 scale=”cols”
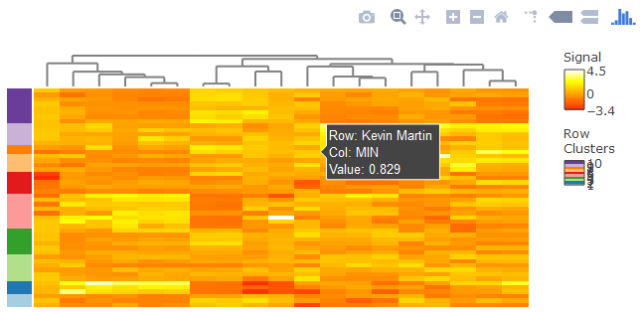
图 10. 用 iheatmap 绘制的热图 (一看就是plotly的效果,ggplot2的图也可以直接plotly)
八、ComplexHeatmap 绘制热图
首先需要特别强调的是,ComplexHeatmap 是 Bioconductor 里面的一个 R 包,安装的方式与一般 CRAN 上的包的安装方式不同,安装和加载代码如下:
source("https://bioconductor.org/biocLite.R")
biocLite("ComplexHeatmap")
library(ComplexHeatmap)ComplexHeatmap 包里面用于绘图的函数也叫做 Heatmap,注意这里的 H 是大写。
具体的参数可用 ?ComplexHeatmap::Heatmap 进行查询,试一下以下这段代码:
Heatmap(nba_matrix, cluster_rows = TRUE,
cluster_columns = TRUE,
col = heat.colors(100))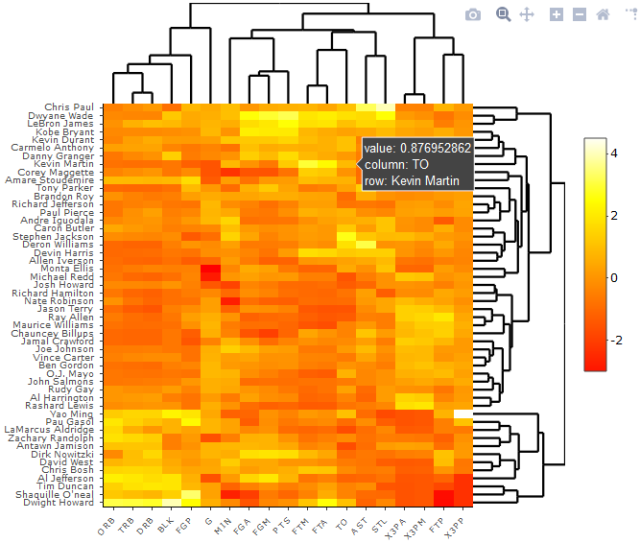
图 11. 用 Heatmap 绘制的热图
这样也是可以得到类似于基础安装里面的 heatmap 的。
关于用 ComplexHeatmap 绘制热图,可以看这一个链接:
http://bioconductor.org/packages/release/bioc/vignettes/ComplexHeatmap/inst/doc/s2.single_heatmap.html
如果阅读英文有困难,可以看这里:http://www.bioinfo-scrounger.com/archives/122
另外,看到 Y 叔(生信领域有名的 Y 叔)关于热图的总结,利用 Y 叔写的 simplot 函数,可用于可视化相似性矩阵。试着对 nba_matrix 做了一下相似性分析,由于数据有 50 行,因此只能把标签和字体尺寸调到非常小,才勉强可以看见。
#安装DOSE
source("https://bioconductor.org/biocLite.R")
biocLite("DOSE")
library(DOSE)
simplot(nba_matrix,labs.size = 1,font.size = 6)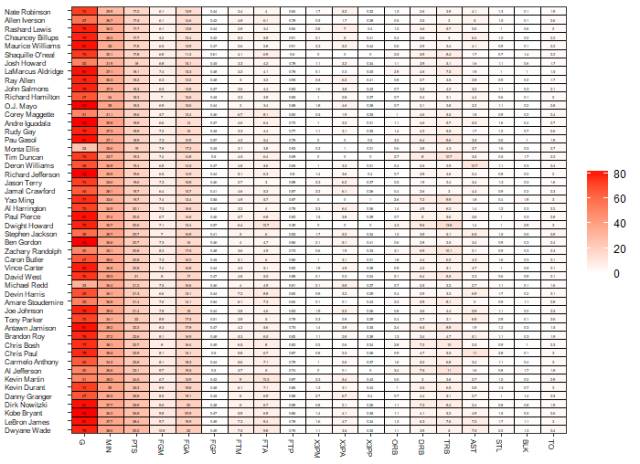
图 12. 用 simplot 进行相似性分析
九、Lattice包里面的 levelplot 函数
Lattice 包是基于 grid 创建的,有自己独特的绘图风格。其中 lattice 包里面的 levelplot 函数可以用来绘制热图。
library("lattice")
library(latticeExtra)
#准备nba_matrix矩阵
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep=",")
nba <- nba[order(nba$PTS),]
row.names(nba) <- nba$Name
nba <- nba[,2:20]
nba_matrix <- data.matrix(nba)
#对参数进行设置
hc=hclust(dist(nba_matrix)) #按行聚类
dd.row=as.dendrogram(hc) #保存行聚类树形
row.ord=order.dendrogram(dd.row) #保存行聚类顺序
hc=hclust(dist(t(nba_matrix))) #按列聚类
dd.col=as.dendrogram(hc) #保存列聚类树形
col.rod=order.dendrogram(dd.col) #保存列聚类顺序
temp1=nba_matrix[row.ord,] #只对行聚类(是否对行、列聚类)
levelplot(t(temp1),aspect="fill",
colorkey=list(space="left",width=1.5),
xlab="",ylab="",
legend=list(right=list(fun=dendrogramGrob,
args=list(x=dd.row,rod=row.ord,side='right',
size=5)),
scales=list(x=list(rot=90))))
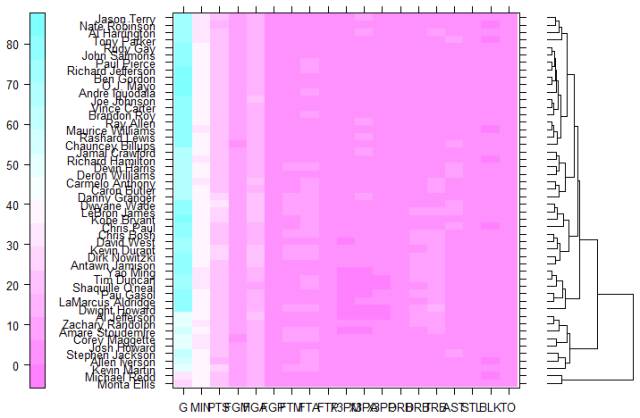
图 13. 用 levelplot 绘制的热图
十、ggplot2 包里面的 ggplot 函数绘制热图
最后,不得不提到十分强大的 ggplot2。ggplot2 中,图是采用串联(+)号创建的,每个函数修改属于自己的部分。下面参考 R-blogger 上的代码,原网页为:https://www.r-bloggers.com/ggplot2-quick-heatmap-plotting/
尝试以下代码:
library(ggplot2)
library(plyr)
require(reshape2)
require(scales)
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
nba$Name <- with(nba, reorder(Name, PTS))
nba.m <- melt(nba) #对数据进行融合
nba.m <- ddply(nba.m, .(variable), transform, rescale = rescale(value))
ggplot(nba.m, aes(variable, Name)) + geom_tile(aes(fill = rescale), colour = "white") + scale_fill_gradient(low = "white", high = "steelblue")
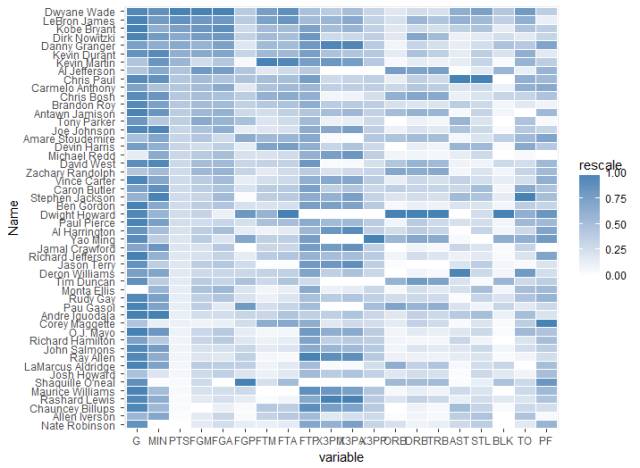
图 14. 用 ggplot 绘制的热图
总结:这篇文章带领我们快速浏览了 R 语言里面的 10 种热图的绘制方式,并且提供了代码。首先介绍了 4 种绘制非交互式热图的包,其次介绍了 4 种绘制交互式热图的包,最后介绍了 lattice 和 ggplot2 绘图系统中绘制热图的方法。但由于笔者的水平十分有限,本次的讲解比较浅显,但显而易见的是,每一种函数提供的热图的绘制参数很多,耐心钻研,一定可以绘制出绚丽的热图。
封面图来源于Wikipedia: https://en.wikipedia.org/wiki/Binary_number
如果看完代码还不会的,可以参加我们的视频课 http://bioinfo.ke.qq.com;或点击原文使用线上版,各种绘图。
精品回顾
好多朋友还没有养成看完转发、点赞的习惯,应该有的!!
