一杯DNA装下全世界? MIT团队突破DNA新检索技术,实现DNA数据「冷存储」!

新智元报道
新智元报道
来源:MIT NEWS
编辑:LQ、小匀
【新智元导读】我们能把数据存储到DNA上吗?目前这项技术的主要瓶颈是,我们很难从所有文件中挑选出想要的特定文件。近日,麻省理工学院开发了一种检索DNA数据文件的新方法,或许能成为DNA存储数据的重要一步。
一个咖啡杯就能装下全世界?
有了DNA数据存储,这是可能的。
1988年,艺术家Joe Davis和哈佛大学研究人员合作,首次证明了DNA存储数字化数据的原理。

Davis通过明暗像素将代表35bits数据的符文符号图像表示为二进制0和1,并将其编码成了大肠杆菌DNA中的28个碱基对。
随后,存储在DNA中的数据也从简单文本变成高清音乐视频、整个数据库、MPEG、JPG、PDF等文件,甚至还有恶意软件。
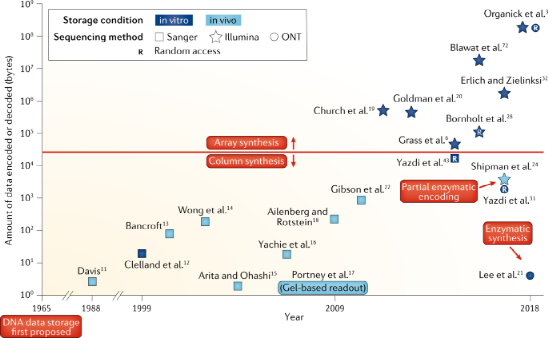
DNA数据存储发展过程(1965-2018)(图源:nature)
DNA数据存储是什么
DNA数据存储是一个将二进制数据转换成人工合成DNA链的编码过程。
为了在DNA中存储二进制数字文件,比特(bits)将从1和0转换成字母A,C,G,T,这四个字母代表组成DNA的四种核苷酸:腺嘌呤,胞嘧啶,鸟嘌呤,胸腺嘧啶。
物理存储介质是一条序列中包含As, Cs, Gs, Ts的合成DNA链,其顺序与数字文件中的bits相对应,如果要恢复数据,需要对DNA链进行测序,根据As, Cs, Gs, Ts还原成初始的数字序列。
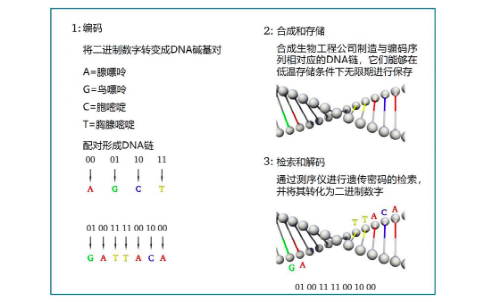
数字化的数据在DNA中编码和解码的过程(图源:https://www.ssbt.org.cn/upload/20190905163302_429.pdf)
在人类创造数据能力不断增长的今天,基于DNA的数据存储似乎是个「潜力股」。
因为与其他大多数媒介相比,DNA提供了惊人的「数据存储密度」,而且相比传统数据存储,它具有高度稳定性:DNA分子半衰期超过「500年」,低温条件下可保存「成千上万年」。
但DNA并非没有缺点,成本高昂是阻碍其发展的主要问题。
目前,DNA链的碱基模式中没有编码比特的标准方法,合成特定的序列仍然很昂贵。而用目前的方法访问数据不仅慢,而且会消耗用于存储的DNA。如果试图访问数据的次数太多,就必须以某种方式恢复它,这有可能引入错误。
近日,麻省理工学院和Broad研究所(Broad Institute)的一个团队找到了一个解决方案。在这个过程中,研究人员创建了一个基于DNA的图像存储系统,它介于「文件系统」和「基于元数据的数据库」之间,相关论文已在Nature上发表。

把所有数据存储到DNA上的瓶颈
在DNA中存储数据的系统涉及到向包含数据的DNA片段添加特定的序列标签。
为了得到想要的数据,你只需添加能与正确的标签碱基配对的DNA位,并使用它们来扩增完整的序列。可以把它想象成用一个 ID 标记集合中的每个图像,然后进行设置,只放大一个特定的 ID。

这种方法是有效的,但它有两个方面的限制。
首先,使用称为PCR(聚合酶链式反应)的过程进行的扩增步骤,对可扩增的序列的大小有限制。而每个标签都会占用一些有限的空间,所以添加更多详细的标签(如复杂的文件系统可能需要)会减少数据空间。

一条 8 个 PCR 管,每个管含有 100 μL 反应混合物
另一个限制是,扩增特定数据片段的 PCR 反应会消耗一些原始的 DNA 库。换句话说,每次你拉出一些数据,你都会破坏成堆的不相关的数据。频繁地访问数据,最终会耗尽整个存储库。虽然有办法重新放大一切信息,但每次这样做都会增加引入错误的机会。
而这项新的研究已经将标签信息从数据存储中分离出来。此外,研究人员创建了一个系统,其中可以只访问你感兴趣的DNA数据,而不触及其余的数据,提高了数据存储的寿命。
给二氧化硅磁珠添加「涂层」
该基本技术是基于这样一个事实,即DNA会粘在二氧化硅磁珠(beads)上。
但这种吸力与DNA的大小无关,因此你可以使用这个系统存储任意大的数据块(在这种情况下,这些片段的大小是过去使用的典型的DNA数据存储块的10倍以上)。
同样重要的是,DNA中没有标签被存储在数据中,所以数据存储和文件系统信息之间没有竞争。
一旦DNA出现在这些磁珠的表面,研究人员就在其上面聚合一些额外的二氧化硅。这个过程涂抹了DNA并保护它不受环境影响。
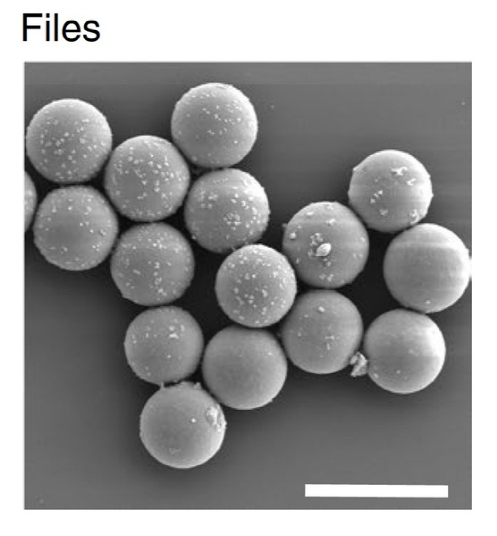
研究人员通过使用荧光标签来确认该系统是有效的;基本上,所有以这种方式创造的颗粒都含有DNA。

只有当这个外壳就位后,研究人员才添加标签,这些标签与外壳进行化学连接。这些标签是由单链DNA制成的,而且有可能在一个玻璃外壳上附着几个不同的标签。
研究人员对每个数据块分别进行了处理,一旦一切就绪,被标记的玻璃球就可以混入一个单一的数据库。
虽然没有纯DNA的存储那么紧凑,但仍然具有长期稳定和不需要能源维护的优势。
取代PCR
有趣的部分是访问数据。
除了成本之外,使用DNA存储数据的另一个主要瓶颈是,很难从所有文件中挑选出想要的文件。
此次开发的新的检索技术,希望取代PCR方法。
研究人员将每个DNA文件封装到一个微小的二氧化硅磁珠中,每个磁珠都贴上了由单链DNA组成的「条形码」,与文件内容相对应。
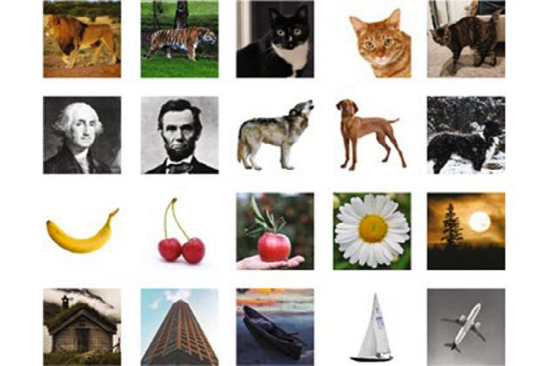
为了证明这种方法的成本效益,研究人员将20个不同的图像编码到大约长度为3000个核苷酸的DNA片段中,这大致相当于100个字节(研究还显示,这些磁珠可以容纳高达1GB的DNA文件)。
研究中的每个文件都有相应的条形码标签,如「猫」或「飞机」等。
当研究人员想要提取一个特定的图像时,他们会取出一个DNA样本,加入与目标标签相对应的引物。例如,老虎的图像对应的标签是「猫」「橘色」和「野生」,而家猫的图像对应「猫」「橘色」和「家养」。
这些引物用荧光或磁性颗粒标记,便于从样本中提取并识别匹配片段。
通过这种方法,研究人员可以将需要的文件移出来,剩下的DNA则完整地放回去,继续存储数据。
他们的检索过程允许「布尔逻辑语句」,如「总统和18世纪」会生成「乔治·华盛顿」的结果,这很类似谷歌的图像检索。
在目前的概念验证阶段,搜索速度是每秒1000字节(1KB)。文件系统的搜索速度是由每个磁珠的数据量大小决定的,而目前限制数据量大小的因素就是在DNA上写入100兆字节(MB)数据所需的高昂成本,以及可以并行使用的分类器的数量。
如果DNA合成变得足够便宜,就能够用这种方法将每个文件存储的数据量最大化
DNA数据存储目前局限于「冷存储」
该系统还允许用多个术语进行「布尔搜索」(Boolean search)。
通过一个接一个地选择不同的标签,你可以建立起相当复杂的条件:猫为真,驯养的为假,黑为真,等等。
给两个标签贴上相同的荧光颜色,如果你抓到任何带有这种颜色的东西,你就可以得到相当于逻辑OR的结果。
因为这些标签中的每一个都可以被看作是关于DNA所存储的图像的元数据,磁珠的集合最终作为一个元数据驱动的图像数据库。
虽然这项研究代表了基于DNA的存储在复杂性方面的一个重大飞跃,但它仍然只是基于DNA的存储。
这意味着它的速度之慢,甚至还不如磁带驱动器。

根据研究人员的计算,即使他们把更多的数据塞进每颗磁珠,搜索上限只是每秒约1GB的数据。这将意味着搜索PB级的数据将需要「两周多」的时间。
而这仅仅是找到合适的磁珠。敲开它们,将DNA放进去,然后进行必要的测序,以实际确定磁珠中储存的内容,这又会使实验过程增加几天。
当然,没有人会因为DNA存储「速度快」而推荐它;正如上面提到的,它的优势在能源使用和数据稳定性方面。
我们只有在确定不会经常访问某些数据时才会将它储存在DNA中,也即「冷存档存储」。
不过,目前,该实验室已经成立了一家名为Cache DNA的初创公司,正在开发DNA的长期存储技术,既可以用于长期的DNA数据存储,也能用于短期的临床和其他现有的DNA样品存储。

https://www.cache-dna.com/
虽然可能还需要一段时间才能将DNA作为数据存储介质,但目前在Covid-19检测、人类基因组测序和其他基因组学领域中,对于DNA和RNA样品的低成本和大规模存储的解决方案都有很大需求。
参考资料:
https://arstechnica.com/science/2021/06/researchers-build-a-metadata-based-image-database-using-dna-storage/
https://www.ssbt.org.cn/upload/20190905163302_429.pdf
https://news.mit.edu/2021/dna-data-storage-0610
-往期精彩-



