Python数学建模系列(五):微分方程
1、微分方程分类
微分方程是用来描述某一类函数与其导数之间关系的方程,其解是一个符合方程的函数。
微分方程按自变量个数可分为常微分方程和偏微分方程
常微分方程(ODE:ordinary differential equation)
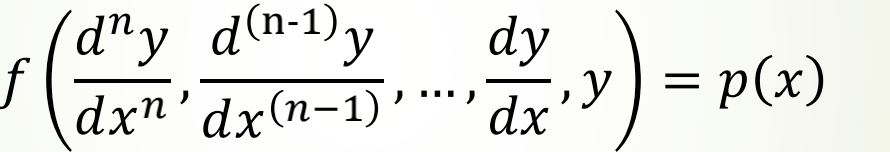
偏微分方程(两个以上的自变量)
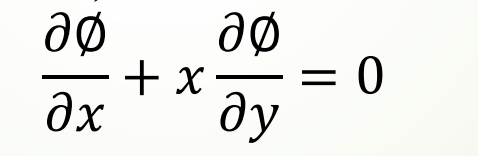
2、微分方程解析解
具备解析解的ODE(常微分方程),我们可以利用SymPy库进行求解
以求解阻尼谐振子的二阶ODE为例,其表达式为:
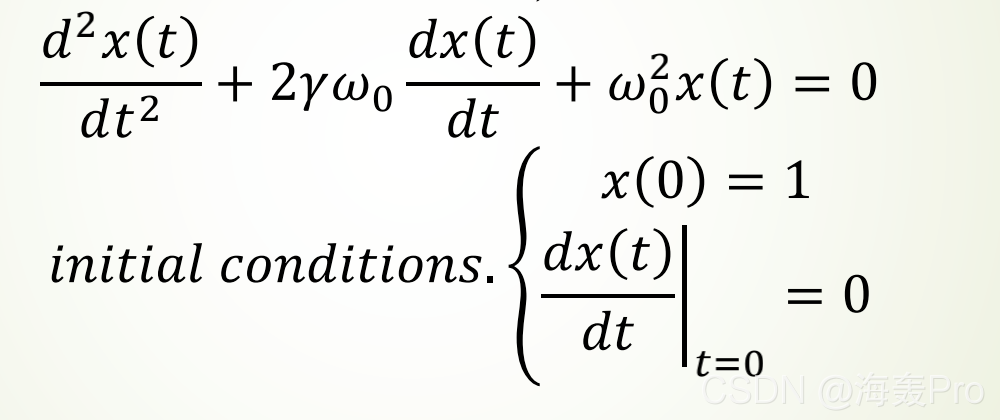
Demo代码
import sympy
def apply_ics(sol, ics, x, known_params):
free_params = sol.free_symbols - set(known_params)
eqs = [(sol.lhs.diff(x, n) - sol.rhs.diff(x, n)).subs(x, 0).subs(ics) for n in range(len(ics))]
sol_params = sympy.solve(eqs, free_params)
return sol.subs(sol_params)
# 初始化打印环境
sympy.init_printing()
# 标记参数,且均为正
t, omega0, gamma = sympy.symbols("t, omega_0, gamma", positive=True)
# 标记x是微分函数,非变量
x = sympy.Function("x")
# 用diff()和dsolve得到通解
# ode 微分方程等号左边的部分,等号右边为0
ode = x(t).diff(t, 2) + 2 * gamma * omega0 * x(t).diff(t) + omega0 ** 2 * x(t)
ode_sol = sympy.dsolve(ode)
# 初始条件:字典匹配
ics = {x(0): 1, x(t).diff(t).subs(t, 0): 0}
x_t_sol = apply_ics(ode_sol, ics, t, [omega0, gamma])
sympy.pprint(x_t_sol)
运行结果:
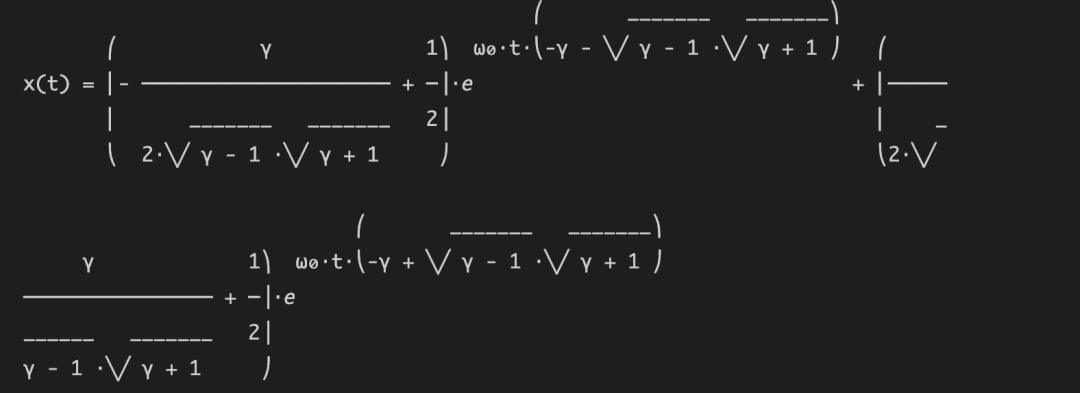
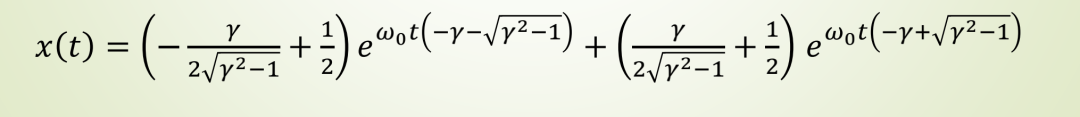
3、微分方程数值解
当ODE无法求得解析解时,可以用scipy中的integrate.odeint求 数值解来探索其解的部分性质,并辅以可视化,能直观地展现 ODE解的函数表达。
以如下一阶非线性(因为函数y幂次为2)ODE为例:
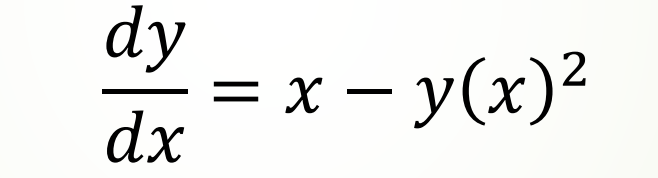
现用odeint求其数值解
3.1 场线图与数值解
Demo代码
import numpy as np
from scipy import integrate
import matplotlib.pyplot as plt
import sympy
def plot_direction_field(x, y_x, f_xy, x_lim=(-5, 5), y_lim=(-5, 5), ax=None):
f_np = sympy.lambdify((x, y_x), f_xy, 'numpy')
x_vec = np.linspace(x_lim[0], x_lim[1], 20)
y_vec = np.linspace(y_lim[0], y_lim[1], 20)
if ax is None:
_, ax = plt.subplots(figsize=(4, 4))
dx = x_vec[1] - x_vec[0]
dy = y_vec[1] - y_vec[0]
for m, xx in enumerate(x_vec):
for n, yy in enumerate(y_vec):
Dy = f_np(xx, yy) * dx
Dx = 0.8 * dx**2 / np.sqrt(dx**2 + Dy**2)
Dy = 0.8 * Dy*dy / np.sqrt(dx**2 + Dy**2)
ax.plot([xx - Dx/2, xx + Dx/2], [yy - Dy/2, yy + Dy/2], 'b', lw=0.5)
ax.axis('tight')
ax.set_title(r"$%s$" %(sympy.latex(sympy.Eq(y_x.diff(x), f_xy))), fontsize=18)
return ax
x = sympy.symbols('x')
y = sympy.Function('y')
f = x-y(x)**2
f_np = sympy.lambdify((y(x), x), f)
## put variables (y(x), x) into lambda function f.
y0 = 1
xp = np.linspace(0, 5, 100)
yp = integrate.odeint(f_np, y0, xp)
## solve f_np with initial conditons y0, and x ranges as xp.
xn = np.linspace(0, -5, 100)
yn = integrate.odeint(f_np, y0, xn)
fig, ax = plt.subplots(1, 1, figsize=(4, 4))
plot_direction_field(x, y(x), f, ax=ax)
## plot direction field of function f
ax.plot(xn, yn, 'b', lw=2)
ax.plot(xp, yp, 'r', lw=2)
plt.show()
运行结果:

3.2 洛伦兹曲线与数值解
以求解洛伦兹曲线为例,以下方程组代表曲线在xyz三个方向 上的速度,给定一个初始点,可以画出相应的洛伦兹曲线:
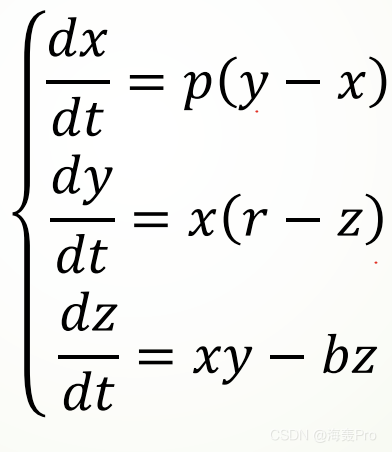
Demo代码
import numpy as np
from scipy.integrate import odeint
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
def dmove(Point, t, sets):
p, r, b = sets
x, y, z = Point
return np.array([p * (y - x), x * (r - z), x * y - b * z])
t = np.arange(0, 30, 0.001)
P1 = odeint(dmove, (0., 1., 0.), t, args=([10., 28., 3.],))
P2 = odeint(dmove, (0., 1.01, 0.), t, args=([10., 28., 3.],))
fig = plt.figure()
ax = Axes3D(fig)
ax.plot(P1[:, 0], P1[:, 1], P1[:, 2])
ax.plot(P2[:, 0], P2[:, 1], P2[:, 2])
plt.show()
运行结果:
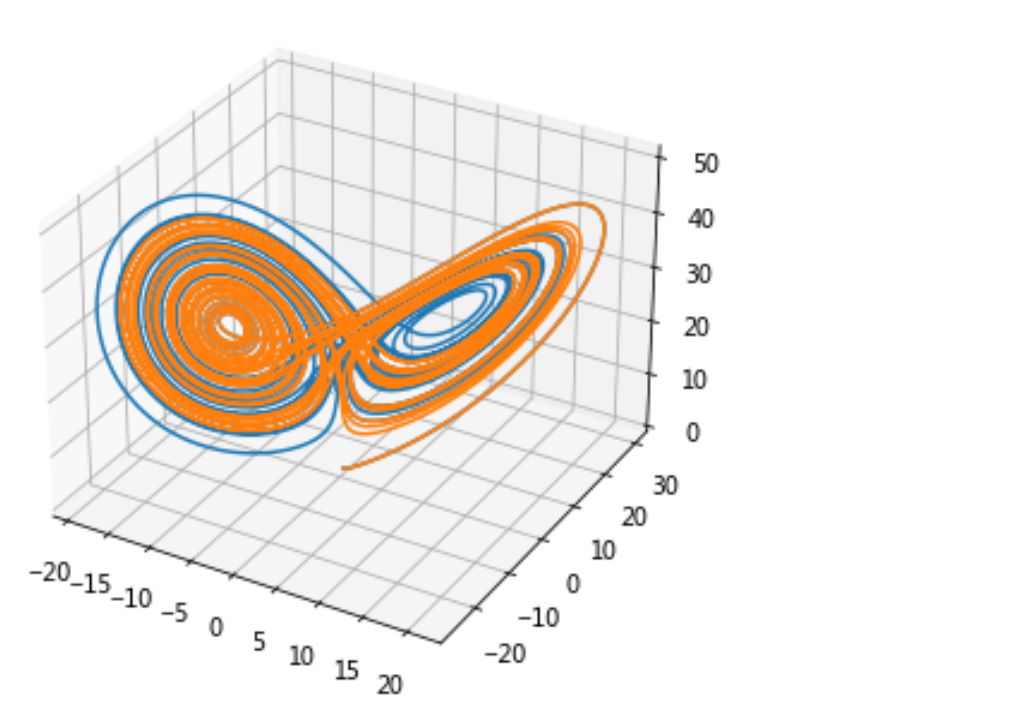
4、传染病模型

模型一:SI-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0
# I_0为感染者的初始人数
I_0 = 1
# S_0为易感者的初始人数
S_0 = N - I_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0)
def funcSI(inivalue,_):
Y = np.zeros(2)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N + gamma * X[1]
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSI,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'red',label = 'Infection',marker = '.')
plt.title('SI Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()
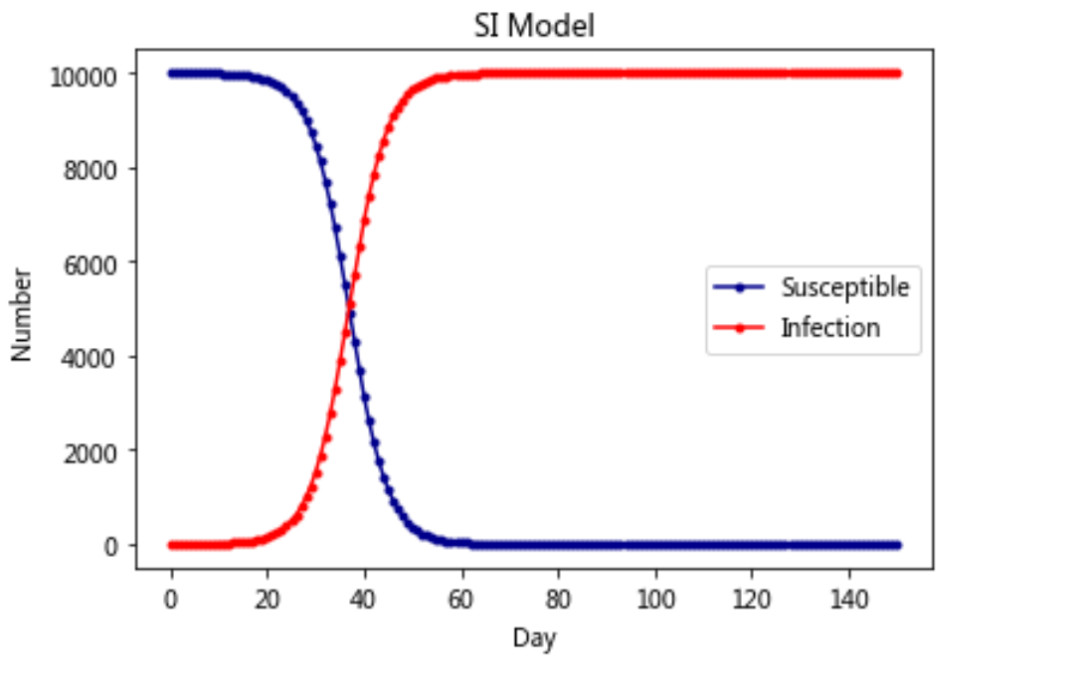
模型二:SIS model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# I_0为感染者的初始人数
I_0 = 1
# S_0为易感者的初始人数
S_0 = N - I_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0)
def funcSIS(inivalue,_):
Y = np.zeros(2)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0]) / N * X[1] + gamma * X[1]
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIS,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'red',label = 'Infection',marker = '.')
plt.title('SIS Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()
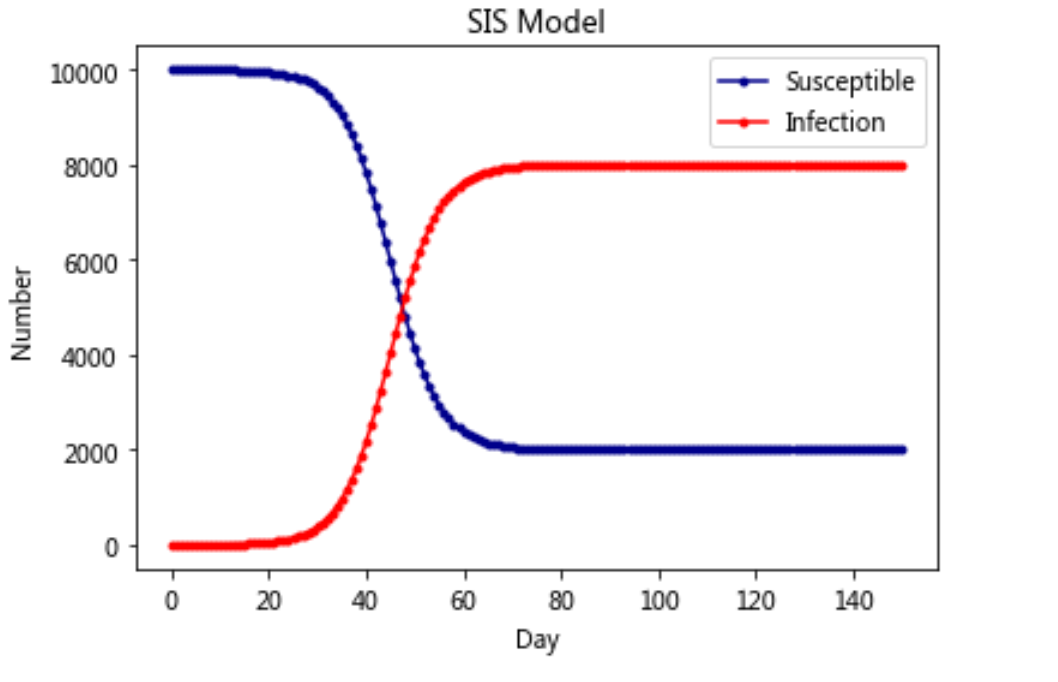
模型三:SIR model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# I_0为感染者的初始人数
I_0 = 1
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0,R_0)
def funcSIR(inivalue,_):
Y = np.zeros(3)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
# 治愈个体变化
Y[2] = gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIR,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'red',label = 'Infection',marker = '.')
plt.plot(RES[:,2],color = 'green',label = 'Recovery',marker = '.')
plt.title('SIR Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()
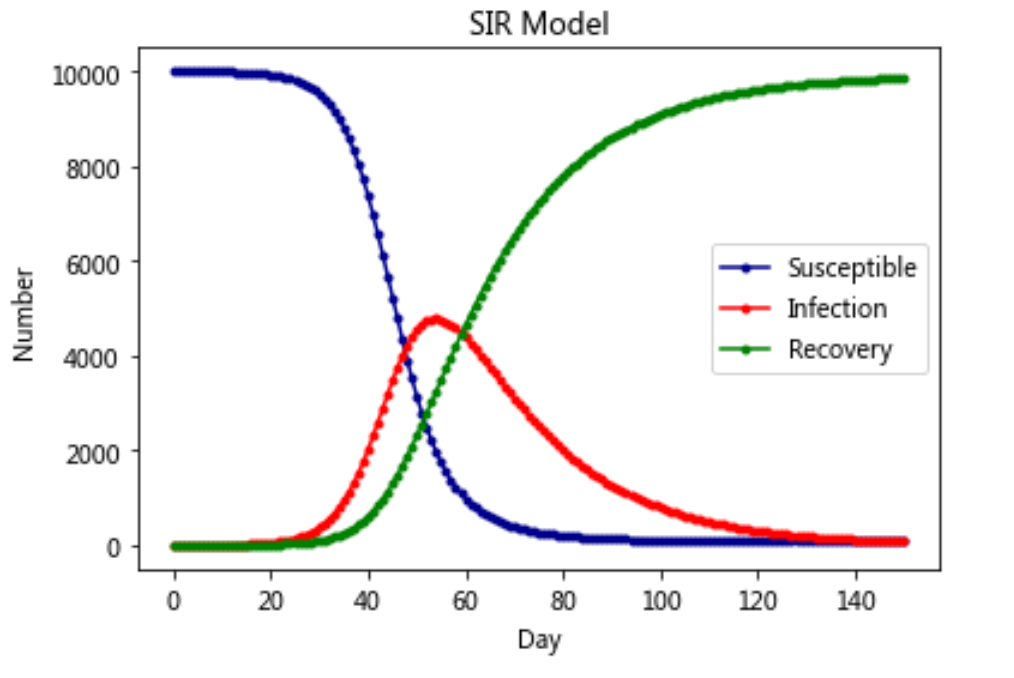
模型四:SIRS-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# Ts为抗体持续时间
Ts = 7
# I_0为感染者的初始人数
I_0 = 1
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0,R_0)
def funcSIRS(inivalue,_):
Y = np.zeros(3)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N + X[2] / Ts
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
# 治愈个体变化
Y[2] = gamma * X[1] - X[2] / Ts
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIRS,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'red',label = 'Infection',marker = '.')
plt.plot(RES[:,2],color = 'green',label = 'Recovery',marker = '.')
plt.title('SIRS Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()
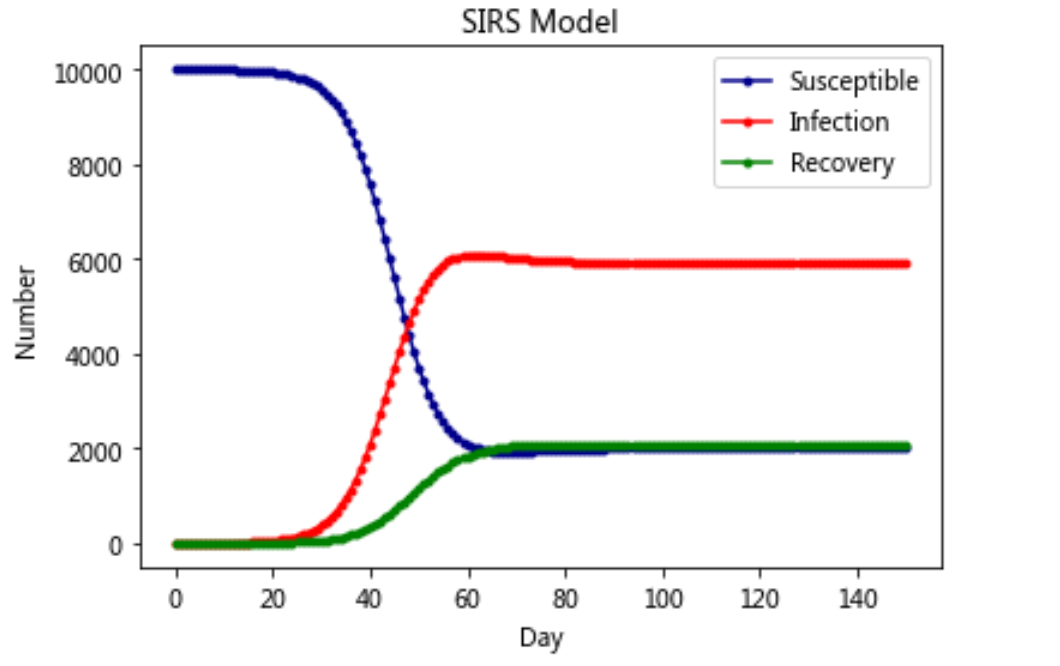
模型五:SEIR-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.6
# gamma为恢复率系数
gamma = 0.1
# Te为疾病潜伏期
Te = 14
# I_0为感染者的初始人数
I_0 = 1
# E_0为潜伏者的初始人数
E_0 = 0
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - E_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,E_0,I_0,R_0)
def funcSEIR(inivalue,_):
Y = np.zeros(4)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[2]) / N
# 潜伏个体变化
Y[1] = (beta * X[0] * X[2]) / N - X[1] / Te
# 感染个体变化
Y[2] = X[1] / Te - gamma * X[2]
# 治愈个体变化
Y[3] = gamma * X[2]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSEIR,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'orange',label = 'Exposed',marker = '.')
plt.plot(RES[:,2],color = 'red',label = 'Infection',marker = '.')
plt.plot(RES[:,3],color = 'green',label = 'Recovery',marker = '.')
plt.title('SEIR Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()

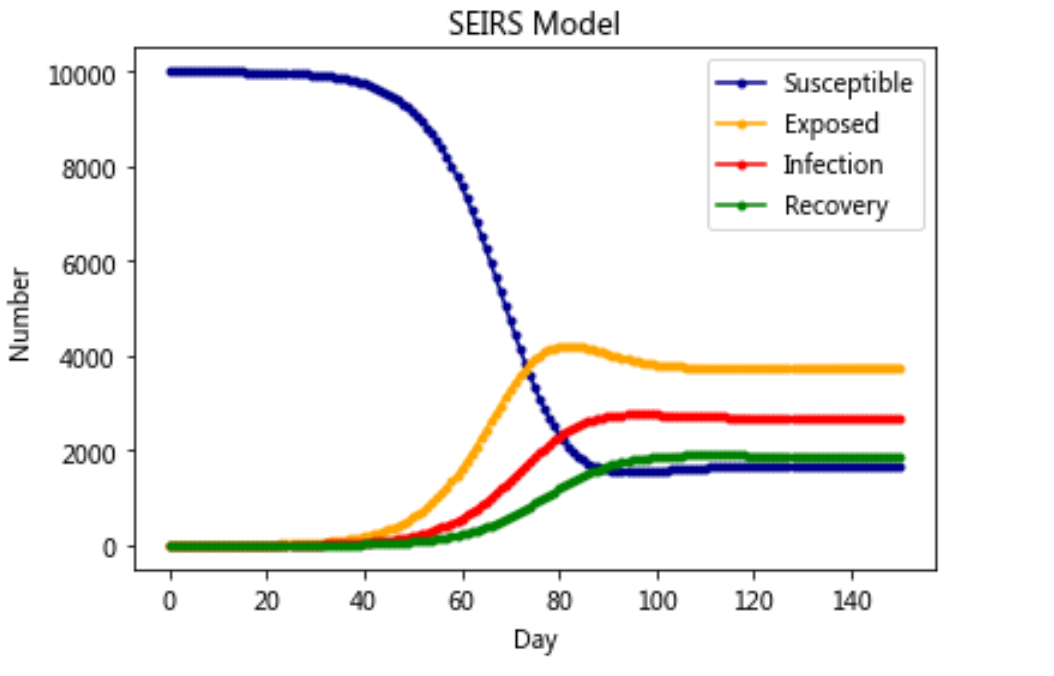
模型六:SEIRS-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.6
# gamma为恢复率系数
gamma = 0.1
# Ts为抗体持续时间
Ts = 7
# Te为疾病潜伏期
Te = 14
# I_0为感染者的初始人数
I_0 = 1
# E_0为潜伏者的初始人数
E_0 = 0
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - E_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,E_0,I_0,R_0)
def funcSEIRS(inivalue,_):
Y = np.zeros(4)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[2]) / N + X[3] / Ts
# 潜伏个体变化
Y[1] = (beta * X[0] * X[2]) / N - X[1] / Te
# 感染个体变化
Y[2] = X[1] / Te - gamma * X[2]
# 治愈个体变化
Y[3] = gamma * X[2] - X[3] / Ts
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSEIRS,INI,T_range)
plt.plot(RES[:,0],color = 'darkblue',label = 'Susceptible',marker = '.')
plt.plot(RES[:,1],color = 'orange',label = 'Exposed',marker = '.')
plt.plot(RES[:,2],color = 'red',label = 'Infection',marker = '.')
plt.plot(RES[:,3],color = 'green',label = 'Recovery',marker = '.')
plt.title('SEIRS Model')
plt.legend()
plt.xlabel('Day')
plt.ylabel('Number')
plt.show()
结语
参考:
https://www.bilibili.com/video/BV12h411d7Dm https://zhuanlan.zhihu.com/p/104091330
学习来源:
B站及其课堂PPT 对其中代码进行了复现
「文章仅作为学习笔记,记录从0到1的一个过程」
希望对您有所帮助,如有错误欢迎小伙伴指正~
评论
