易汉博承建的数据库再发Nature子刊
数据资源是未来重要的战略资源。生物数据积累越来越多,高效规范地将其呈现出来有利于数据的进一步挖掘和利用,之前分享了我们承建的三篇NAR的数据库,包括中药数据、海洋天然产物数据和噬菌体预测工具等。这次介绍的是微生物根系细菌分离和培养在线工具,发表于`Nature protocols`,影响因子15。

原文链接:https://www.nature.com/articles/s41596-020-00444-7
Nature Protocols:中科院遗传发育所白洋组发表文章详细介绍高通量分离培养和鉴定植物根系细菌的实验流程与分析方法
植物根系微生物组的研究主要依赖于高通量扩增子和宏基因组测序技术,对微生物组的物种分类和基因组成进行描述。原位分离培养微生物对于揭示微生物在植物生长和健康中的功能非常重要。分离培养的微生物和无菌体系相结合,将揭示根系微生物与植物生长表型之间的因果关系和互作机制,是推动根系微生物组从描述向功能研究发展的重要技术。
白洋组在Nature Protocols 杂志撰写文章详细介绍高通量分离培养和鉴定植物根系细菌的实验流程与分析方法。该方法从新鲜植物根系中高通量分离培养细菌,使用梯度稀释的方法增加获得单一细菌的比例,采用双侧标签PCR扩增法高通量鉴定分离培养细菌的16S rRNA基因(图1)。为便于数据处理,开发了一个简单易用的生物信息分析流程Culturome (https://github.com/YongxinLiu/Culturome)和一个图形用户界面网络服务器(http://bailab.genetics.ac.cn/culturome/)。该方法允许任何研究组(2-3个实验室成员,无需生物信息学专业知识)在8-9周内系统地培养植物根系相关细菌。利用该方法,白洋组系统地建立了植物根系细菌资源库(Nature, 2015),揭示拟南芥三萜类化合物选择性调控根系微生物组的功能和机制(Science, 2019),以及水稻协同根系微生物组利用土壤氮元素(Nature Biotechnology, 2019)。
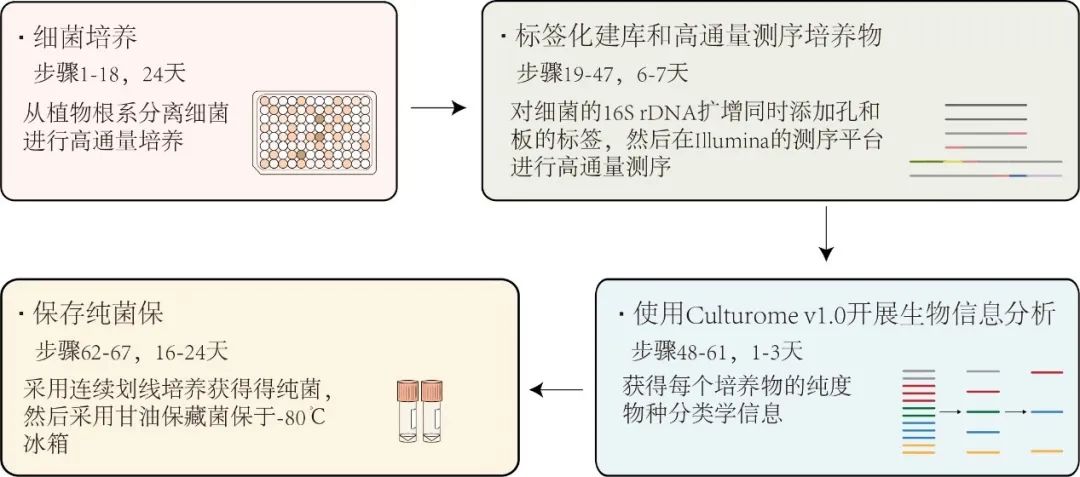
图1. 高通量细菌分离培养和鉴定方法概述
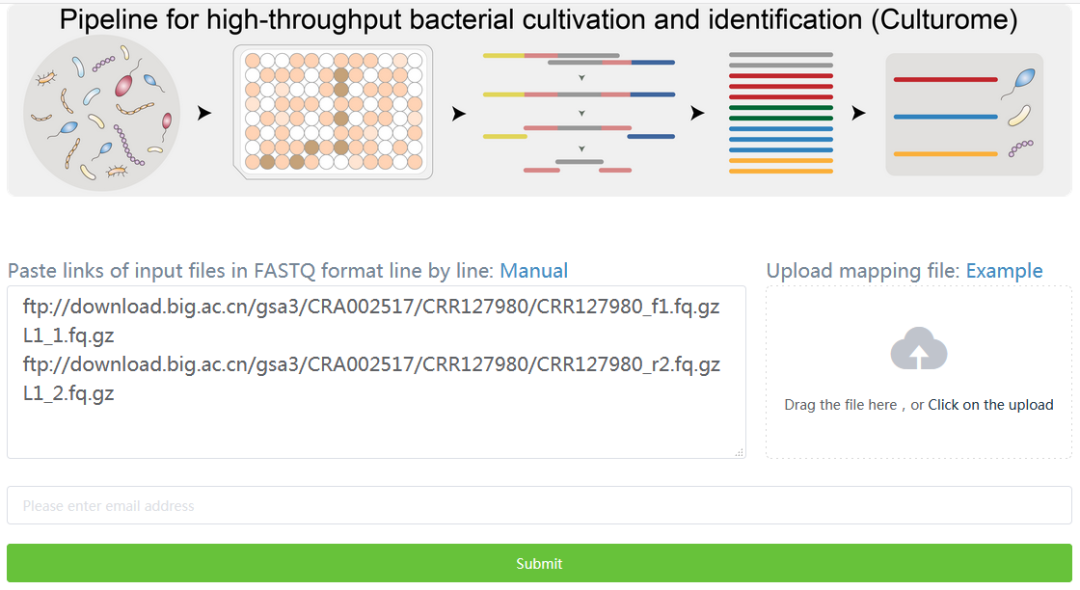
数据平台界面简洁大方
此项成果于2021年1月13日在线发表于Nature Protocols 杂志(doi: 10.1038/s41596-020-00444-7)。白洋组张婧赢助理研究员、刘永鑫工程师为共同第一作者,中科院遗传发育所的白洋研究员、德国马普植物育种研究所的Paul Schulze-Lefert和Ruben Garrido-Oter研究员为共同通讯作者。白洋研究组的郭晓璇和秦媛参与了本项目。该研究得到了中国科学院战略性先导科技专项、中国科学院前沿科学重点研究项目、国家自然科学基金面上项目和青年项目、以及中国科学院青年创新促进会的支持。
Jingying Zhang, Yong-Xin Liu, Xiaoxuan Guo, Yuan Qin, Ruben Garrido-Oter, Paul Schulze-Lefert & Yang Bai. (2021). High-throughput cultivation and identification of bacteria from the plant root microbiota. Nature Protocols, doi: https://doi.org/10.1038/s41596-020-00444-7
