单个基因集富集分析泡泡图绘制
富集分析是生物信息分析中快速了解目标基因或目标区域功能倾向性的最重要方法之一。其中代表性的计算方式有两种:
一是基于筛选的差异基因,采用超几何检验判断上调或下调基因在哪些GO或KEGG或其它定义的通路富集。假设背景基因数目为t,背景基因中某一通路pathway中注释的基因有m个;上调基因有k个,上调基因中落于通路pathway的数目为q。简单来讲就是比较q/k是否显著高于m/t,即上调基因中落在通路pathway的比例是否高于背景基因在这一通路的比例。(实际计算时,是算的odds ratio的差异,q/(k-q) vs (m-q)/(t-k-m+q))。这就是常说的GO富集分析或KEGG富集分析,可以做的工具很多,GOEAST是其中一个最好用的在线功能富集分析工具,数据库更新实时,操作简单,并且可以直接用之前介绍的方法绘制DotPlot。
另一种方式是不硬筛选差异基因,而是对其根据表达量或与表型的相关度排序,然后判断对应的基因集是否倾向于落在有序列表的顶部或底部,从而判断基因集合对表型差异的影响和筛选有影响的基因子集。这叫GSEA富集分析,注释信息可以是GO,KEGG,也可以是其它任何符合格式的信息。GSEA富集分析 - 界面操作详细讲述了GSEA分析的原理、可视化操作和结果解读。
具体原理解释见我们在B站的免费视频:易生信转录组高级课程系列节选
GOEAST结果绘制富集分析泡泡图
单个基因集富集结果展示
在去东方,最好用的在线GO富集分析工具一文中介绍了一款高引用、操作简单、数据库每周同步更新的在线富集工具GOEAST,很受好评。美中不足的是,这个工具不能输出泡泡图。下面我们展示下如何用GOEAST输出的富集结果表格自行筛选条目绘制富集分析泡泡图。
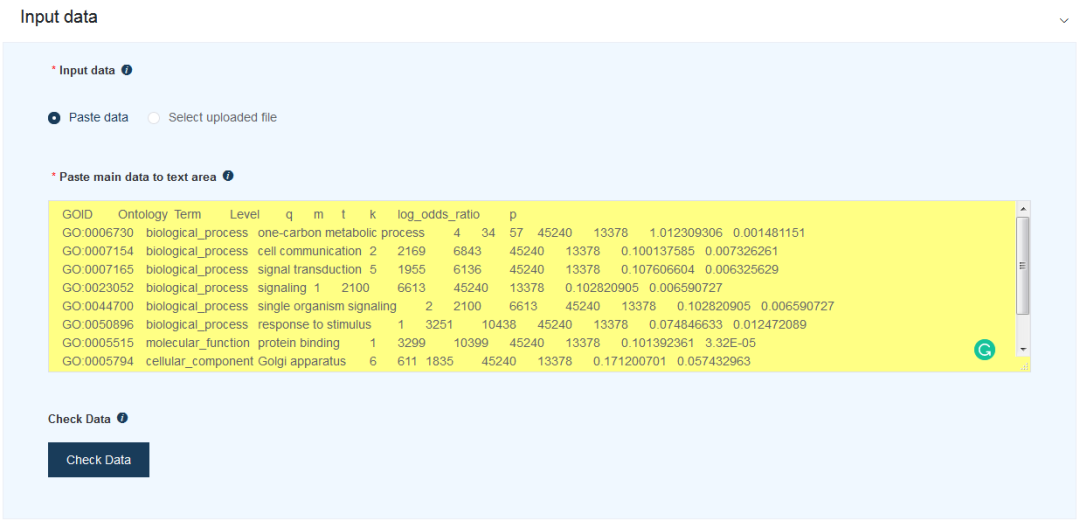
GOEAST输出的表格内容如下 (geneIDs symbols 列内容较长,此处没用到,故未展示):
GOID Ontology Term Level q m t k log_odds_ratio p
GO:0006730 biological_process one-carbon metabolic process 4 34 57 45240 13378 1.012309306 0.001481151
GO:0007154 biological_process cell communication 2 2169 6843 45240 13378 0.100137585 0.007326261
GO:0007165 biological_process signal transduction 5 1955 6136 45240 13378 0.107606604 0.006325629
GO:0023052 biological_process signaling 1 2100 6613 45240 13378 0.102820905 0.006590727
GO:0044700 biological_process single organism signaling 2 2100 6613 45240 13378 0.102820905 0.006590727
GO:0050896 biological_process response to stimulus 1 3251 10438 45240 13378 0.074846633 0.012472089
GO:0005515 molecular_function protein binding 1 3299 10399 45240 13378 0.101392361 3.32E-05
GO:0005794 cellular_component Golgi apparatus 6 611 1835 45240 13378 0.171200701 0.057432963
GO:0012505 cellular_component endomembrane system 2 1521 4648 45240 13378 0.146146563 0.000353056
GO:0071944 cellular_component cell periphery 2 2059 6559 45240 13378 0.086204434 0.065663723我们先看下其中几列的含义是什么:
q: 用于分析的基因集中匹配到该通路的基因数目m: 背景基因集中落在该通路的基因数目t: 背景基因集中总的基因数目k: 用于分析的基因集中总的基因数目p: 富集显著性值(FDR,多重假设检验校正后的p-value)log_odds_ratio: 富集比,具体见上面基础部分
富集分析泡泡图实际是一种散点图,这个图怎么绘制需要我们先理解这个图每一部分的含义。理解了图,剩下的就是把对应列的信息赋值到图上。
我们先把数据导入平台http://www.ehbio.com/Cloud_Platform/front/#/analysis?page=b%27MTA%3D%27,
选择一些参数,体会下它们在图上的体现和意义。
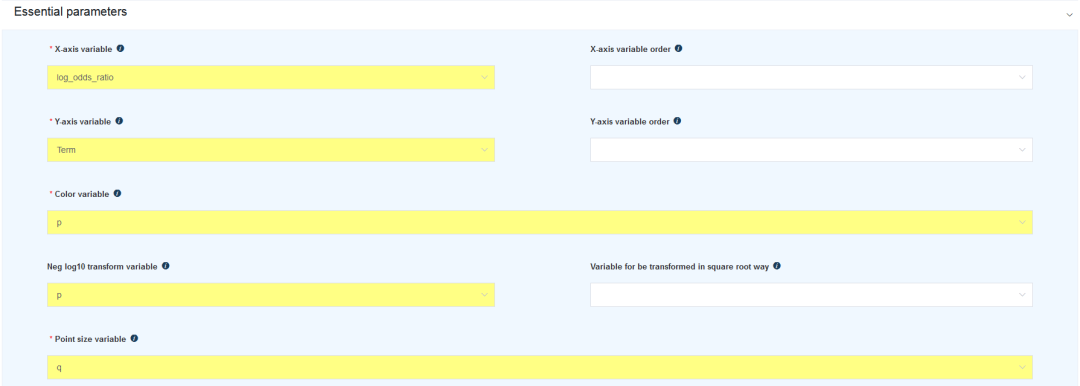
然后选择参数
log_odds_ratio列作为横轴(X-axis)信息Term列作为纵轴(Y-axis)信息
这两列就确定了点的分布,下面三个参数是给点的属性赋值
统计显著性
p列作为Color variable,给每个点根据数值大小进行上色,从颜色上区分富集显著性q列用于设置点的大小Point size variable,点越大表示目标基因集中落在对应通路的基因越多Neg log10 transform variable是指定哪个变量进行对数转换,这是可选参数,但通常我们会对p-value列做这个转换。转换后越小的
p-value值就会变得越大
提交后,获得结果图如下:
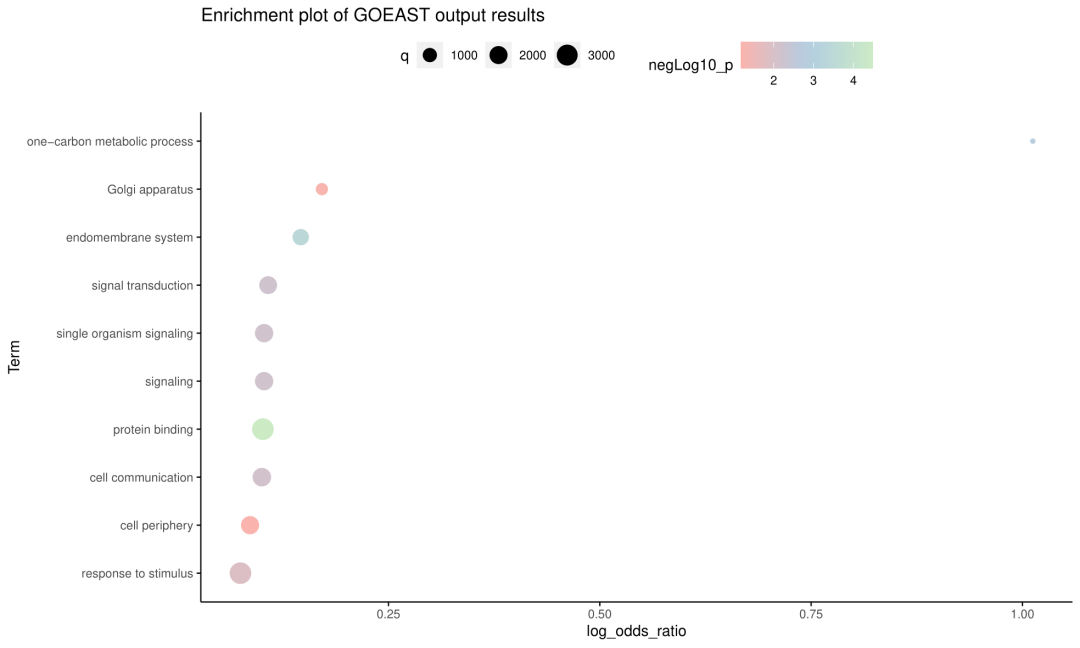
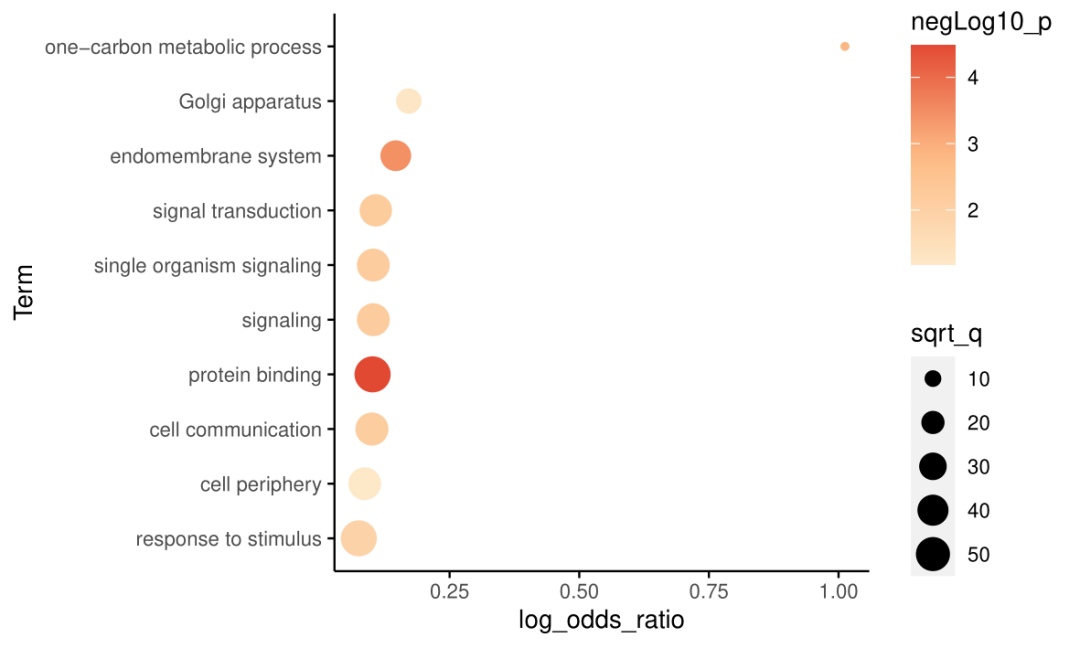
图中每个点代表一个富集的条目,在Y轴有对应标记。这些条目按其log_odds_ratio的值排序后展示,log_odds_ratio高的条目在Y轴上方展示;每个点的大小代表用于分析的基因集中匹配到该通路的基因数目,颜色代表富集程度。
但这个图中,点的大小有些太分散,颜色是绿色饱和度越高表示富集越显著,可能跟常规认知不同。修改两个参数:
Variable for be transformed in square root way选择q,通过平方根降低数据之间的差距设置颜色
Manual color vector (color set)为OrRd
获得结果如下
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集