90后斯坦福博士论文登Science封面!AI算法准确预测RNA三维结构

来源:七月在线实验室 本文约1200字,建议阅读5分钟
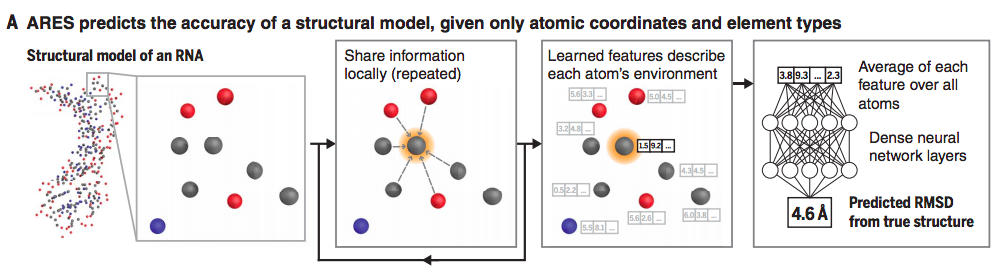
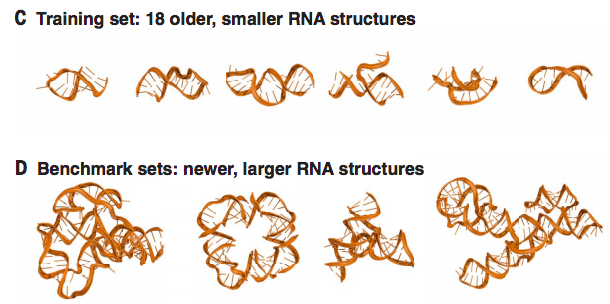
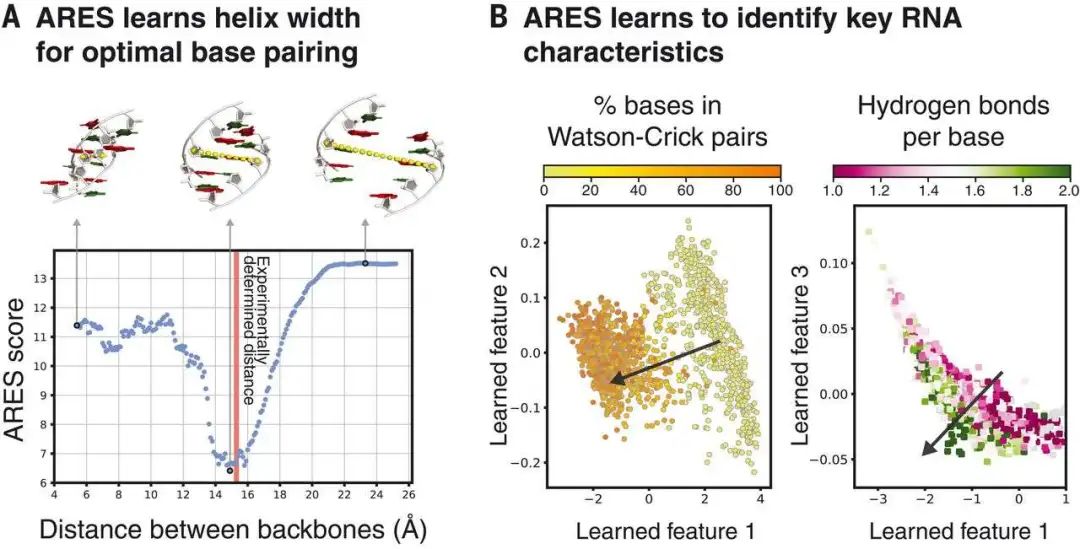
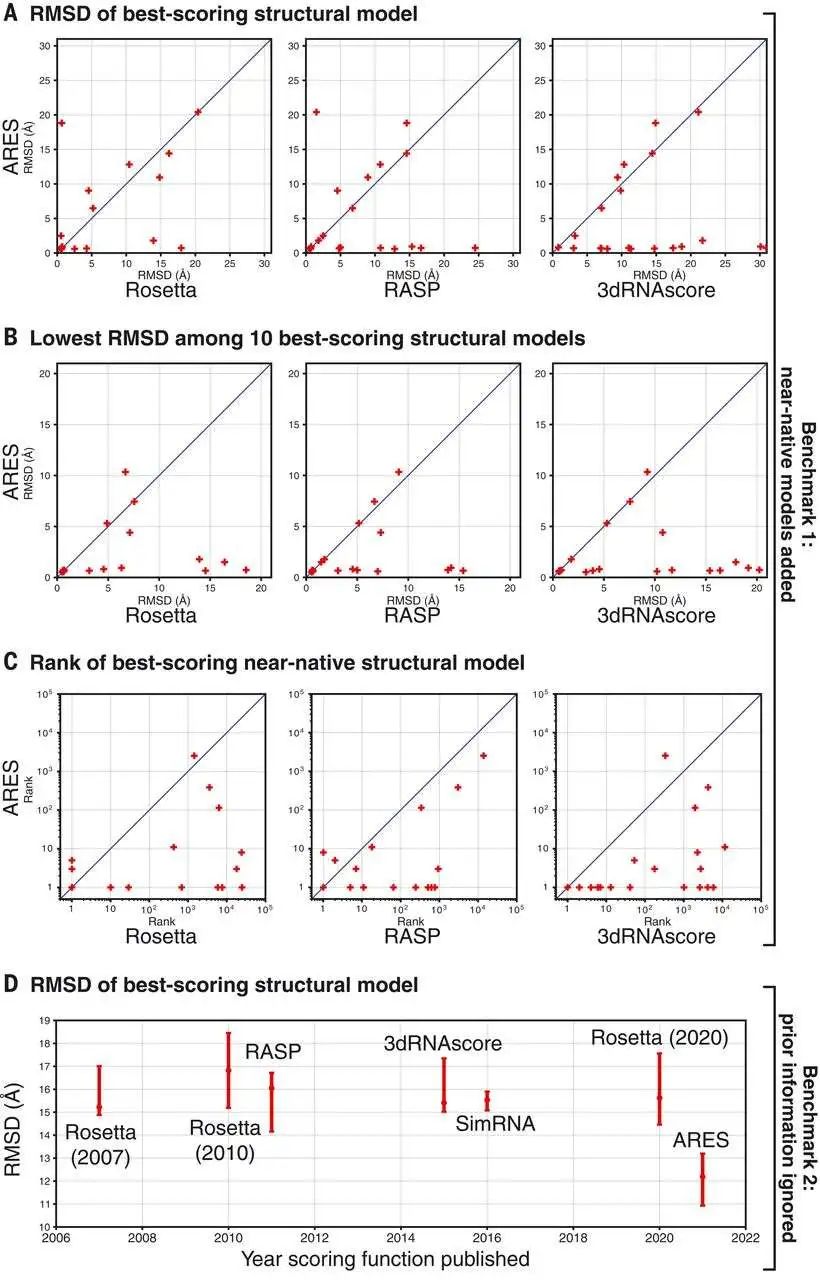
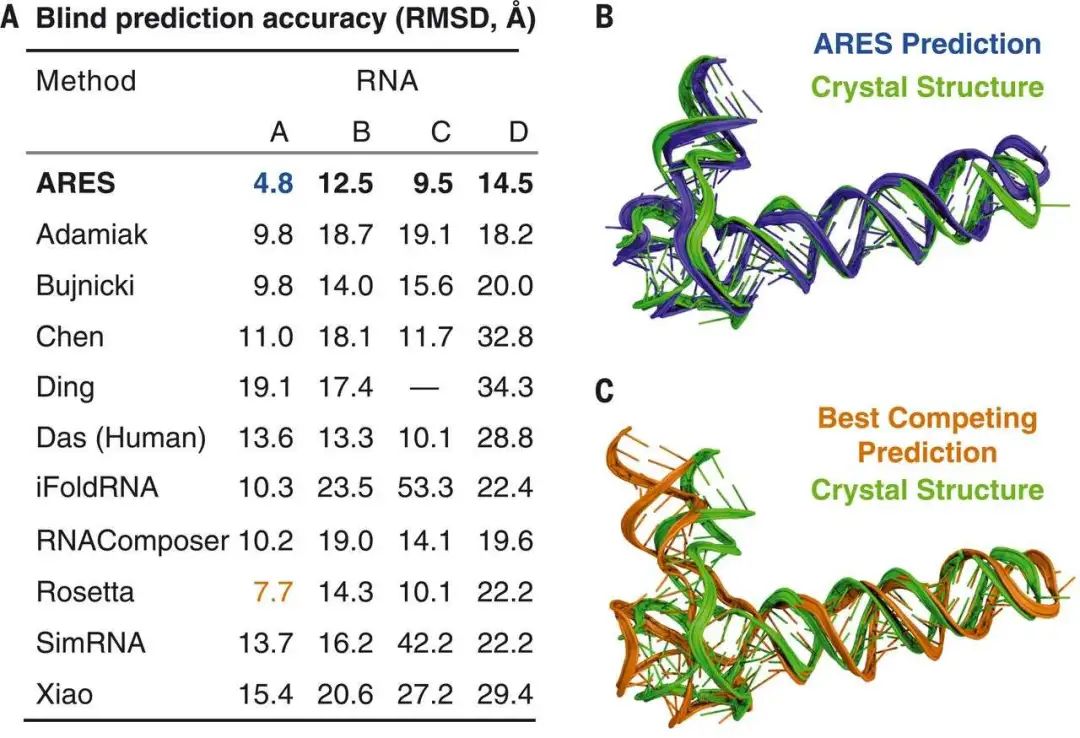
本文介绍了Science 封面的这项研究的最新成果,利用目前先进的神经网络技术,成功开发出了一种全新 RNA 三维结构预测模型——ARES。
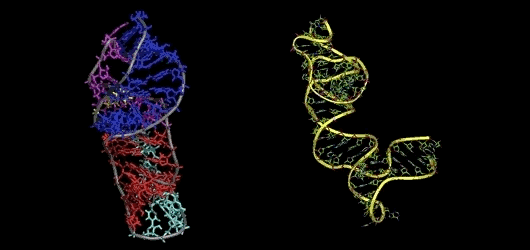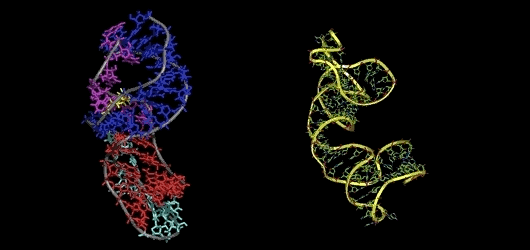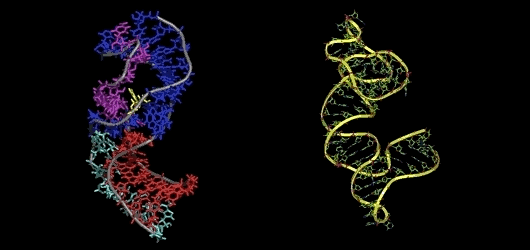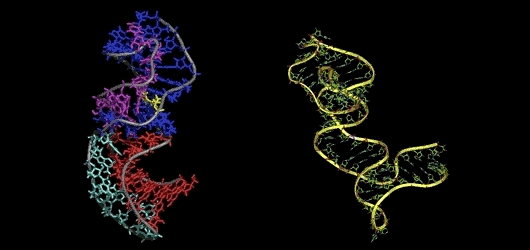



编辑:王菁
校对:林亦霖
评论
 下载APP
下载APP
来源:七月在线实验室 本文约1200字,建议阅读5分钟
本文介绍了Science 封面的这项研究的最新成果,利用目前先进的神经网络技术,成功开发出了一种全新 RNA 三维结构预测模型——ARES。











编辑:王菁
校对:林亦霖