Nature methods | Alevin-fry, 一种高效准确的单细胞测序数据预处理工具
随着单细胞以及单核测序(single-cell and single-nucleus RNA-sequencing)的快速发展以及逐渐普及,越来越多的单细胞测序数据集在近几年不断的出现。这些数据集不仅有着成倍增加的细胞数量,也包括了更多维度的生物特征信息,这使得预处理单细胞测序数据的所需的时间不断增加。例如,10x Genomics的官方预处理软件CellRanger有时需要几十个小时来处理一个单一数据集。虽然目前有些轻量化工具可较快的处理单细胞测序数据,比如kallisto|bustools 和alevin,但轻量化工具的假阳性和准确性问题一直有待解决。

在2022年三月的Nature Methods期刊中,美国马里兰大学的Rob Patro团队发表的了名为alevin-fry的单细胞测序数据预处理工具来一站式高效便捷的量化sc/snRNA-seq数据。通常情况下,alevin-fry可在15分钟内以小于8 GB的内存用量处理单细胞或单核测序数据集,并与任何其他工具所能达到的最佳准确率持平。该工具用当前热门的Rust编程语言书写,不仅在GitHub (https://github.com/COMBINE-lab/alevin-fry)上完全开源, 并且上传至crates.io (https://crates.io/crates/alevin-fry)及 bioconda (https://anaconda.org/bioconda/alevin-fry)以供下载安装,还有有十分完善的配套工具 (https://github.com/COMBINE-lab/usefulaf)用于构建参考序列以及导入量化分析结果。
除了比其他任何量化方法都更快、更节省内存之外,alevin-fry还改进了量化工具中普遍存在的内存可伸缩性问题和其他轻量化工具表现出的假阳性表达问题。除此之外,alevin-fry所产生的量化结果还可被直接用于多种下游分析,例如对单细胞速率(RNA velocity)的研究。
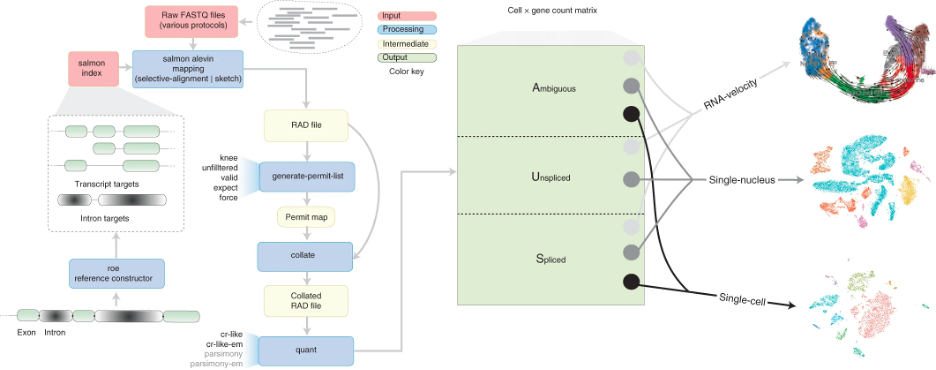
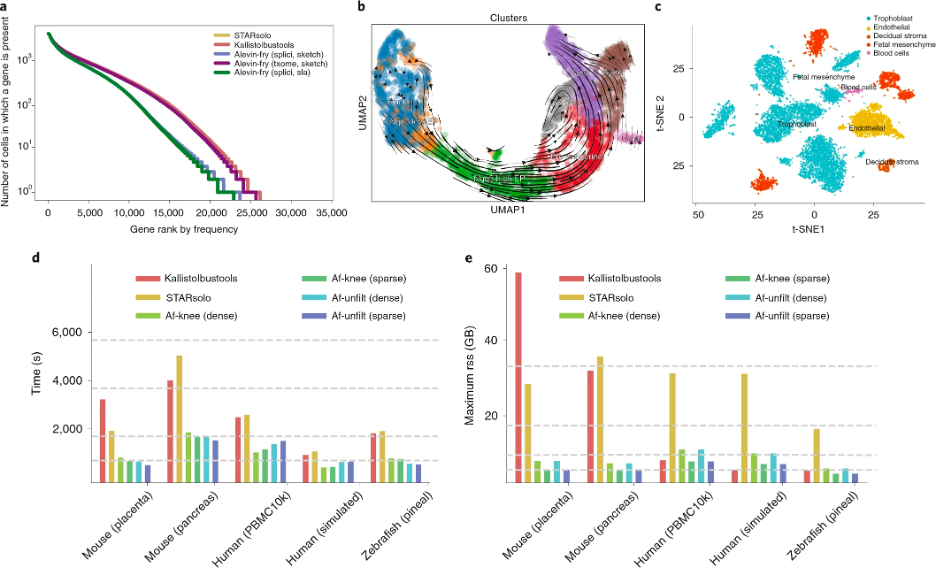
在业界普遍认为的效率(速度及内存占用)或准确性二选一的难题中,论文展示了alevin-fry做到了不仅在效率上处于全业最优,而且达成了其他以全基因组为基础的量化工具用牺牲效率换来的高准确性。可以说alevin-fry不仅是现今最高效的单细胞预处理工具,也是现如今最准确的单细胞预处理工具之一。在结果中,该论文指出了alevin-fry不仅解决了在轻量化工具中普遍存在的假阳性问题,还在其他工具普遍需要额外的内存及时间来处理单核测序数据的情况下做到了可将单细胞及单核测序数据统一化高效处理。除此之外,该论文还示范了如何用alevin-fry来处理各种类型的数据集,例如单细胞测序,单核测序和RNA速率的示例数据集,并展示了alevin-fry的量化结果相比于其他任何的量化工具保留了最多的生物信号。
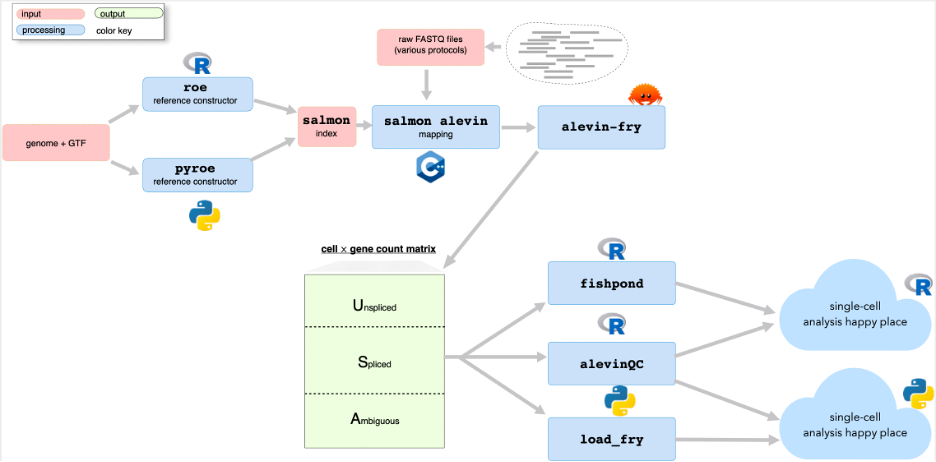
为方便用户使用,该团队为alevin-fry准备了完整的配套工具 https://hub.docker.com/r/combinelab/usefulaf。对于构建参考序列,R用户可参考roe (https://github.com/COMBINE-lab/roe),python用户可参考 pyroe (https://github.com/COMBINE-lab/pyroe)。对于导入量化结果,该团队也对 R用户 (https://mikelove.github.io/fishpond/reference/loadFry.html)和 python用户 (https://github.com/COMBINE-lab/usefulaf/blob/main/python/load_fry.py)分别提供了支持。除此之外,usefulaf (https://hub.docker.com/r/combinelab/usefulaf)还提供了bash scripts和Docker与singularity镜像,让用户可以通过简单的几行命令预处理任何单细胞数据集。该团队还为用户提供了一种模拟CellRanger筛选细胞的方法,并被纳入DropletUtils (https://github.com/MarioniLab/DropletUtils/blob/master/R/emptyDropsCellRanger.R) R包中。并且,该团队还会不定期更新各种alevin-fry相关教程 (https://combine-lab.github.io/alevin-fry-tutorials/),截至发稿时,已有关于利用alevin-fry完成空间转录组分析,CITE-seq,sci-RNA-seq3及split-seq数据分析和RNA速率分析的相关教程。
Twitter 原文:https://twitter.com/nomad421/status/1503391195514482688
论文链接:https://www.nature.com/articles/s41592-022-01408-3
马里兰大学Rob Patro教授为本文通讯作者,博士生和东泽为本文第一作者。Rob 实验室还开发有Salmon, Sailfish, Mantis, Pufferfish 等工具。
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集