南大提出VM-UNet v2 | 在VM-UNet基础上细节&上下文信息都不放过,更小更快性能更强
点击下方卡片,关注「AI视界引擎」公众号

在医学图像分割领域,基于CNN和Transformer的模型已经得到了深入研究。然而,CNN在建模长距离依赖方面存在局限性,这使得充分利用图像内的语义信息变得具有挑战性。另一方面,二次计算复杂度为Transformers提出了挑战。近来,状态空间模型(SSMs),如Mamba,被认为是一种有前景的方法。它们不仅在建模长距离交互方面表现出卓越的性能,同时保持了线性的计算复杂度。
受到Mamba架构的启发,作者提出了Vison Mamba-UNetV2,引入了视觉状态空间(VSS)块来捕捉广泛的上下文信息,引入了语义与细节注入(SDI)来增强低层次和高层次特征的融合。
作者在ISIC17、ISIC18、CVC-300、CVC-ClinicDB、Kvasir、CVC-ColonDB和ETIS-LaribPolyDB等公共数据集上进行了全面实验。结果表明,VM-UNetV2在医学图像分割任务中表现出竞争力。
代码:https://github.com/nobodyplayer1/VM-UNetV2
1 Introduction
随着医学成像技术的不断发展,医学影像已成为诊断疾病和规划治疗的关键工具。在医学图像分析的基本和关键技术中,医学图像分割占有重要位置。这个过程涉及到在医学图像中区分器官或病变的像素,例如CT扫描和内窥镜视频。医学图像分割是医学图像分析中最困难的任务之一,其目标是为这些器官或组织的形状和体积提供并提取关键信息。近年来,深度学习技术已被用于提高医学图像分割的性能。这些模型从图像中提取有用信息,提高准确性,并适应不同的数据集和任务。
在语义图像分割中,常用的方法是采用带有跳跃连接的编码器-解码器网络。在这个框架中,编码器从输入图像中捕获分层和抽象的特征。另一方面,解码器使用编码器生成的特征图来构建像素级的分割 Mask 或地图,为输入图像中的每个像素赋予一个类别标签。已经进行了许多研究,以将全局信息整合到特征图中并增强多尺度特征,这导致了分割性能的显著提升。
U-Net 是一种关键架构,因其平衡的编码器-解码器设计和跳层连接的融合而备受赞誉。这种结构通过其各种编码器和解码器允许在多个层级上提取特征信息。此外,跳层连接有效地促进了这一特征信息的转换。对U-Net的众多研究主要关注以下方面:编码器部分 - 用不同的 Backbone 网来获得不同层级的特征图;跳层连接 - 采用各种通道注意力机制,并交换不同的连接部分;解码器部分 - 使用不同的采样和特征融合方案。
基于卷积神经网络(CNN)的模型由于其局部感受野,难以捕捉长距离信息,这可能导致特征提取不充分和分割结果不理想。基于Transformer的模型在全球建模方面表现出色,但它们自注意力机制的双曲复杂性造成了高昂的计算成本,特别是在像医学图像分割这类需要密集预测的任务中。这些局限性使得作者需要一种新的架构来进行医学图像分割,这种架构能够高效捕捉长距离信息同时保持线性计算复杂性。
近期在状态空间模型(SSMs)方面的进展,尤其是结构化SSMs(S4),由于它们在处理长序列方面的专长,提供了一个有效的解决方案。例如,Mamba。Mamba模型通过增加选择机制和硬件优化来增强S4,在密集数据领域展示了卓越的性能。在视觉状态空间模型(VMDaba)中融入交叉扫描模块(CSM)进一步提升了Mamba适用于计算机视觉任务的能力。它通过促进空间域的遍历,将非因果视觉图像转换为有序的斑块序列来实现这一点。
受到VMDaba 在图像分类任务和VM-Unet在医学图像分割中成功的启发。遵循UNetV2的框架,本文提出了视觉Mamba-UNetV2(VM-UNetV2),作者重新整合了低 Level 和高 Level 的特征,将语义信息融入到低 Level 特征中,同时使用更详细的信息来细化高级特征。
作者在与胃肠病学语义分割任务以及皮肤病变分割相关的任务上进行了详尽的实验,以展示纯基于SSM的模型在医学图像分割领域的性能。特别是对ISIC17、ISIC18、CVC-300、CVC-ClinicDB、Kvasir、CVC-ColonDB和ETIS-LaribPolyDB等公开数据集以及作者的私有数据集ZD-LCI-EGGIM进行了广泛的测试。结果表明,VM-UNetV2能够提供具有竞争力的分割结果。
本研究的主要贡献可以概括为以下几点:
-
作者提出了VM-UnetV2,在基于SSM的医疗图像分割算法探索方面开创了先河。 -
在七个数据集上进行了详尽的实验,结果证明了VM-UNetV2具有显著竞争力。 -
作者正在开创将基于SSM的方法与Unet变体结合的探索,推动更高效、更有效的基于SSM的分割算法的发展。
2 Methods
Preliminaries
在当前的基于SSM的模型中,即结构化状态空间序列模型(S4)和Mamba,都依赖于一个传统的连续系统,这个系统将一维输入函数或序列,表示为 ,通过中间隐含状态 映射到一个输出 。这个过程可以描述为一个线性常微分方程(ODE):
在这里, 代表状态矩阵,而 和 分别表示投影参数。
S4和Mamba通过离散化这个连续系统,使其更适合深度学习环境。具体来说,它们引入了一个时间尺度参数 ,并使用一致的离散化规则将 和 转换为离散参数 和 。零阶保持(ZOH)通常被用作离散化规则,可以概述如下:
在离散化之后,基于SSM的模型可以通过两种不同的方法进行计算:线性递推或全局卷积,分别表示为方程(3)和方程(4)。
其中 表示一个结构化的卷积核,而 指的是输入序列 的长度。
VM-UNetV2 Architecture
视觉Mamba UNetV2的全面结构如图1所示。它由三个主要模块组成:编码器、SDI(语义和细节注入)模块和解码器。给定一个输入图像I,其中 ,编码器在M个层次上生成特征。作者将第 级的特征表示为 ,其中 。这些累积的特征, 随后被转发到SDI模块进行进一步增强。

如图所示,编码器输出通道 的大小为 , 共同输入到SDI模块进行特征融合,并且 对应于 作为第 阶段的输出。 的特征大小为 。在VM-UNetV2中,作者使用深度监督来计算 和 特征的损失。
在本文中,作者在编码器的四个阶段使用了 VSS块,每个阶段的通道数分别为 。根据作者在VMamba [13]中的观察, 和 的不同值是区分Tiny、Small和Base框架规格的重要因素。遵循VMamba的规格,作者让C取值为96, 和 各自取值为2, 取值从集合中选取。这表示作者有意使用VMamba的Tiny和Small模型作为作者消融实验的主干。
VSS And SDI Block
VSS块源自V Mamba,作为VMTUeV2编码器的基础部分,VSS块的结构如图2 所示。输入首先通过一个初始的线性嵌入层进行处理,之后分为两个独立的信息流。一个信息流通过一个 深度卷积[11]层,然后经过一个Silu激活[19]函数,再进入主要的2D-Selective-Scan(SS2D)模块。SS2D的输出接着通过一个层归一化层,并与另一个信息流的输出合并,该信息流也经过Silu激活处理。这个合并后的输出构成了VSS块的最终结果。
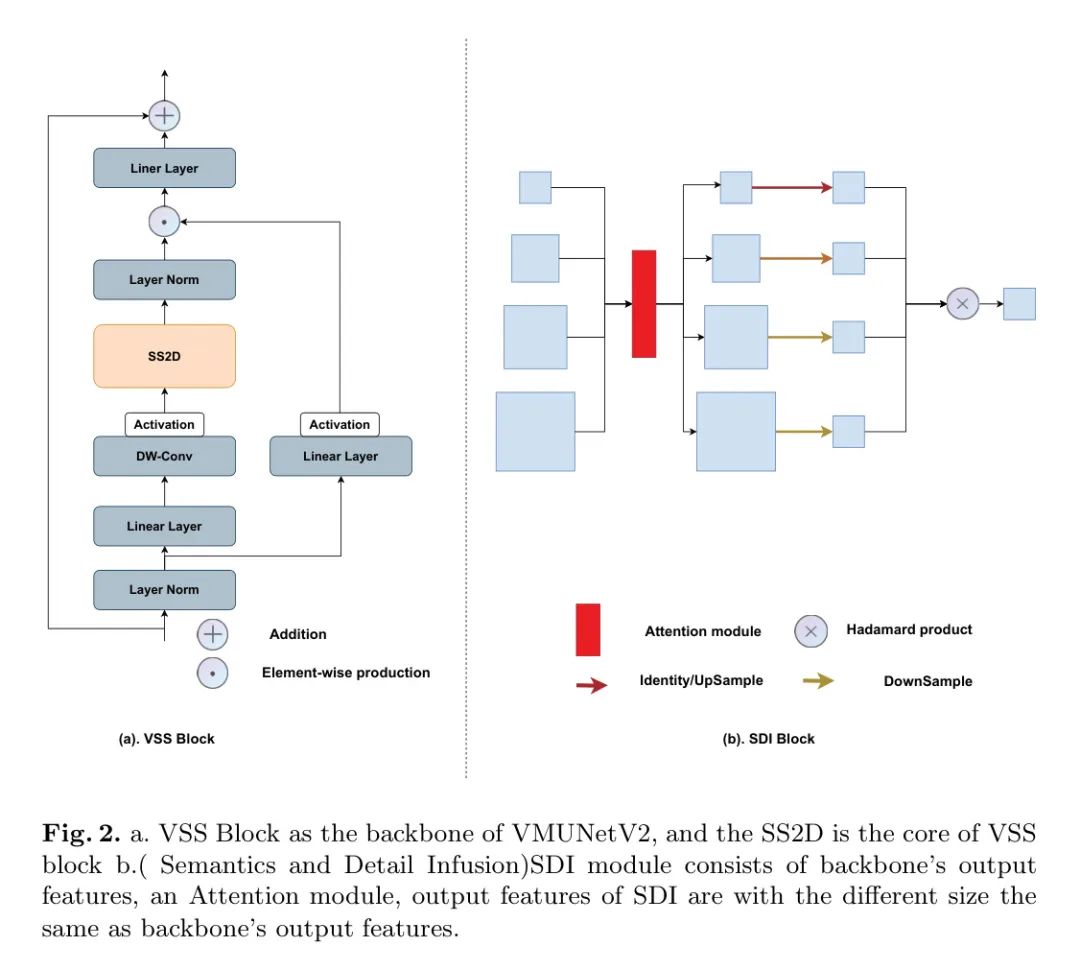
SDI模块,如图2(b)所示。利用编码器生成的分层特征图 ,其中 表示第 Level 。
不同的注意力机制可以用于SDI模块中,以计算空间和通道的注意力得分。遵循UNetV2 [15]中提到的方法,作者使用CBAM来实现空间和时间的注意力。计算公式如下,其中 表示第 注意力计算:
然后作者使用 卷积将 的通道对齐到 ,得到的结果特征图表示为 。
在SDI解码器的第 阶段, 表示目标参考。然后作者调整每个 级特征图的大小,以匹配 的大小,如下公式所示:
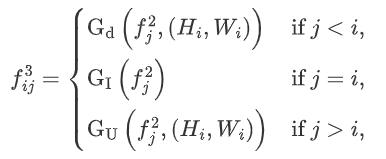
在公式6中, 、 和 分别代表自适应平均池化、身份映射和双线性插值。在公式7中, 表示平滑卷积的参数, 是第 个层次上的第 个平滑特征图。这里, 表示哈达玛积(Hadamard product)。随后, 会被送入第 个层次的解码器,进行进一步的分辨率重建和分割。
Loss function
对于作者的医学图像分割任务,作者主要采用基本的交叉熵和Dice损失作为损失函数,因为作者的所有数据集 Mask 都包含两个类别:单一目标和背景。
( )是常数,通常选择(1,1)作为默认参数。
3 Experiments and results
Datasets
作者使用了三种类型的数据集来验证作者框架的有效性。第一种类型是开源的皮肤病数据集,包括ISIC 2017和ISIC 2018,作者将皮肤数据集以7:3的比例划分为训练集和测试集。第二种是开源的胃肠道息肉数据集,其中包括Kvasir-SEG、ClinicDB、ColonDB、Endoscene和ETIS,在这一类数据集中,作者遵循PraNet中的实验设置。对于这些数据集,作者提供了在几个指标上的详细评估,包括平均交并比(mIoU)、Dice相似系数(DSC)、准确率(Acc)、敏感性(Sen)和特异性(Spe)。
Experimental setup
在V Mamba工作的基础上,作者将所有数据集中的图像尺寸调整为256×256像素。为了抑制过拟合,作者还引入了数据增强方法,比如随机翻转和随机旋转。在操作参数方面,作者将批处理大小设置为80,采用AdamW优化器,初始学习率为1e-3。作者使用CosineAnnealingLR作为调度器,其操作最多持续50个迭代,学习率最低降至1e-5。作者的训练历时300个周期。
对于VM-UNetV2,编码器单元的权重最初设置为与V Mamba-S相匹配。实施工作在一个Ubuntu 20.04系统上进行,使用Python3.9.12、PyTorch2.0.1和CUDA11.7,所有实验都在单个NVIDIA RTX V100 GPU上完成。
Results
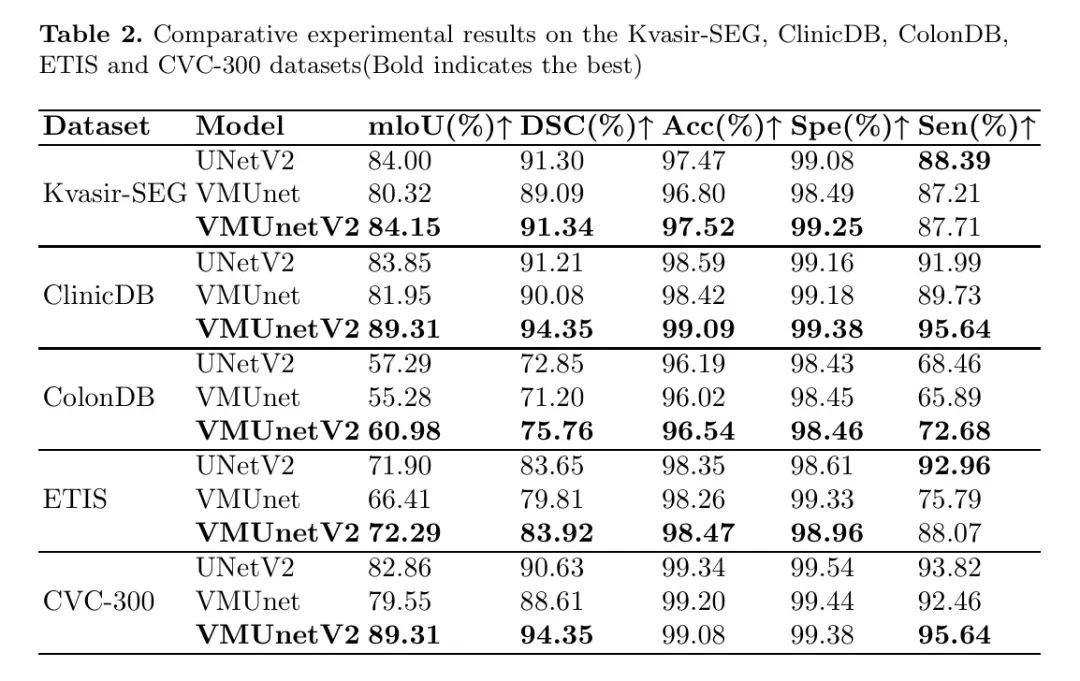
作者对比了VM-UNetV2与一些最先进模型的性能,并在表1和表2中展示了实验结果。
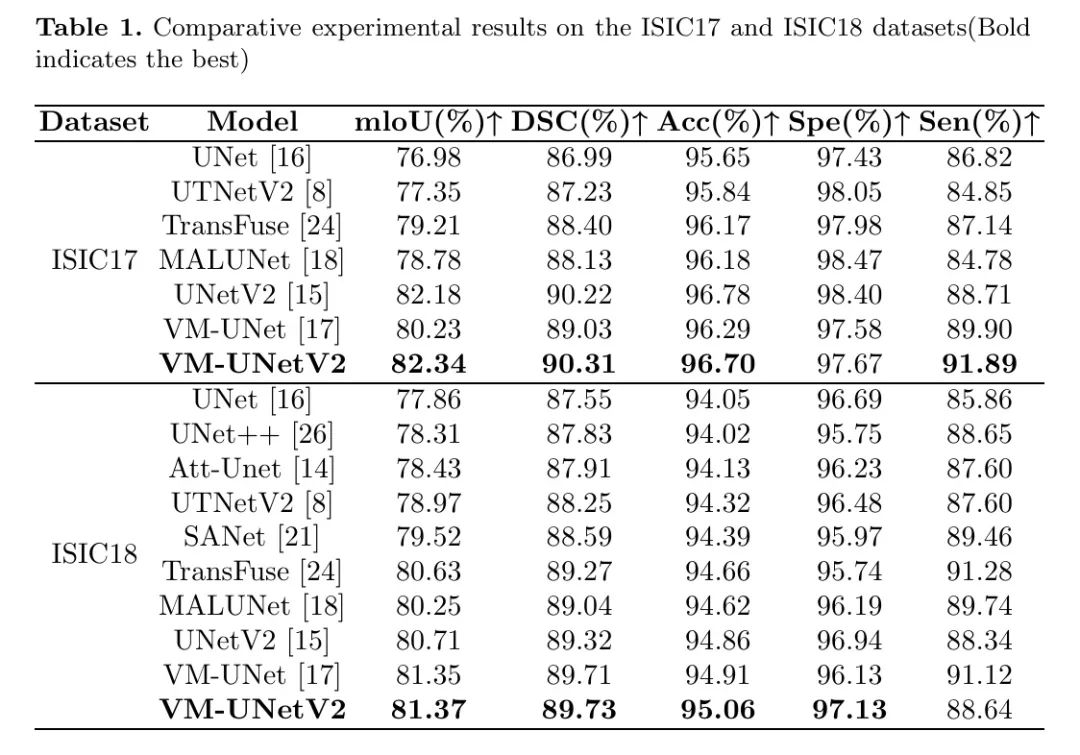
对于ISIC数据集,VM-UNetV2在mIoU、DSC和Acc指标上均优于其他模型。在息肉相关数据集中,VM-UNetV2在所有指标上也超过了最先进的UNetV2模型,在mIoU参数上提高了高达7%。
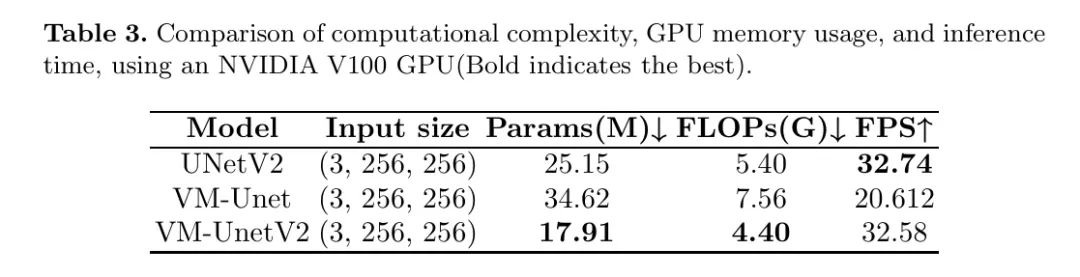
Ablation studies
在本节中,作者针对VM-UNetV2编码器的初始化和解码器中的深度监督操作,使用息肉数据集进行消融实验。正如VMamba论文中所指出的,编码器的深度和特征图中的通道数决定了VMamba的规模。在本文中,所提出的VM-UNetV2仅在编码器部分使用在ImageNet-1k上预训练的VMamba权重。
因此,在进行本研究中的模型规模消融实验时,作者仅改变编码器的深度,如表4所示。对于输出特征,作者采用深度监督机制,融合了两层输出特征,然后与真实标签进行比较以计算损失。
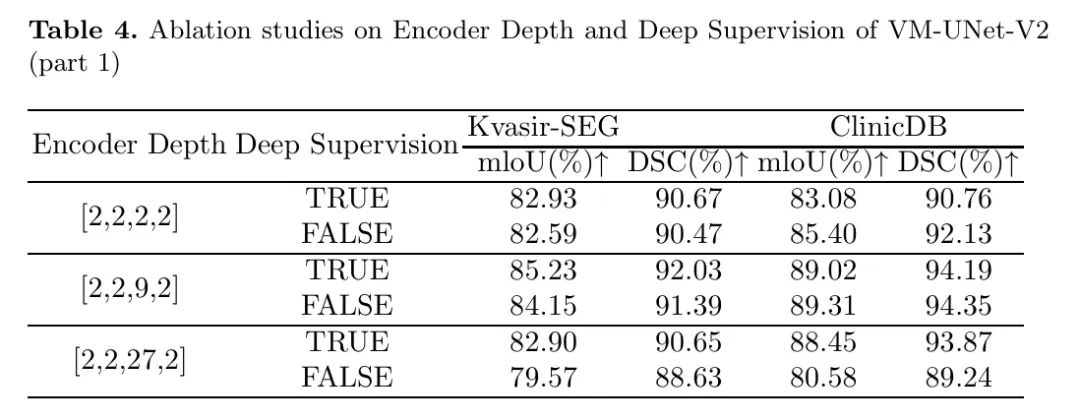
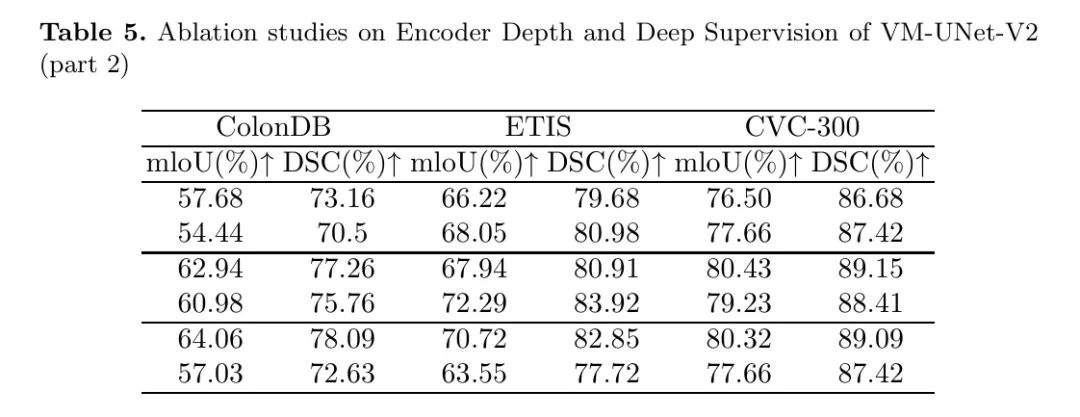
如表4和表5所示,当编码器的深度设置为 时,分割评估指标相对较好。因此,在使用VM-UNetV2时,无需选择特别大的深度。在大多数使用深度监督机制的情况下,分割评估指标相对较好,但这不是一个决定性因素。对于不同的数据集,需要分别进行消融实验以确定是否采用深度监督机制。
4 Conclusions
在本文中,作者提出了一种基于SSM的UNet类型医疗图像分割模型VM-UNetV2,充分利用了基于SSM模型的性能。作者分别使用VSS块和SDI来处理编码器和解码器连接部分。使用VMamba的预训练权重来初始化VM-UNetV2的编码器部分,并采用深度监督机制来监督多个输出特征。
VM-UNetV2已经在皮肤病和息肉数据集上进行了广泛测试。结果显示,在分割任务中,VM-UNetV2具有很高的竞争力。复杂性分析表明,VM-UNetV2在FLOPs、Params和FPS方面也是高效的。
参考
[1].VM-UNet-V2: Rethinking Vision Mamba UNet for Medical Image Segmentation.
点击上方卡片,关注「AI视界引擎」公众号
